+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22386 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of chicken CLC-7 | |||||||||
 マップデータ マップデータ | Density map of chicken CLC-7 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Lysosomal / chloride-proton antiporter / chloride transport / ion transport / proton transport / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Stimuli-sensing channels / chloride:proton antiporter activity / voltage-gated chloride channel activity / chloride transmembrane transporter activity / transmembrane transporter activity / membrane => GO:0016020 / intracellular membrane-bounded organelle / nucleoplasm / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.92 Å | |||||||||
 データ登録者 データ登録者 | Schrecker M / Hite R | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Cryo-EM structure of the lysosomal chloride-proton exchanger CLC-7 in complex with OSTM1. 著者: Marina Schrecker / Julia Korobenko / Richard K Hite /  要旨: The chloride-proton exchanger CLC-7 plays critical roles in lysosomal homeostasis and bone regeneration and its mutation can lead to osteopetrosis, lysosomal storage disease and neurological ...The chloride-proton exchanger CLC-7 plays critical roles in lysosomal homeostasis and bone regeneration and its mutation can lead to osteopetrosis, lysosomal storage disease and neurological disorders. In lysosomes and the ruffled border of osteoclasts, CLC-7 requires a β-subunit, OSTM1, for stability and activity. Here, we present electron cryomicroscopy structures of CLC-7 in occluded states by itself and in complex with OSTM1, determined at resolutions up to 2.8 Å. In the complex, the luminal surface of CLC-7 is entirely covered by a dimer of the heavily glycosylated and disulfide-bonded OSTM1, which serves to protect CLC-7 from the degradative environment of the lysosomal lumen. OSTM1 binding does not induce large-scale rearrangements of CLC-7, but does have minor effects on the conformation of the ion-conduction pathway, potentially contributing to its regulatory role. These studies provide insights into the role of OSTM1 and serve as a foundation for understanding the mechanisms of CLC-7 regulation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22386.map.gz emd_22386.map.gz | 200.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22386-v30.xml emd-22386-v30.xml emd-22386.xml emd-22386.xml | 14.7 KB 14.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_22386.png emd_22386.png | 182.6 KB | ||
| Filedesc metadata |  emd-22386.cif.gz emd-22386.cif.gz | 6.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22386 http://ftp.pdbj.org/pub/emdb/structures/EMD-22386 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22386 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22386 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22386_validation.pdf.gz emd_22386_validation.pdf.gz | 389.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22386_full_validation.pdf.gz emd_22386_full_validation.pdf.gz | 389.1 KB | 表示 | |
| XML形式データ |  emd_22386_validation.xml.gz emd_22386_validation.xml.gz | 7.2 KB | 表示 | |
| CIF形式データ |  emd_22386_validation.cif.gz emd_22386_validation.cif.gz | 8.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22386 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22386 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22386 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22386 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22386.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22386.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Density map of chicken CLC-7 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.71487 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Chicken CLC-7
| 全体 | 名称: Chicken CLC-7 |
|---|---|
| 要素 |
|
-超分子 #1: Chicken CLC-7
| 超分子 | 名称: Chicken CLC-7 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Chloride channel protein
| 分子 | 名称: Chloride channel protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 88.684609 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MANVAKKVSW SGRDPRDDED ERAGETTPLL NGTGPGSAGG ARQFTPSSFL RPGQLSNVDL NEDIRELETE LPRPYPNEIP HNEKLLSLK YESLDYDNSE NQLFLEEERR INHAAFRTVE IKRWVICAMI GILTGLVACF IDIVVENLAG LKYRVVKDNI D KFTEKGGL ...文字列: MANVAKKVSW SGRDPRDDED ERAGETTPLL NGTGPGSAGG ARQFTPSSFL RPGQLSNVDL NEDIRELETE LPRPYPNEIP HNEKLLSLK YESLDYDNSE NQLFLEEERR INHAAFRTVE IKRWVICAMI GILTGLVACF IDIVVENLAG LKYRVVKDNI D KFTEKGGL SFSLLLWATL NASVVMVGSV IVAFIEPVAA GSGIPQIKCY LNGVKIPHVV RLKTLVIKVC GVILSVVGGL AV GKEGPMI HSGAVIAAGI SQGRSTSLKR DFKIFEYFRR DTEKRDFVSA GAAAGVSAAF GAPVGGVLFS LEEGASFWNQ FLT WRIFFA SMISTFTLNS VLSVYHGNAW DLSSPGLINF GRFDSEKMGY TIQEIPIFIF MGVVGGILGA LFNALNYWLT MFRI RYIHR PCLQVIEAML VAAVTAAVGF VMIYCSRDCQ PIQGSSVAYP LQLFCADGEY NSMATAFFNT PEKSVVNLFH DPPGS YNPM TLGMFTLMYF FLACWTYGLT VSAGVFIPSL LIGAAWGRLF GISLSYLSKG SIWADPGKYA LMGAAAQLGG IVRMTL SLT VIMMEATGNV TYGFPIMLVL MTAKIVGDYF VEGLYDMHIQ LQSVPFLHWE APVTSHSLTA REVMSTPVTC LRRIERV GT VVDILSDTSS NHNGFPVVES NPNTTQVAGL RGLILRSQLI VLLKHKVFVE RANLNLVQRR LKLKDFRDAY PRFPPIQS I HVSQDERECM IDLSEFMNPS PYTVPREASL PRVFKLFRAL GLRHLVVVNN HNEVVGMVTR KDLARYRLGK EGLEELSLA QT UniProtKB: Chloride channel protein |
-分子 #2: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: CHLORIDE ION
| 分子 | 名称: CHLORIDE ION / タイプ: ligand / ID: 4 / コピー数: 6 / 式: CL |
|---|---|
| 分子量 | 理論値: 35.453 Da |
-分子 #5: (2R)-3-{[(R)-hydroxy{[(1S,2R,3S,4S,5R,6R)-2,3,4,6-tetrahydroxy-5-...
| 分子 | 名称: (2R)-3-{[(R)-hydroxy{[(1S,2R,3S,4S,5R,6R)-2,3,4,6-tetrahydroxy-5-(phosphonooxy)cyclohexyl]oxy}phosphoryl]oxy}propane-1,2-diyl dinonanoate タイプ: ligand / ID: 5 / コピー数: 2 / 式: 0J1 |
|---|---|
| 分子量 | 理論値: 694.64 Da |
| Chemical component information | 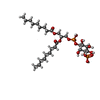 ChemComp-0J1: |
-分子 #6: water
| 分子 | 名称: water / タイプ: ligand / ID: 6 / コピー数: 72 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. | ||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均露光時間: 8.0 sec. / 平均電子線量: 44.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: OTHER / Cs: 2.7 mm / 最大 デフォーカス(公称値): -2.5 µm / 最小 デフォーカス(公称値): -1.2 µm / 倍率(公称値): 22500 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-7jm6: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)





















