+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21509 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human mAbs broadly protect against infection of arthritiogenic alphaviruses by recognizing conserved elements of the MXR8 receptor binding domain | |||||||||
 マップデータ マップデータ | unsharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | virus / monoclonal antibody / complex / VIRUS-IMMUNE SYSTEM complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報togavirin / T=4 icosahedral viral capsid / symbiont-mediated suppression of host toll-like receptor signaling pathway / host cell cytoplasm / serine-type endopeptidase activity / fusion of virus membrane with host endosome membrane / symbiont entry into host cell / virion attachment to host cell / host cell nucleus / host cell plasma membrane ...togavirin / T=4 icosahedral viral capsid / symbiont-mediated suppression of host toll-like receptor signaling pathway / host cell cytoplasm / serine-type endopeptidase activity / fusion of virus membrane with host endosome membrane / symbiont entry into host cell / virion attachment to host cell / host cell nucleus / host cell plasma membrane / virion membrane / structural molecule activity / proteolysis / RNA binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Mayaro virus (strain Brazil) (ウイルス) / Mayaro virus (strain Brazil) (ウイルス) /  Mayaro virus (マヤロウイルス) Mayaro virus (マヤロウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.3 Å | |||||||||
 データ登録者 データ登録者 | Miller AS / Kuhn RJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Host Microbe / 年: 2020 ジャーナル: Cell Host Microbe / 年: 2020タイトル: Human mAbs Broadly Protect against Arthritogenic Alphaviruses by Recognizing Conserved Elements of the Mxra8 Receptor-Binding Site. 著者: Laura A Powell / Andrew Miller / Julie M Fox / Nurgun Kose / Thomas Klose / Arthur S Kim / Robin Bombardi / Rashika N Tennekoon / A Dharshan de Silva / Robert H Carnahan / Michael S Diamond / ...著者: Laura A Powell / Andrew Miller / Julie M Fox / Nurgun Kose / Thomas Klose / Arthur S Kim / Robin Bombardi / Rashika N Tennekoon / A Dharshan de Silva / Robert H Carnahan / Michael S Diamond / Michael G Rossmann / Richard J Kuhn / James E Crowe /  要旨: Mosquito inoculation of humans with arthritogenic alphaviruses results in a febrile syndrome characterized by debilitating musculoskeletal pain and arthritis. Despite an expanding global disease ...Mosquito inoculation of humans with arthritogenic alphaviruses results in a febrile syndrome characterized by debilitating musculoskeletal pain and arthritis. Despite an expanding global disease burden, no approved therapies or licensed vaccines exist. Here, we describe human monoclonal antibodies (mAbs) that bind to and neutralize multiple distantly related alphaviruses. These mAbs compete for an antigenic site and prevent attachment to the recently discovered Mxra8 alphavirus receptor. Three cryoelectron microscopy structures of Fab in complex with Ross River (RRV), Mayaro, or chikungunya viruses reveal a conserved footprint of the broadly neutralizing mAb RRV-12 in a region of the E2 glycoprotein B domain. This mAb neutralizes virus in vitro by preventing virus entry and spread and is protective in vivo in mouse models. Thus, the RRV-12 mAb and its defined epitope have potential as a therapeutic agent or target of vaccine design against multiple emerging arthritogenic alphavirus infections. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21509.map.gz emd_21509.map.gz | 279.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21509-v30.xml emd-21509-v30.xml emd-21509.xml emd-21509.xml | 20.2 KB 20.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21509.png emd_21509.png | 199.6 KB | ||
| Filedesc metadata |  emd-21509.cif.gz emd-21509.cif.gz | 6.3 KB | ||
| その他 |  emd_21509_half_map_1.map.gz emd_21509_half_map_1.map.gz emd_21509_half_map_2.map.gz emd_21509_half_map_2.map.gz | 279.7 MB 279.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21509 http://ftp.pdbj.org/pub/emdb/structures/EMD-21509 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21509 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21509 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21509_validation.pdf.gz emd_21509_validation.pdf.gz | 928.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21509_full_validation.pdf.gz emd_21509_full_validation.pdf.gz | 928.2 KB | 表示 | |
| XML形式データ |  emd_21509_validation.xml.gz emd_21509_validation.xml.gz | 22.2 KB | 表示 | |
| CIF形式データ |  emd_21509_validation.cif.gz emd_21509_validation.cif.gz | 27.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21509 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21509 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21509 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21509 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21509.map.gz / 形式: CCP4 / 大きさ: 1.1 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21509.map.gz / 形式: CCP4 / 大きさ: 1.1 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.62 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
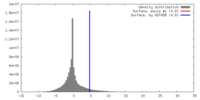
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: half map 1, unsharpened
| ファイル | emd_21509_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1, unsharpened | ||||||||||||
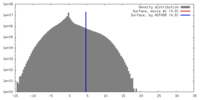
| 投影像・断面図 |
| ||||||||||||
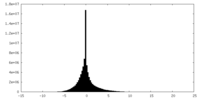
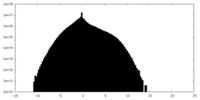
| 密度ヒストグラム |
-ハーフマップ: half map 3, unsharpened
| ファイル | emd_21509_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 3, unsharpened | ||||||||||||
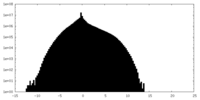
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mayaro virus-Fab CHK-265 complex
| 全体 | 名称: Mayaro virus-Fab CHK-265 complex |
|---|---|
| 要素 |
|
-超分子 #1: Mayaro virus-Fab CHK-265 complex
| 超分子 | 名称: Mayaro virus-Fab CHK-265 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #3: Fab CHK-265
| 超分子 | 名称: Fab CHK-265 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3-#4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #2: Mayaro virus
| 超分子 | 名称: Mayaro virus / タイプ: virus / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 / NCBI-ID: 59301 / 生物種: Mayaro virus / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|
-分子 #1: E1 glycoprotein
| 分子 | 名称: E1 glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mayaro virus (strain Brazil) (ウイルス) / 株: Brazil Mayaro virus (strain Brazil) (ウイルス) / 株: Brazil |
| 分子量 | 理論値: 41.612867 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: YEHTAIIPNQ VGFPYKAHVA REGYSPLTLQ MQVIETSLEP TLNLEYITCD YKTKVPSPYV KCCGTAECRT QDKPEYKCAV FTGVYPFMW GGAYCFCDSE NTQMSEAYVE RADVCKHDHA AAYRAHTASL RAKIKVTYGT VNQTVEAYVN GDHAVTIAGT K FIFGPVST ...文字列: YEHTAIIPNQ VGFPYKAHVA REGYSPLTLQ MQVIETSLEP TLNLEYITCD YKTKVPSPYV KCCGTAECRT QDKPEYKCAV FTGVYPFMW GGAYCFCDSE NTQMSEAYVE RADVCKHDHA AAYRAHTASL RAKIKVTYGT VNQTVEAYVN GDHAVTIAGT K FIFGPVST PWTPFDTKIL VYKGELYNQD FPRYGAGQPG RFGDIQSRTL DSRDLYANTG LKLARPAAGN IHVPYTQTPS GF KTWQKDR DSPLNAKAPF GCIIQTNPVR AMNCAVGNIP VSMDIADSAF TRLTDAPVIS ELTCTVSTCT HSSDFGGIAV LSY KVEKSG RCDIHSHSNV AVLQEVSIET EGRSVIHFST ASASPSFVVS VCSSRATCTA KCEP UniProtKB: Structural polyprotein |
-分子 #2: E2 glycoprotein
| 分子 | 名称: E2 glycoprotein / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mayaro virus (strain Brazil) (ウイルス) / 株: Brazil Mayaro virus (strain Brazil) (ウイルス) / 株: Brazil |
| 分子量 | 理論値: 38.041957 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: STANHFNAYK LTRPYVAYCA DCGMGHSCHS PAMIENIQAD ATDGTLKIQF ASQIGLTKTD THDHTKIRYA EGHDIAEAAR STLKVHSSS ECTVTGTMGH FILAKCPPGE RISVSFVDSK NEHRTCRIAY HHEQRLIGRE RFTVRPHHGI ELPCTTYQLT T AETSEEID ...文字列: STANHFNAYK LTRPYVAYCA DCGMGHSCHS PAMIENIQAD ATDGTLKIQF ASQIGLTKTD THDHTKIRYA EGHDIAEAAR STLKVHSSS ECTVTGTMGH FILAKCPPGE RISVSFVDSK NEHRTCRIAY HHEQRLIGRE RFTVRPHHGI ELPCTTYQLT T AETSEEID MHMPPDIPDR TILSQQSGNV KITVNGRTVR YSSSCGSQAV GTTTTDKTIN SCTVDKCQAY VTSHTKWQFN SP FVPRRMQ AERKGKVHIP FPLINTTCRV PLAPEALVRS GKREATLSLH PIHPTLLSYR TFGAERVFDE QWITAQTEVT IPV PVEGVE YQWGNHKPQR FVVA UniProtKB: Structural polyprotein |
-分子 #3: Fab CHK-265 heavy chain
| 分子 | 名称: Fab CHK-265 heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.743719 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QIQLVQSGRE VKNPGETVKI SCKASGYTFT EYPMLWVKQA PGKGFRWMGL IYTNTGEPTY AEEFKGRFVF SLEISASTAY LQINNLTNE DTATYFCVRD YFISLDYWGQ GTTLTVSSAK TTAPSVYPLA PVCGGTTGSS VTLGCLVKGY FPEPVTLTWN S GSLSSGVH ...文字列: QIQLVQSGRE VKNPGETVKI SCKASGYTFT EYPMLWVKQA PGKGFRWMGL IYTNTGEPTY AEEFKGRFVF SLEISASTAY LQINNLTNE DTATYFCVRD YFISLDYWGQ GTTLTVSSAK TTAPSVYPLA PVCGGTTGSS VTLGCLVKGY FPEPVTLTWN S GSLSSGVH TFPALLQSGL YTLSSSVTVT SNTWPSQTIT CNVAHPASST KVDKKIESRR |
-分子 #4: Fab CHK-265 light chain
| 分子 | 名称: Fab CHK-265 light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.888404 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QAVVTQESAL TTSPGETVTL TCRSNIGAVT SSNCANWVQE KPDHFFTGLI GDTNNRRSGV PARFSGSLIG DKAALTITGA QTEDEAIYF CALWYNNLWV FGGGTKLTVL GQPKSSPSVT LFPPSSEELE TNKATLVCTI TDFYPGVVTV DWKVDGTPVT Q GMETTQPS ...文字列: QAVVTQESAL TTSPGETVTL TCRSNIGAVT SSNCANWVQE KPDHFFTGLI GDTNNRRSGV PARFSGSLIG DKAALTITGA QTEDEAIYF CALWYNNLWV FGGGTKLTVL GQPKSSPSVT LFPPSSEELE TNKATLVCTI TDFYPGVVTV DWKVDGTPVT Q GMETTQPS KQSNNKYMAS SYLTLTARAW ERHSSYSCQV THEGHTVEKS LSR |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: C-flat / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 85 % / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 BASE (4k x 4k) 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 18000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / 解像度のタイプ: BY AUTHOR / 解像度: 5.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: jspr / 使用した粒子像数: 18410 |
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: jspr |
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-6w1c: |
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)