+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21383 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of CAP256-VRC26.25 Fab bound to HIV-1 Env trimer CAP256.wk34.c80 SOSIP.RnS2 | |||||||||
 マップデータ マップデータ | Sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | V1V2 / VRC26 / CAP256 / HIV-1 / SOSIP / Vaccine / Superinfecting / VIRAL PROTEIN-IMMUNE SYSTEM complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / extracellular region / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Gorman J / Kwong PD | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2020 ジャーナル: Cell Rep / 年: 2020タイトル: Structure of Super-Potent Antibody CAP256-VRC26.25 in Complex with HIV-1 Envelope Reveals a Combined Mode of Trimer-Apex Recognition. 著者: Jason Gorman / Gwo-Yu Chuang / Yen-Ting Lai / Chen-Hsiang Shen / Jeffrey C Boyington / Aliaksandr Druz / Hui Geng / Mark K Louder / Krisha McKee / Reda Rawi / Raffaello Verardi / Yongping ...著者: Jason Gorman / Gwo-Yu Chuang / Yen-Ting Lai / Chen-Hsiang Shen / Jeffrey C Boyington / Aliaksandr Druz / Hui Geng / Mark K Louder / Krisha McKee / Reda Rawi / Raffaello Verardi / Yongping Yang / Baoshan Zhang / Nicole A Doria-Rose / Bob Lin / Penny L Moore / Lynn Morris / Lawrence Shapiro / John R Mascola / Peter D Kwong /   要旨: Antibodies targeting the V1V2 apex of the HIV-1 envelope (Env) trimer comprise one of the most commonly elicited categories of broadly neutralizing antibodies. Structures of these antibodies indicate ...Antibodies targeting the V1V2 apex of the HIV-1 envelope (Env) trimer comprise one of the most commonly elicited categories of broadly neutralizing antibodies. Structures of these antibodies indicate diverse modes of Env recognition typified by antibodies of the PG9 class and the PGT145 class. The mode of recognition, however, has been unclear for the most potent of the V1V2 apex-targeting antibodies, CAP256-VRC26.25 (named for donor-lineage.clone and referred to hereafter as VRC26.25). Here, we determine the cryoelectron microscopy structure at 3.7 Å resolution of the antigen-binding fragment of VRC26.25 in complex with the Env trimer thought to have initiated the lineage. The 36-residue protruding loop of VRC26.25 displays recognition incorporating both strand-C interactions similar to the PG9 class and V1V2 apex insertion similar to the PGT145 class. Structural elements of separate antibody classes can thus intermingle to form a "combined" class, which in this case yields an antibody of extraordinary potency. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21383.map.gz emd_21383.map.gz | 108.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21383-v30.xml emd-21383-v30.xml emd-21383.xml emd-21383.xml | 24.8 KB 24.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21383.png emd_21383.png | 79 KB | ||
| マスクデータ |  emd_21383_msk_1.map emd_21383_msk_1.map | 115.9 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-21383.cif.gz emd-21383.cif.gz | 7.3 KB | ||
| その他 |  emd_21383_additional.map.gz emd_21383_additional.map.gz emd_21383_half_map_1.map.gz emd_21383_half_map_1.map.gz emd_21383_half_map_2.map.gz emd_21383_half_map_2.map.gz | 23.4 MB 107.6 MB 107.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21383 http://ftp.pdbj.org/pub/emdb/structures/EMD-21383 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21383 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21383 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21383.map.gz / 形式: CCP4 / 大きさ: 115.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21383.map.gz / 形式: CCP4 / 大きさ: 115.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0961 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
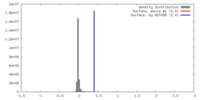
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_21383_msk_1.map emd_21383_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
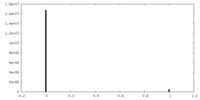
| 密度ヒストグラム |
-追加マップ: Unsharpened map
| ファイル | emd_21383_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
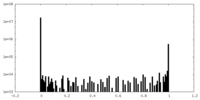
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_21383_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
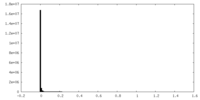
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_21383_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM Structure of CAP256-VRC26.25 Fab bound to HIV-1 Env trime...
| 全体 | 名称: Cryo-EM Structure of CAP256-VRC26.25 Fab bound to HIV-1 Env trimer CAP256.wk34.c80 SOSIP.RnS2 |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM Structure of CAP256-VRC26.25 Fab bound to HIV-1 Env trime...
| 超分子 | 名称: Cryo-EM Structure of CAP256-VRC26.25 Fab bound to HIV-1 Env trimer CAP256.wk34.c80 SOSIP.RnS2 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|
-超分子 #2: HIV-1 Env trimer CAP256.wk34.c80 SOSIP.RnS2
| 超分子 | 名称: HIV-1 Env trimer CAP256.wk34.c80 SOSIP.RnS2 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1, #4 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-超分子 #3: CAP256-VRC26.25 Fab
| 超分子 | 名称: CAP256-VRC26.25 Fab / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Envelope glycoprotein gp120
| 分子 | 名称: Envelope glycoprotein gp120 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 53.322434 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GLWVTVYYGV PVWREAKTTL FCASDAKSYE KEVHNVWATH ACVPTDPNPQ ELVLENVTEN FNMWKNDMVD QMHEDIISLW DQSLKPCVK LTPLCVTLNC SDAKVNATYK GTREEIKNCS FNATTELRDK KRREYALFYR LDIVPLSGEG NNNSEYRLIN C NTSVITQI ...文字列: GLWVTVYYGV PVWREAKTTL FCASDAKSYE KEVHNVWATH ACVPTDPNPQ ELVLENVTEN FNMWKNDMVD QMHEDIISLW DQSLKPCVK LTPLCVTLNC SDAKVNATYK GTREEIKNCS FNATTELRDK KRREYALFYR LDIVPLSGEG NNNSEYRLIN C NTSVITQI CPKVTFDPIP IHYCAPAGYA ILKCNNKTFN GTGPCNNVST VQCTHGIKPV VSTQLLLNGS LAEEEIIIRS EN LTDNVKT IIVHLNESVE ITCTRPNNMT RKSVRIGPGQ TFYALGDIIG DIRQPHCNIS EIKWEKTLQR VSEKLREHFN KTI IFNQSS GGDLEITTHS FNCGGEFFYC NTSDLFFNKT FNETYSTGSN STNSTITLPC RIKQIINMWQ EVGRAMYAPP IAGN ITCKS NITGLLLTRD GGGNNSTKET FRPGGGNMRD NWRSELYKYK VVEVKPLGIA PTECNRTVVQ RRRRRR |
-分子 #2: VRC26.25 Heavy Chain
| 分子 | 名称: VRC26.25 Heavy Chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 28.083482 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLVESGGG VVQPGTSLRL SCAASQFRFD GYGMHWVRQA PGKGLEWVAS ISHDGIKKYH AEKVWGRFTI SRDNSKNTLY LQMNSLRPE DTALYYCAKD LREDECEEWW SD(TYS)(TYS)DFGKQL PCAKSRGGLV GIADNWGQGT MVTVSSASTK GPS VFPLAP ...文字列: QVQLVESGGG VVQPGTSLRL SCAASQFRFD GYGMHWVRQA PGKGLEWVAS ISHDGIKKYH AEKVWGRFTI SRDNSKNTLY LQMNSLRPE DTALYYCAKD LREDECEEWW SD(TYS)(TYS)DFGKQL PCAKSRGGLV GIADNWGQGT MVTVSSASTK GPS VFPLAP SSKSTSGGTA ALGCLVKDYF PEPVTVSWNS GALTSGVHTF PAVLQSSGLY SLSSVVTVPS SSLGTQTYIC NVNH KPSNT KVDKRVEPKS CDKGLEVLFQ |
-分子 #3: VRC26.25 Light Chain
| 分子 | 名称: VRC26.25 Light Chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.052611 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QSVLTQPPSV SAAPGQKVTI SCSGNTSNIG NNFVSWYQQR PGRAPQLLIY ETDKRPSGIP DRFSASKSGT SGTLAITGLQ TGDEADYYC ATWAASLSSA RVFGTGTKVI VLVQPKANPT VTLFPPSSEE LQANKATLVC LISDFYPGAV TVAWKADSSP V KAGVETTT ...文字列: QSVLTQPPSV SAAPGQKVTI SCSGNTSNIG NNFVSWYQQR PGRAPQLLIY ETDKRPSGIP DRFSASKSGT SGTLAITGLQ TGDEADYYC ATWAASLSSA RVFGTGTKVI VLVQPKANPT VTLFPPSSEE LQANKATLVC LISDFYPGAV TVAWKADSSP V KAGVETTT PSKQSNNKYA ASSYLSLTPE QWKSHRSYSC QVTHEGSTVE KTVAPTECS UniProtKB: Lambda-chain (AA -20 to 215) |
-分子 #4: Envelope glycoprotein gp41
| 分子 | 名称: Envelope glycoprotein gp41 / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 17.355703 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AVVGLGAVFL GFLGAAGSTM GAASNTLTVQ ARQLLSGIVQ QQSNLLRAPE AQQHMLQLGV WGFKQLQARV LAIERYLEVQ QLLGMWGCS GKLICCTNVP WNSSWSNKTY NEIWDNMTWM QWDREIGNYT DTIYKLLEVS QFQQEINEKD NLTLD |
-分子 #11: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 11 / コピー数: 41 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 式: PBS |
| グリッド | モデル: C-flat-1.2/1.3 4C / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | Cryo-EM Structure of Fab CAP256-VRC26.25 bound to HIV-1 Env trimer CAP256 SU.wk34.80 SOSIP.RnS2 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 1777 / 平均露光時間: 10.0 sec. / 平均電子線量: 64.48 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)