+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21113 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo EM Structure of VPS13 protein, 1-1390 from C. thermophilum | |||||||||
 マップデータ マップデータ | VPS13 protein | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) | |||||||||
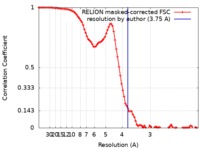
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.75 Å | |||||||||
 データ登録者 データ登録者 | Li P / Lees JA / Reinisch KM | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Cell Biol / 年: 2020 ジャーナル: J Cell Biol / 年: 2020タイトル: Cryo-EM reconstruction of a VPS13 fragment reveals a long groove to channel lipids between membranes. 著者: PeiQi Li / Joshua Aaron Lees / C Patrick Lusk / Karin M Reinisch /  要旨: A single particle cryo-EM reconstruction of an ∼160-kD N-terminal fragment of the lipid transport protein VPS13 reveals an ∼160-Å long channel lined with hydrophobic residues suitable for ...A single particle cryo-EM reconstruction of an ∼160-kD N-terminal fragment of the lipid transport protein VPS13 reveals an ∼160-Å long channel lined with hydrophobic residues suitable for solubilizing multiple lipid fatty acid moieties. The structure suggests that VPS13 and related proteins, like the autophagy protein ATG2, can act as bridges between organelle membranes to allow bulk lipid flow between organelles. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21113.map.gz emd_21113.map.gz | 55.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21113-v30.xml emd-21113-v30.xml emd-21113.xml emd-21113.xml | 9.9 KB 9.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21113_fsc.xml emd_21113_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21113.png emd_21113.png | 94.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21113 http://ftp.pdbj.org/pub/emdb/structures/EMD-21113 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21113 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21113 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21113_validation.pdf.gz emd_21113_validation.pdf.gz | 78 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21113_full_validation.pdf.gz emd_21113_full_validation.pdf.gz | 77.1 KB | 表示 | |
| XML形式データ |  emd_21113_validation.xml.gz emd_21113_validation.xml.gz | 492 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21113 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21113 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21113 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21113 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21113.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21113.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | VPS13 protein | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Chaetomium thermophilum VPS13 1-1390
| 全体 | 名称: Chaetomium thermophilum VPS13 1-1390 |
|---|---|
| 要素 |
|
-超分子 #1: Chaetomium thermophilum VPS13 1-1390
| 超分子 | 名称: Chaetomium thermophilum VPS13 1-1390 / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) |
| 分子量 | 理論値: 161.84 kDa/nm |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: Expi293F Homo sapiens (ヒト) / 組換細胞: Expi293F |
-分子 #1: Vacuolar protein sorting-associate protein 13
| 分子 | 名称: Vacuolar protein sorting-associate protein 13 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDYKDHDGDY KDHDIDYKDD DDKLAAA LE GLVAGLLNRF LGMYVKNFDP KQLKWEVWNG KVRLDNLELQ REALDQLKLP INVIKGHLGH LVLHIPW KT LASEQVKINI EDVFLLASPK EEAEYDEDEE ARRRHRLKME KLDSAELLKE RSQEGLSEEE QKRTQTFA Q ...文字列: MDYKDHDGDY KDHDIDYKDD DDKLAAA LE GLVAGLLNRF LGMYVKNFDP KQLKWEVWNG KVRLDNLELQ REALDQLKLP INVIKGHLGH LVLHIPW KT LASEQVKINI EDVFLLASPK EEAEYDEDEE ARRRHRLKME KLDSAELLKE RSQEGLSEEE QKRTQTFA Q ALVTKIVDNL QITIRNIHIR YEDAISAPGH PFALGITLEE FSAVSTDSDW TPAFITSIQS AHKLATLES LAIYWDTDAK LIGPGREPHE HSDQIPHDEM LKFFREMIAK GEADLSSEHQ FILKPVSGQA KIEIDKTGSH TVPRYKANL LFDEIGVVLD DQQYRDALMM VDLFHYFIRH QEYKKFQPKG VTPKEDPRAW FRFAGNAVLS K IHERNRRW SWDYFRERRD DRRRYIELFK KTKQNIQLTP EEREDLDKLE WKLSYEDLRF WRSLARNQLK KE NAEALKN KPPPQPQQQQ GWLSWVWGSK PVQPQQEEQQ GDENTRITEA QRKELYEVIQ WDEKAALAAE IDV PRDSVR LLIETSLSTG SFTLRQNPHG DARDLISLHF DLFRAKGLTR PDSFLIDISL GGFRVNDNTT PDSL YKEIV RVKDAPNTEG QKRYSIADLE LTVDEEAFFE LQVEQHPLDG QGDVAVTMKL KPLEIIWNPN VVVGI ADFF RPPERHMESI NALLETANAT VEGLRAQTRA GLQFALEEHK TVNAKLDLQA PLIILPESIT TPNSTC LIV DAGHISVNSE LVDKETMKQV QSTQDRPCTE EDLRRLEELM YDRFLVKLTS TQVLIGPSVE ITKQQLV QR DEKRQLHIVD QINLDFVVAM SILPKAPNLT KLKISGHLPV LQVNASDSKY KHLMRIIEVA IPKLYDVE P VLAPSGSHPT IRPRLASDAS TRSRRASFRK ASTQFLQFVS QQQEIVLDES DSDDDSEKFE DAKDTSVDE QLRIQQRIFD FKFTVDQLRG SLYRSDPEGK HPDQLLVELV AENFGVEYHL RPYDMSAMVS LGSVTMDDFV ENPPAEFKS IVSSGDIEDR KQARDLVRVK FVRVKKESPE FMSVYDGIET NVDVAISTIN LVVTRKTLLT L LDFILVTF SNPQPAAPVA TRMAVTDQES ETNIIVQPPP IESGPIRVKV DLKSIRMILN NDGIRLATLS FN HADVGVY ILGRYMRVSA KLGDLSLVDD VNLGVSEDSS LRQLVTIQGN ELADFRYEYF DPDKPEKNPG YDS SIYLRA GSVKVNFIEE PFRKIVDFLV KFGKMQAIYN AARMAAANQA QQLQQSQSRI KFDIVVKTPI VVFP RVVMS PKPKRDVITA YLGEIYAQNA FVPLDDSEKA DMAMKLTTGI RNIRLTSHFH YSEGRDEVLE LIDHV DLGF TIIYAEHKEG IKRPDLEIEG SMSDFNLRIT PYQLSALLAI SQSVPTVFAA DVEQHT |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)