+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20484 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of HIV cleaved synaptic complex (CSC) intasome bound with magnesium and INSTI XZ426 (compound 4d) | ||||||||||||
 マップデータ マップデータ | Structure of HIV cleaved synaptic complex intasome bound to 4d; sharpened | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | integrase / intasome / transposition / VIRAL PROTEIN / VIRAL PROTEIN-DNA complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA endonuclease activity / DNA integration / viral genome integration into host DNA / establishment of integrated proviral latency / RNA stem-loop binding / RNA-directed DNA polymerase activity / endonuclease activity / DNA recombination / symbiont entry into host cell / DNA binding ...RNA endonuclease activity / DNA integration / viral genome integration into host DNA / establishment of integrated proviral latency / RNA stem-loop binding / RNA-directed DNA polymerase activity / endonuclease activity / DNA recombination / symbiont entry into host cell / DNA binding / zinc ion binding / cytoplasm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | ||||||||||||
 データ登録者 データ登録者 | Lyumkis D / Passos D | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2020 ジャーナル: Science / 年: 2020タイトル: Structural basis for strand-transfer inhibitor binding to HIV intasomes. 著者: Dario Oliveira Passos / Min Li / Ilona K Jóźwik / Xue Zhi Zhao / Diogo Santos-Martins / Renbin Yang / Steven J Smith / Youngmin Jeon / Stefano Forli / Stephen H Hughes / Terrence R Burke / ...著者: Dario Oliveira Passos / Min Li / Ilona K Jóźwik / Xue Zhi Zhao / Diogo Santos-Martins / Renbin Yang / Steven J Smith / Youngmin Jeon / Stefano Forli / Stephen H Hughes / Terrence R Burke / Robert Craigie / Dmitry Lyumkis /  要旨: The HIV intasome is a large nucleoprotein assembly that mediates the integration of a DNA copy of the viral genome into host chromatin. Intasomes are targeted by the latest generation of ...The HIV intasome is a large nucleoprotein assembly that mediates the integration of a DNA copy of the viral genome into host chromatin. Intasomes are targeted by the latest generation of antiretroviral drugs, integrase strand-transfer inhibitors (INSTIs). Challenges associated with lentiviral intasome biochemistry have hindered high-resolution structural studies of how INSTIs bind to their native drug target. Here, we present high-resolution cryo-electron microscopy structures of HIV intasomes bound to the latest generation of INSTIs. These structures highlight how small changes in the integrase active site can have notable implications for drug binding and design and provide mechanistic insights into why a leading INSTI retains efficacy against a broad spectrum of drug-resistant variants. The data have implications for expanding effective treatments available for HIV-infected individuals. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20484.map.gz emd_20484.map.gz | 200.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20484-v30.xml emd-20484-v30.xml emd-20484.xml emd-20484.xml | 27.6 KB 27.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20484.png emd_20484.png | 88.1 KB | ||
| Filedesc metadata |  emd-20484.cif.gz emd-20484.cif.gz | 7.1 KB | ||
| その他 |  emd_20484_additional_1.map.gz emd_20484_additional_1.map.gz emd_20484_additional_2.map.gz emd_20484_additional_2.map.gz emd_20484_half_map_1.map.gz emd_20484_half_map_1.map.gz emd_20484_half_map_2.map.gz emd_20484_half_map_2.map.gz | 196.6 MB 11.8 MB 26.4 MB 26.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20484 http://ftp.pdbj.org/pub/emdb/structures/EMD-20484 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20484 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20484 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20484_validation.pdf.gz emd_20484_validation.pdf.gz | 710.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20484_full_validation.pdf.gz emd_20484_full_validation.pdf.gz | 709.9 KB | 表示 | |
| XML形式データ |  emd_20484_validation.xml.gz emd_20484_validation.xml.gz | 15.9 KB | 表示 | |
| CIF形式データ |  emd_20484_validation.cif.gz emd_20484_validation.cif.gz | 18.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20484 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20484 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20484 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20484 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6puyMC  6putC  6puwC  6puzC  6v3kC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10387 (タイトル: Structural basis for strand-transfer inhibitor binding to HIV intasomes EMPIAR-10387 (タイトル: Structural basis for strand-transfer inhibitor binding to HIV intasomesData size: 4.4 TB Data #1: Aligned, summed and exposure weighted micrographs of HIV-1 intasome bound to compound 4d. [micrographs - single frame] Data #2: Unaligned raw movies of HIV-1 intasome bound to compound 4d [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20484.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20484.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of HIV cleaved synaptic complex intasome bound to 4d; sharpened | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.79 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
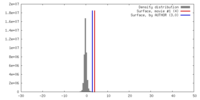
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Structure of HIV cleaved synaptic complex intasome bound...
| ファイル | emd_20484_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
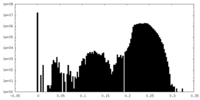
| 注釈 | Structure of HIV cleaved synaptic complex intasome bound to 4d. Full map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
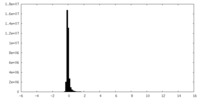
| 密度ヒストグラム |
-追加マップ: local resolution
| ファイル | emd_20484_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
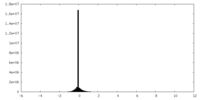
| 注釈 | local resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
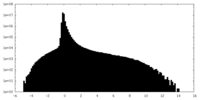
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_20484_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
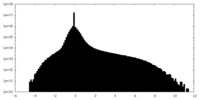
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
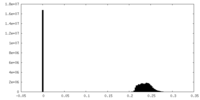
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_20484_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
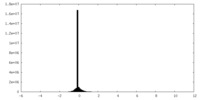
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Assembly of HIV integrase and viral DNA
| 全体 | 名称: Assembly of HIV integrase and viral DNA |
|---|---|
| 要素 |
|
-超分子 #1: Assembly of HIV integrase and viral DNA
| 超分子 | 名称: Assembly of HIV integrase and viral DNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 400 KDa |
-分子 #1: Chimeric Sso7d and HIV-1 integrase
| 分子 | 名称: Chimeric Sso7d and HIV-1 integrase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 42.321258 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MATVKFKYKG EEKEVDISKI KKVWRVGKMI SFTYDEGGGK TGRGAVSEKD APKELLQMLE KQKKGGGGG GGGGGGFLDG IDKAQEEHEK YHSNWRAMAS DFNLPPVVAK EIVASCDKCQ LKGEAMHGQV DCSPGIWQLD C THLEGKVI ...文字列: MGSSHHHHHH SSGLVPRGSH MATVKFKYKG EEKEVDISKI KKVWRVGKMI SFTYDEGGGK TGRGAVSEKD APKELLQMLE KQKKGGGGG GGGGGGFLDG IDKAQEEHEK YHSNWRAMAS DFNLPPVVAK EIVASCDKCQ LKGEAMHGQV DCSPGIWQLD C THLEGKVI LVAVHVASGY IEAEVIPAET GQETAYFLLK LAGRWPVKTV HTDNGSNFTS TTVKAACWWA GIKQEFGIPY NP QSQGVIE SMNKELKKII GQVRDQAEHL KTAVQMAVFI HNFKRKGGIG GYSAGERIVD IIATDIQTKE LQKQITKIQN FRV YYRDSR DPVWKGPAKL LWKGEGAVVI QDNSDIKVVP RRKAKIIRDY GKQMAGDDCV ASRQDED UniProtKB: DNA-binding protein 7d, Gag-Pol polyprotein |
-分子 #2: viral DNA non-transferred strand
| 分子 | 名称: viral DNA non-transferred strand / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 8.188271 KDa |
| 配列 | 文字列: (DA)(DC)(DT)(DG)(DC)(DT)(DA)(DG)(DA)(DG) (DA)(DT)(DT)(DT)(DT)(DC)(DC)(DC)(DG)(DC) (DC)(DC)(DA)(DC)(DG)(DC)(DT) |
-分子 #3: viral DNA transferred strand
| 分子 | 名称: viral DNA transferred strand / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 7.773023 KDa |
| 配列 | 文字列: (DA)(DG)(DC)(DG)(DT)(DG)(DG)(DG)(DC)(DG) (DG)(DG)(DA)(DA)(DA)(DA)(DT)(DC)(DT)(DC) (DT)(DA)(DG)(DC)(DA) |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 5 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #6: 4-amino-N-[(2,4-difluorophenyl)methyl]-1-hydroxy-6-(6-hydroxyhexy...
| 分子 | 名称: 4-amino-N-[(2,4-difluorophenyl)methyl]-1-hydroxy-6-(6-hydroxyhexyl)-2-oxo-1,2-dihydro-1,8-naphthyridine-3-carboxamide タイプ: ligand / ID: 6 / コピー数: 1 / 式: OZ1 |
|---|---|
| 分子量 | 理論値: 446.447 Da |
| Chemical component information | 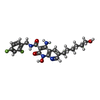 ChemComp-OZ1: |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 202 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6.2 構成要素:
| ||||||||||||||||||
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 7 sec. | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 4-80 / 実像数: 2024 / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 63291 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 37000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: Correlation coefficient | ||||||||
| 得られたモデル |  PDB-6puy: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)