+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human Tip60 complex locally refined on RUVBL1/2 | |||||||||
 マップデータ マップデータ | Human Tip60 complex - RUVBL local refined map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Eukaryotic transcription / Histone acetyltransferase / chromatin remodeling / Complex / TRANSCRIPTION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報piccolo histone acetyltransferase complex / promoter-enhancer loop anchoring activity / positive regulation of norepinephrine uptake / telomerase RNA localization to Cajal body / regulation of DNA strand elongation / positive regulation of telomere maintenance in response to DNA damage / sperm DNA condensation / histone chaperone activity / establishment of protein localization to chromatin / cellular response to cytochalasin B ...piccolo histone acetyltransferase complex / promoter-enhancer loop anchoring activity / positive regulation of norepinephrine uptake / telomerase RNA localization to Cajal body / regulation of DNA strand elongation / positive regulation of telomere maintenance in response to DNA damage / sperm DNA condensation / histone chaperone activity / establishment of protein localization to chromatin / cellular response to cytochalasin B / bBAF complex / npBAF complex / R2TP complex / nBAF complex / brahma complex / regulation of transepithelial transport / dynein axonemal particle / neural retina development / morphogenesis of a polarized epithelium / structural constituent of postsynaptic actin cytoskeleton / GBAF complex / Formation of annular gap junctions / Formation of the dystrophin-glycoprotein complex (DGC) / protein localization to adherens junction / Gap junction degradation / regulation of G0 to G1 transition / Folding of actin by CCT/TriC / Swr1 complex / dense body / Cell-extracellular matrix interactions / postsynaptic actin cytoskeleton / Tat protein binding / RPAP3/R2TP/prefoldin-like complex / protein antigen binding / chromatin-protein adaptor activity / Prefoldin mediated transfer of substrate to CCT/TriC / RSC-type complex / Ino80 complex / blastocyst formation / regulation of nucleotide-excision repair / regulation of double-strand break repair / adherens junction assembly / RHOF GTPase cycle / Adherens junctions interactions / apical protein localization / Sensory processing of sound by outer hair cells of the cochlea / tight junction / box C/D snoRNP assembly / Interaction between L1 and Ankyrins / SWI/SNF complex / regulation of mitotic metaphase/anaphase transition / protein folding chaperone complex / Sensory processing of sound by inner hair cells of the cochlea / positive regulation of T cell differentiation / regulation of norepinephrine uptake / apical junction complex / transporter regulator activity / Formation of Senescence-Associated Heterochromatin Foci (SAHF) / positive regulation of double-strand break repair / spinal cord development / maintenance of blood-brain barrier / nitric-oxide synthase binding / negative regulation of gene expression, epigenetic / establishment or maintenance of cell polarity / cortical cytoskeleton / NuA4 histone acetyltransferase complex / regulation of chromosome organization / positive regulation of stem cell population maintenance / RUNX1 interacts with co-factors whose precise effect on RUNX1 targets is not known / Transcriptional Regulation by E2F6 / Regulation of MITF-M-dependent genes involved in pigmentation / Recycling pathway of L1 / brush border / regulation of G1/S transition of mitotic cell cycle / TFIID-class transcription factor complex binding / regulation of DNA replication / MLL1 complex / kinesin binding / regulation of embryonic development / Telomere Extension By Telomerase / EPH-ephrin mediated repulsion of cells / negative regulation of cell differentiation / somatic stem cell population maintenance / spermatid development / regulation of synaptic vesicle endocytosis / RHO GTPases Activate WASPs and WAVEs / enzyme-substrate adaptor activity / positive regulation of myoblast differentiation / RHO GTPases activate IQGAPs / regulation of protein localization to plasma membrane / positive regulation of double-strand break repair via homologous recombination / RNA polymerase II core promoter sequence-specific DNA binding / regulation of DNA repair / cytoskeleton organization / EPHB-mediated forward signaling / Deposition of new CENPA-containing nucleosomes at the centromere / substantia nigra development / telomere maintenance / axonogenesis / DNA helicase activity 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
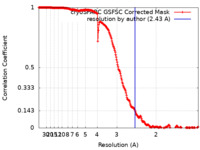
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.43 Å | |||||||||
 データ登録者 データ登録者 | Li C / Smirnova E / Schnitzler C / Crucifix C / Concordet JP / Brion A / Poterszman A / Schultz P / Papai G / Ben-Shem A | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Structure of the human TIP60-C histone exchange and acetyltransferase complex. 著者: Changqing Li / Ekaterina Smirnova / Charlotte Schnitzler / Corinne Crucifix / Jean Paul Concordet / Alice Brion / Arnaud Poterszman / Patrick Schultz / Gabor Papai / Adam Ben-Shem /  要旨: Chromatin structure is a key regulator of DNA transcription, replication and repair. In humans, the TIP60-EP400 complex (TIP60-C) is a 20-subunit assembly that affects chromatin structure through two ...Chromatin structure is a key regulator of DNA transcription, replication and repair. In humans, the TIP60-EP400 complex (TIP60-C) is a 20-subunit assembly that affects chromatin structure through two enzymatic activities: ATP-dependent exchange of histone H2A-H2B for H2A.Z-H2B, and histone acetylation. In yeast, however, these activities are performed by two independent complexes-SWR1 and NuA4, respectively. How the activities of the two complexes are merged into one supercomplex in humans, and what this association entails for the structure and mechanism of the proteins and their recruitment to chromatin, are unknown. Here we describe the structure of the endogenous human TIP60-C. We find a three-lobed architecture composed of SWR1-like (SWR1L) and NuA4-like (NuA4L) parts, which associate with a TRRAP activator-binding module. The huge EP400 subunit contains the ATPase motor, traverses the junction between SWR1L and NuA4L twice and constitutes the scaffold of the three-lobed architecture. NuA4L is completely rearranged compared with its yeast counterpart. TRRAP is flexibly tethered to NuA4L-in stark contrast to its robust connection to the completely opposite side of NuA4 in yeast. A modelled nucleosome bound to SWR1L, supported by tests of TIP60-C activity, suggests that some aspects of the histone exchange mechanism diverge from what is seen in yeast. Furthermore, a fixed actin module (as opposed to the mobile actin subcomplex in SWR1; ref. ), the flexibility of TRRAP and the weak effect of extranucleosomal DNA on exchange activity lead to a different, activator-based mode of enlisting TIP60-C to chromatin. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18591.map.gz emd_18591.map.gz | 616 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18591-v30.xml emd-18591-v30.xml emd-18591.xml emd-18591.xml | 26.8 KB 26.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18591_fsc.xml emd_18591_fsc.xml | 22.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18591.png emd_18591.png | 68.6 KB | ||
| マスクデータ |  emd_18591_msk_1.map emd_18591_msk_1.map | 1.2 GB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18591.cif.gz emd-18591.cif.gz | 8.8 KB | ||
| その他 |  emd_18591_half_map_1.map.gz emd_18591_half_map_1.map.gz emd_18591_half_map_2.map.gz emd_18591_half_map_2.map.gz | 1.1 GB 1.1 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18591 http://ftp.pdbj.org/pub/emdb/structures/EMD-18591 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18591 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18591 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18591_validation.pdf.gz emd_18591_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18591_full_validation.pdf.gz emd_18591_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_18591_validation.xml.gz emd_18591_validation.xml.gz | 30.4 KB | 表示 | |
| CIF形式データ |  emd_18591_validation.cif.gz emd_18591_validation.cif.gz | 39.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18591 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18591 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18591 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18591 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8qr1C  8qriC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18591.map.gz / 形式: CCP4 / 大きさ: 1.2 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18591.map.gz / 形式: CCP4 / 大きさ: 1.2 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human Tip60 complex - RUVBL local refined map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.73 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18591_msk_1.map emd_18591_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
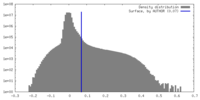
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_18591_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_18591_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tip60 complex
| 全体 | 名称: Tip60 complex |
|---|---|
| 要素 |
|
-超分子 #1: Tip60 complex
| 超分子 | 名称: Tip60 complex / タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 株: K562 Homo sapiens (ヒト) / 株: K562 |
-分子 #1: EP400
| 分子 | 名称: EP400 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MHHGTGPQNV QHQLQRSRAC PGSEGEEQPA HPNPPPSPAA PFAPSASPSA PQSPSYQIQQ LMNRSPATGQ NVNITLQSVG PVVGGNQQIT LAPLPLPSPT SPGFQFSAQP RRFEHGSPSY IQVTSPLSQQ VQTQSPTQPS PGPGQALQNV RAGAPGPGLG LCSSSPTGGF ...文字列: MHHGTGPQNV QHQLQRSRAC PGSEGEEQPA HPNPPPSPAA PFAPSASPSA PQSPSYQIQQ LMNRSPATGQ NVNITLQSVG PVVGGNQQIT LAPLPLPSPT SPGFQFSAQP RRFEHGSPSY IQVTSPLSQQ VQTQSPTQPS PGPGQALQNV RAGAPGPGLG LCSSSPTGGF VDASVLVRQI SLSPSSGGHF VFQDGSGLTQ IAQGAQVQLQ HPGTPITVRE RRPSQPHTQS GGTIHHLGPQ SPAAAGGAGL QPLASPSHIT TANLPPQISS IIQGQLVQQQ QVLQGPPLPR PLGFERTPGV LLPGAGGAAG FGMTSPPPPT SPSRTAVPPG LSSLPLTSVG NTGMKKVPKK LEEIPPASPE MAQMRKQCLD YHYQEMQALK EVFKEYLIEL FFLQHFQGNM MDFLAFKKKH YAPLQAYLRQ NDLDIEEEEE EEEEEEEKSE VINDEVKVVT GKDGQTGTPV AIATQLPPKV SAAFSSQQQP FQQALAGSLV AGAGSTVETD LFKRQQAMPS TGMAEQSKRP RLEVGHQGVV FQHPGADAGV PLQQLMPTAQ GGMPPTPQAA QLAGQRQSQQ QYDPSTGPPV QNAASLHTPL PQLPGRLPPA GVPTAALSSA LQFAQQPQVV EAQTQLQIPV KTQQPNVPIP APPSSQLPIP PSQPAQLALH VPTPGKVQVQ ASQLSSLPQM VASTRLPVDP APPCPRPLPT SSTSSLAPVS GSGPGPSPAR SSPVNRPSSA TNKALSPVTS RTPGVVASAP TKPQSPAQNA TSSQDSSQDT LTEQITLENQ VHQRIAELRK AGLWSQRRLP KLQEAPRPKS HWDYLLEEMQ WMATDFAQER RWKVAAAKKL VRTVVRHHEE KQLREERGKK EEQSRLRRIA ASTAREIECF WSNIEQVVEI KLRVELEEKR KKALNLQKVS RRGKELRPKG FDALQESSLD SGMSGRKRKA SISLTDDEVD DEEETIEEEE ANEGVVDHQT ELSNLAKEAE LPLLDLMKLY EGAFLPSSQW PRPKPDGEDT SGEEDADDCP GDRESRKDLV LIDSLFIMDQ FKAAERMNIG KPNAKDIADV TAVAEAILPK GSARVTTSVK FNAPSLLYGA LRDYQKIGLD WLAKLYRKNL NGILADEAGL GKTVQIIAFF AHLACNEGNW GPHLVVVRSC NILKWELELK RWCPGLKILS YIGSHRELKA KRQEWAEPNS FHVCITSYTQ FFRGLTAFTR VRWKCLVIDE MQRVKGMTER HWEAVFTLQS QQRLLLIDSP LHNTFLELWT MVHFLVPGIS RPYLSSPLRA PSEESQDYYH KVVIRLHRVT QPFILRRTKR DVEKQLTKKY EHVLKCRLSN RQKALYEDVI LQPGTQEALK SGHFVNVLSI LVRLQRICNH PGLVEPRHPG SSYVAGPLEY PSASLILKAL ERDFWKEADL SMFDLIGLEN KITRHEAELL SKKKIPRKLM EEISTSAAPA ARPAAAKLKA SRLFQPVQYG QKPEGRTVAF PSTHPPRTAA PTTASAAPQG PLRGRPPIAT FSANPEAKAA AAPFQTSQAS ASAPRHQPAS ASSTAASPAH PAKLRAQTTA QASTPGQPPP QPQAPSHAAG QSALPQRLVL PSQAQARLPS GEVVKIAQLA SITGPQSRVA QPETPVTLQF QGSKFTLSHS QLRQLTAGQP LQLQGSVLQI VSAPGQPYLR APGPVVMQTV SQAGAVHGAL GSKPPAGGPS PAPLTPQVGV PGRVAVNALA VGEPGTASKP ASPIGGPTQE EKTRLLKERL DQIYLVNERR CSQAPVYGRD LLRICALPSH GRVQWRGSLD GRRGKEAGPA HSYTSSSESP SELMLTLCRC GESLQDVIDR VAFVIPPVVA APPSLRVPRP PPLYSHRMRI LRQGLREHAA PYFQQLRQTT APRLLQFPEL RLVQFDSGKL EALAILLQKL KSEGRRVLIL SQMILMLDIL EMFLNFHYLT YVRIDENASS EQRQELMRSF NRDRRIFCAI LSTHSRTTGI NLVEADTVVF YDNDLNPVMD AKAQEWCDRI GRCKDIHIYR LVSGNSIEEK LLKNGTKDLI REVAAQGNDY SMAFLTQRTI QELFEVYSPM DDAGFPVKAE EFVVLSQEPS VTETIAPKIA RPFIEALKSI EYLEEDAQKS AQEGVLGPHT DALSSDSENM PCDEEPSQLE ELADFMEQLT PIEKYALNYL ELFHTSIEQE KERNSEDAVM TAVRAWEFWN LKTLQEREAR LRLEQEEAEL LTYTREDAYS MEYVYEDVDG QTEVMPLWTP PTPPQDDSDI YLDSVMCLMY EATPIPEAKL PPVYVRKERK RHKTDPSAAG RKKKQRHGEA VVPPRSLFDR ATPGLLKIRR EGKEQKKNIL LKQQVPFAKP LPTFAKPTAE PGQDNPEWLI SEDWALLQAV KQLLELPLNL TIVSPAHTPN WDLVSDVVNS CSRIYRSSKQ CRNRYENVII PREEGKSKNN RPLRTSQIYA QDENATHTQL YTSHFDLMKM TAGKRSPPIK PLLGMNPFQK NPKHASVLAE SGINYDKPLP PIQVASLRAE RIAKEKKALA DQQKAQQPAV AQPPPPQPQP PPPPQQPPPP LPQPQAAGSQ PPAGPPAVQP QPQPQPQTQP QPVQAPAKAQ PAITTGGSAA VLAGTIKTSV TGTSMPTGAV SGNVIVNTIA GVPAATFQSI NKRLASPVAP GALTTPGGSA PAQVVHTQPP PRAVGSPATA TPDLVSMATT QGVRAVTSVT ASAVVTTNLT PVQTPARSLV PQVSQATGVQ LPGKTITPAH FQLLRQQQQQ QQQQQQQQQQ QQQQQQQQQQ QQQQTTTTSQ VQVPQIQGQA QSPAQIKAVG KLTPEHLIKM QKQKLQMPPQ PPPPQAQSAP PQPTAQVQVQ TSQPPQQQSP QLTTVTAPRP GALLTGTTVA NLQVARLTRV PTSQLQAQGQ MQTQAPQPAQ VALAKPPVVS VPAAVVSSPG VTTLPMNVAG ISVAIGQPQK AAGQTVVAQP VHMQQLLKLK QQAVQQQKAI QPQAAQGPAA VQQKITAQQI TTPGAQQKVA YAAQPALKTQ FLTTPISQAQ KLAGAQQVQT QIQVAKLPQV VQQQTPVASI QQVASASQQA SPQTVALTQA TAAGQQVQMI PAVTATAQVV QQKLIQQQVV TTASAPLQTP GAPNPAQVPA SSDSPSQQPK LQMRVPAVRL KTPTKPPCQ UniProtKB: E1A-binding protein p400 |
-分子 #2: EPC1
| 分子 | 名称: EPC1 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSKLSFRARA LDASKPLPVF RCEDLPDLHE YASINRAVPQ MPTGMEKEEE SEHHLQRAIS AQQVYGEKRD NMVIPVPEAE SNIAYYESIY PGEFKMPKQL IHIQPFSLDA EQPDYDLDSE DEVFVNKLKK KMDICPLQFE EMIDRLEKGS GQQPVSLQEA KLLLKEDDEL ...文字列: MSKLSFRARA LDASKPLPVF RCEDLPDLHE YASINRAVPQ MPTGMEKEEE SEHHLQRAIS AQQVYGEKRD NMVIPVPEAE SNIAYYESIY PGEFKMPKQL IHIQPFSLDA EQPDYDLDSE DEVFVNKLKK KMDICPLQFE EMIDRLEKGS GQQPVSLQEA KLLLKEDDEL IREVYEYWIK KRKNCRGPSL IPSVKQEKRD GSSTNDPYVA FRRRTEKMQT RKNRKNDEAS YEKMLKLRRD LSRAVTILEM IKRREKSKRE LLHLTLEIME KRYNLGDYNG EIMSEVMAQR QPMKPTYAIP IIPITNSSQF KHQEAMDVKE FKVNKQDKAD LIRPKRKYEK KPKVLPSSAA ATPQQTSPAA LPVFNAKDLN QYDFPSSDEE PLSQVLSGSS EAEEDNDPDG PFAFRRKAGC QYYAPHLDQT GNWPWTSPKD GGLGDVRYRY CLTTLTVPQR CIGFARRRVG RGGRVLLDRA HSDYDSVFHH LDLEMLSSPQ HSPVNQFANT SETNTSDKSF SKDLSQILVN IKSCRWRHFR PRTPSLHDSD NDELSCRKLY RSINRTGTAQ PGTQTCSTST QSKSSSGSAH FAFTAEQYQQ HQQQLALMQK QQLAQIQQQQ ANSNSSTNTS QNLASNQQKS GFRLNIQGLE RTLQGFVSKT LDSASAQFAA SALVTSEQLM GFKMKDDVVL GIGVNGVLPA SGVYKGLHLS STTPTALVHT SPSTAGSALL QPSNITQTSS SHSALSHQVT AANSATTQVL IGNNIRLTVP SSVATVNSIA PINARHIPRT LSAVPSSALK LAAAANCQVS KVPSSSSVDS VPRENHESEK PALNNIADNT VAMEVT UniProtKB: Enhancer of polycomb homolog 1 |
-分子 #3: ACL6A
| 分子 | 名称: ACL6A / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSGGVYGGDE VGALVFDIGS YTVRAGYAGE DCPKVDFPTA IGMVVERDDG STLMEIDGDK GKQGGPTYYI DTNALRVPRE NMEAISPLKN GMVEDWDSFQ AILDHTYKMH VKSEASLHPV LMSEAPWNTR AKREKLTELM FEHYNIPAFF LCKTAVLTAF ANGRSTGLIL ...文字列: MSGGVYGGDE VGALVFDIGS YTVRAGYAGE DCPKVDFPTA IGMVVERDDG STLMEIDGDK GKQGGPTYYI DTNALRVPRE NMEAISPLKN GMVEDWDSFQ AILDHTYKMH VKSEASLHPV LMSEAPWNTR AKREKLTELM FEHYNIPAFF LCKTAVLTAF ANGRSTGLIL DSGATHTTAI PVHDGYVLQQ GIVKSPLAGD FITMQCRELF QEMNIELVPP YMIASKEAVR EGSPANWKRK EKLPQVTRSW HNYMCNCVIQ DFQASVLQVS DSTYDEQVAA QMPTVHYEFP NGYNCDFGAE RLKIPEGLFD PSNVKGLSGN TMLGVSHVVT TSVGMCDIDI RPGLYGSVIV AGGNTLIQSF TDRLNRELSQ KTPPSMRLKL IANNTTVERR FSSWIGGSIL ASLGTFQQMW ISKQEYEEGG KQCVERKCP UniProtKB: Actin-like protein 6A |
-分子 #4: VPS72
| 分子 | 名称: VPS72 / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSLAGGRAPR KTAGNRLSGL LEAEEEDEFY QTTYGGFTEE SGDDEYQGDQ SDTEDEVDSD FDIDEGDEPS SDGEAEEPRR KRRVVTKAYK EPLKSLRPRK VNTPAGSSQK AREEKALLPL ELQDDGSDSR KSMRQSTAEH TRQTFLRVQE RQGQSRRRKG PHCERPLTQE ...文字列: MSLAGGRAPR KTAGNRLSGL LEAEEEDEFY QTTYGGFTEE SGDDEYQGDQ SDTEDEVDSD FDIDEGDEPS SDGEAEEPRR KRRVVTKAYK EPLKSLRPRK VNTPAGSSQK AREEKALLPL ELQDDGSDSR KSMRQSTAEH TRQTFLRVQE RQGQSRRRKG PHCERPLTQE ELLREAKITE ELNLRSLETY ERLEADKKKQ VHKKRKCPGP IITYHSVTVP LVGEPGPKEE NVDIEGLDPA PSVSALTPHA GTGPVNPPAR CSRTFITFSD DATFEEWFPQ GRPPKVPVRE VCPVTHRPAL YRDPVTDIPY ATARAFKIIR EAYKKYITAH GLPPTASALG PGPPPPEPLP GSGPRALRQK IVIK UniProtKB: Vacuolar protein sorting-associated protein 72 homolog |
-分子 #5: DMAP1
| 分子 | 名称: DMAP1 / タイプ: protein_or_peptide / ID: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MATGADVRDI LELGGPEGDA ASGTISKKDI INPDKKKSKK SSETLTFKRP EGMHREVYAL LYSDKKDAPP LLPSDTGQGY RTVKAKLGSK KVRPWKWMPF TNPARKDGAM FFHWRRAAEE GKDYPFARFN KTVQVPVYSE QEYQLYLHDD AWTKAETDHL FDLSRRFDLR ...文字列: MATGADVRDI LELGGPEGDA ASGTISKKDI INPDKKKSKK SSETLTFKRP EGMHREVYAL LYSDKKDAPP LLPSDTGQGY RTVKAKLGSK KVRPWKWMPF TNPARKDGAM FFHWRRAAEE GKDYPFARFN KTVQVPVYSE QEYQLYLHDD AWTKAETDHL FDLSRRFDLR FVVIHDRYDH QQFKKRSVED LKERYYHICA KLANVRAVPG TDLKIPVFDA GHERRRKEQL ERLYNRTPEQ VAEEEYLLQE LRKIEARKKE REKRSQDLQK LITAADTTAE QRRTERKAPK KKLPQKKEAE KPAVPETAGI KFPDFKSAGV TLRSQRMKLP SSVGQKKIKA LEQMLLELGV ELSPTPTEEL VHMFNELRSD LVLLYELKQA CANCEYELQM LRHRHEALAR AGVLGGPATP ASGPGPASAE PAVTEPGLGP DPKDTIIDVV GAPLTPNSRK RRESASSSSS VKKAKKP UniProtKB: DNA methyltransferase 1-associated protein 1 |
-分子 #6: RUVBL1
| 分子 | 名称: RUVBL1 / タイプ: protein_or_peptide / ID: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKIEEVKSTT KTQRIASHSH VKGLGLDESG LAKQAASGLV GQENAREACG VIVELIKSKK MAGRAVLLAG PPGTGKTALA LAIAQELGSK VPFCPMVGSE VYSTEIKKTE VLMENFRRAI GLRIKETKEV YEGEVTELTP CETENPMGGY GKTISHVIIG LKTAKGTKQL ...文字列: MKIEEVKSTT KTQRIASHSH VKGLGLDESG LAKQAASGLV GQENAREACG VIVELIKSKK MAGRAVLLAG PPGTGKTALA LAIAQELGSK VPFCPMVGSE VYSTEIKKTE VLMENFRRAI GLRIKETKEV YEGEVTELTP CETENPMGGY GKTISHVIIG LKTAKGTKQL KLDPSIFESL QKERVEAGDV IYIEANSGAV KRQGRCDTYA TEFDLEAEEY VPLPKGDVHK KKEIIQDVTL HDLDVANARP QGGQDILSMM GQLMKPKKTE ITDKLRGEIN KVVNKYIDQG IAELVPGVLF VDEVHMLDIE CFTYLHRALE SSIAPIVIFA SNRGNCVIRG TEDITSPHGI PLDLLDRVMI IRTMLYTPQE MKQIIKIRAQ TEGINISEEA LNHLGEIGTK TTLRYSVQLL TPANLLAKIN GKDSIEKEHV EEISELFYDA KSSAKILADQ QDKYMK UniProtKB: RuvB-like 1 |
-分子 #7: RUVBL2
| 分子 | 名称: RUVBL2 / タイプ: protein_or_peptide / ID: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MATVTATTKV PEIRDVTRIE RIGAHSHIRG LGLDDALEPR QASQGMVGQL AARRAAGVVL EMIREGKIAG RAVLIAGQPG TGKTAIAMGM AQALGPDTPF TAIAGSEIFS LEMSKTEALT QAFRRSIGVR IKEETEIIEG EVVEIQIDRP ATGTGSKVGK LTLKTTEMET ...文字列: MATVTATTKV PEIRDVTRIE RIGAHSHIRG LGLDDALEPR QASQGMVGQL AARRAAGVVL EMIREGKIAG RAVLIAGQPG TGKTAIAMGM AQALGPDTPF TAIAGSEIFS LEMSKTEALT QAFRRSIGVR IKEETEIIEG EVVEIQIDRP ATGTGSKVGK LTLKTTEMET IYDLGTKMIE SLTKDKVQAG DVITIDKATG KISKLGRSFT RARDYDAMGS QTKFVQCPDG ELQKRKEVVH TVSLHEIDVI NSRTQGFLAL FSGDTGEIKS EVREQINAKV AEWREEGKAE IIPGVLFIDE VHMLDIESFS FLNRALESDM APVLIMATNR GITRIRGTSY QSPHGIPIDL LDRLLIVSTT PYSEKDTKQI LRIRCEEEDV EMSEDAYTVL TRIGLETSLR YAIQLITAAS LVCRKRKGTE VQVDDIKRVY SLFLDESRST QYMKEYQDAF LFNELKGETM DTS UniProtKB: RuvB-like 2 |
-分子 #8: ACTIN
| 分子 | 名称: ACTIN / タイプ: protein_or_peptide / ID: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDDDIAALVV DNGSGMCKAG FAGDDAPRAV FPSIVGRPRH QGVMVGMGQK DSYVGDEAQS KRGILTLKYP IEHGIVTNWD DMEKIWHHTF YNELRVAPEE HPVLLTEAPL NPKANREKMT QIMFETFNTP AMYVAIQAVL SLYASGRTTG IVMDSGDGVT HTVPIYEGYA ...文字列: MDDDIAALVV DNGSGMCKAG FAGDDAPRAV FPSIVGRPRH QGVMVGMGQK DSYVGDEAQS KRGILTLKYP IEHGIVTNWD DMEKIWHHTF YNELRVAPEE HPVLLTEAPL NPKANREKMT QIMFETFNTP AMYVAIQAVL SLYASGRTTG IVMDSGDGVT HTVPIYEGYA LPHAILRLDL AGRDLTDYLM KILTERGYSF TTTAEREIVR DIKEKLCYVA LDFEQEMATA ASSSSLEKSY ELPDGQVITI GNERFRCPEA LFQPSFLGME SCGIHETTFN SIMKCDVDIR KDLYANTVLS GGTTMYPGIA DRMQKEITAL APSTMKIKII APPERKYSVW IGGSILASLS TFQQMWISKQ EYDESGPSIV HRKCF UniProtKB: Actin, cytoplasmic 1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 279 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.2 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)