+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the KdpFABC complex in a nucleotide-free E1 conformation loaded with K+ | ||||||||||||||||||
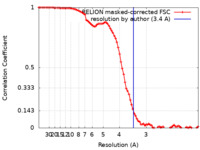
 マップデータ マップデータ | Cryo-EM map of the nucleotide-free KdpFABC complex, at 3.4 A resolution, sharpened at -113 A2. Data obtained in the presence of 50 mM KCl | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | P-type ATPase / superfamily of K+ transporters (SKT) / potassium uptake system / off-cycle post-albers conformation / MEMBRANE PROTEIN | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報P-type K+ transporter / P-type potassium transmembrane transporter activity / potassium:proton antiporter complex / potassium ion-transporting ATPase complex / monoatomic cation transmembrane transport / potassium ion binding / potassium ion transmembrane transport / potassium ion transport / magnesium ion binding / ATP hydrolysis activity ...P-type K+ transporter / P-type potassium transmembrane transporter activity / potassium:proton antiporter complex / potassium ion-transporting ATPase complex / monoatomic cation transmembrane transport / potassium ion binding / potassium ion transmembrane transport / potassium ion transport / magnesium ion binding / ATP hydrolysis activity / ATP binding / plasma membrane 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Hielkema L / Stock C / Silberberg JM / Corey RA / Wunnicke D / Stansfeld PJ / Haenelt I / Paulino C | ||||||||||||||||||
| 資金援助 |  オランダ, オランダ,  ドイツ, ドイツ,  英国, 5件 英国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2022 ジャーナル: Elife / 年: 2022タイトル: Inhibited KdpFABC transitions into an E1 off-cycle state. 著者: Jakob M Silberberg / Charlott Stock / Lisa Hielkema / Robin A Corey / Jan Rheinberger / Dorith Wunnicke / Victor R A Dubach / Phillip J Stansfeld / Inga Hänelt / Cristina Paulino /    要旨: KdpFABC is a high-affinity prokaryotic K uptake system that forms a functional chimera between a channel-like subunit (KdpA) and a P-type ATPase (KdpB). At high K levels, KdpFABC needs to be ...KdpFABC is a high-affinity prokaryotic K uptake system that forms a functional chimera between a channel-like subunit (KdpA) and a P-type ATPase (KdpB). At high K levels, KdpFABC needs to be inhibited to prevent excessive K accumulation to the point of toxicity. This is achieved by a phosphorylation of the serine residue in the TGES motif in the A domain of the pump subunit KdpB (KdpB). Here, we explore the structural basis of inhibition by KdpB phosphorylation by determining the conformational landscape of KdpFABC under inhibiting and non-inhibiting conditions. Under turnover conditions, we identified a new inhibited KdpFABC state that we termed E1P tight, which is not part of the canonical Post-Albers transport cycle of P-type ATPases. It likely represents the biochemically described stalled E1P state adopted by KdpFABC upon KdpB phosphorylation. The E1P tight state exhibits a compact fold of the three cytoplasmic domains and is likely adopted when the transition from high-energy E1P states to E2P states is unsuccessful. This study represents a structural characterization of a biologically relevant off-cycle state in the P-type ATPase family and supports the emerging discussion of P-type ATPase regulation by such states. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14914.map.gz emd_14914.map.gz | 49.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14914-v30.xml emd-14914-v30.xml emd-14914.xml emd-14914.xml | 25 KB 25 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14914_fsc.xml emd_14914_fsc.xml | 8.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14914.png emd_14914.png | 42.1 KB | ||
| マスクデータ |  emd_14914_msk_1.map emd_14914_msk_1.map | 52.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14914.cif.gz emd-14914.cif.gz | 7.7 KB | ||
| その他 |  emd_14914_half_map_1.map.gz emd_14914_half_map_1.map.gz emd_14914_half_map_2.map.gz emd_14914_half_map_2.map.gz | 40.9 MB 40.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14914 http://ftp.pdbj.org/pub/emdb/structures/EMD-14914 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14914 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14914 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14914_validation.pdf.gz emd_14914_validation.pdf.gz | 896.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14914_full_validation.pdf.gz emd_14914_full_validation.pdf.gz | 895.8 KB | 表示 | |
| XML形式データ |  emd_14914_validation.xml.gz emd_14914_validation.xml.gz | 14 KB | 表示 | |
| CIF形式データ |  emd_14914_validation.cif.gz emd_14914_validation.cif.gz | 19.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14914 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14914 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14914 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14914 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zrhMC  7zrdC  7zreC  7zrgC  7zriC  7zrjC  7zrkC  7zrlC  7zrmC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14914.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14914.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of the nucleotide-free KdpFABC complex, at 3.4 A resolution, sharpened at -113 A2. Data obtained in the presence of 50 mM KCl | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.012 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14914_msk_1.map emd_14914_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2 used during refinement and for...
| ファイル | emd_14914_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 used during refinement and for FSC gold-standard resolution calculation of the nucleotide-free KdpFABC complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1 used during refinement and for...
| ファイル | emd_14914_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 used during refinement and for FSC gold-standard resolution calculation of the nucleotide-free KdpFABC complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : KdpFABC
| 全体 | 名称: KdpFABC |
|---|---|
| 要素 |
|
-超分子 #1: KdpFABC
| 超分子 | 名称: KdpFABC / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 157 KDa |
-分子 #1: Potassium-transporting ATPase potassium-binding subunit
| 分子 | 名称: Potassium-transporting ATPase potassium-binding subunit タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 59.218613 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAAQGFLLIA TFLLVLMVLA RPLGSGLARL INDIPLPGTT GVERVLFRAL GVSDREMNWK QYLCAILGLN MLGLAVLFFM LLGQHYLPL NPQQLPGLSW DLALNTAVSF VTNTNWQSYS GETTLSYFSQ MAGLTVQNFL SAASGIAVIF ALIRAFTRQS M STLGNAWV ...文字列: MAAQGFLLIA TFLLVLMVLA RPLGSGLARL INDIPLPGTT GVERVLFRAL GVSDREMNWK QYLCAILGLN MLGLAVLFFM LLGQHYLPL NPQQLPGLSW DLALNTAVSF VTNTNWQSYS GETTLSYFSQ MAGLTVQNFL SAASGIAVIF ALIRAFTRQS M STLGNAWV DLLRITLWVL VPVALLIALF FIQQGALQNF LPYQAVNTVE GAQQLLPMGP VASQEAIKML GTNGGGFFNA NS SHPFENP TALTNFVQML AIFLIPTALC FAFGEVMGDR RQGRMLLWAM SVIFVICVGV VMWAEVQGNP HLLALGTDSS INM EGKESR FGVLVSSLFA VVTTAASCGA VIAMHDSFTA LGGMVPMWLM QIGEVVFGGV GSGLYGMMLF VLLAVFIAGL MIGR TPEYL GKKIDVREMK LTALAILVTP TLVLMGAALA MMTDAGRSAM LNPGPHGFSE VLYAVSSAAN NNGSAFAGLS ANSPF WNCL LAFCMFVGRF GVIIPVMAIA GSLVSKKSQA ASSGTLPTHG PLFVGLLIGT VLLVGALTFI PALALGPVAE YLS UniProtKB: Potassium-transporting ATPase potassium-binding subunit |
-分子 #2: Potassium-transporting ATPase KdpC subunit
| 分子 | 名称: Potassium-transporting ATPase KdpC subunit / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 20.281035 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGLRPALST FIFLLLITGG VYPLLTTVLG QWWFPWQANG SLIREGDTVR GSALIGQNFT GNGYFHGRPS ATAEMPYNPQ ASGGSNLAV SNPELDKLIA ARVAALRAAN PDASASVPVE LVTASASGLD NNITPQAAAW QIPRVAKARN LSVEQLTQLI A KYSQQPLV ...文字列: MSGLRPALST FIFLLLITGG VYPLLTTVLG QWWFPWQANG SLIREGDTVR GSALIGQNFT GNGYFHGRPS ATAEMPYNPQ ASGGSNLAV SNPELDKLIA ARVAALRAAN PDASASVPVE LVTASASGLD NNITPQAAAW QIPRVAKARN LSVEQLTQLI A KYSQQPLV KYIGQPVVNI VELNLALDKL DE UniProtKB: Potassium-transporting ATPase KdpC subunit |
-分子 #3: Potassium-transporting ATPase KdpF subunit
| 分子 | 名称: Potassium-transporting ATPase KdpF subunit / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 2.853463 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSAGVITGVL LVFLLLGYLV YALINAE UniProtKB: Potassium-transporting ATPase KdpF subunit |
-分子 #4: Potassium-transporting ATPase ATP-binding subunit
| 分子 | 名称: Potassium-transporting ATPase ATP-binding subunit / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO / EC番号: P-type K+ transporter |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 72.346859 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSRKQLALFE PTLVVQALKE AVKKLNPQAQ WRNPVMFIVW IGSLLTTCIS IAMASGAMPG NALFSAAISG WLWITVLFAN FAEALAEGR SKAQANSLKG VKKTAFARKL REPKYGAAAD KVPADQLRKG DIVLVEAGDI IPCDGEVIEG GASVDESAIT G E(SEP) ...文字列: MSRKQLALFE PTLVVQALKE AVKKLNPQAQ WRNPVMFIVW IGSLLTTCIS IAMASGAMPG NALFSAAISG WLWITVLFAN FAEALAEGR SKAQANSLKG VKKTAFARKL REPKYGAAAD KVPADQLRKG DIVLVEAGDI IPCDGEVIEG GASVDESAIT G E(SEP)APVIRE SGGDFASVTG GTRILSDWLV IECSVNPGET FLDRMIAMVE GAQRRKTPNE IALTILLIAL TIVFLLAT A TLWPFSAWGG NAVSVTVLVA LLVCLIPTTI GGLLSAIGVA GMSRMLGANV IATSGRAVEA AGDVDVLLLN KTGTITLGN RQASEFIPAQ GVDEKTLADA AQLASLADET PEGRSIVILA KQRFNLRERD VQSLHATFVP FTAQSRMSGI NIDNRMIRKG SVDAIRRHV EANGGHFPTD VDQKVDQVAR QGATPLVVVE GSRVLGVIAL KDIVKGGIKE RFAQLRKMGI KTVMITGDNR L TAAAIAAE AGVDDFLAEA TPEAKLALIR QYQAEGRLVA MTGDGTNDAP ALAQADVAVA MNSGTQAAKE AGNMVDLDSN PT KLIEVVH IGKQMLMTRG SLTTFSIAND VAKYFAIIPA AFAATYPQLN ALNIMCLHSP DSAILSAVIF NALIIVFLIP LAL KGVSYK PLTASAMLRR NLWIYGLGGL LVPFIGIKVI DLLLTVCGLV UniProtKB: Potassium-transporting ATPase ATP-binding subunit |
-分子 #5: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 5 / コピー数: 7 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-分子 #6: CARDIOLIPIN
| 分子 | 名称: CARDIOLIPIN / タイプ: ligand / ID: 6 / コピー数: 2 / 式: CDL |
|---|---|
| 分子量 | 理論値: 1.464043 KDa |
| Chemical component information |  ChemComp-CDL: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 10 mM Tris-HCl pH 8, 10 mM MgCl2, 10 mM NaCl and 0.0125% DDM |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 詳細: at 5 mA |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 温度 | 最低: 90.0 K / 最高: 105.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-60 / 撮影したグリッド数: 19 / 実像数: 17889 / 平均露光時間: 9.0 sec. / 平均電子線量: 52.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 2.0 µm / 最小 デフォーカス(補正後): 0.5 µm / 倍率(補正後): 49407 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 49407 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)