+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-14368 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of USP9X | |||||||||
 マップデータ マップデータ | unsharpened map used for model refinement | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | USP9X / Deubiquitinase / ubiquitin / Structural Genomics / Structural Genomics Consortium / SGC / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytosolic ciliogenesis / K11-linked deubiquitinase activity / protein import into peroxisome matrix, receptor recycling / protein deubiquitination involved in ubiquitin-dependent protein catabolic process / co-SMAD binding / female gamete generation / monoubiquitinated protein deubiquitination / positive regulation of TORC2 signaling / deubiquitinase activity / DNA alkylation repair ...cytosolic ciliogenesis / K11-linked deubiquitinase activity / protein import into peroxisome matrix, receptor recycling / protein deubiquitination involved in ubiquitin-dependent protein catabolic process / co-SMAD binding / female gamete generation / monoubiquitinated protein deubiquitination / positive regulation of TORC2 signaling / deubiquitinase activity / DNA alkylation repair / axon extension / K48-linked deubiquitinase activity / protein K63-linked deubiquitination / K63-linked deubiquitinase activity / RHOV GTPase cycle / RHOU GTPase cycle / molecular sequestering activity / cilium assembly / protein deubiquitination / positive regulation of protein binding / BMP signaling pathway / negative regulation of proteasomal ubiquitin-dependent protein catabolic process / cysteine-type peptidase activity / transforming growth factor beta receptor signaling pathway / Synthesis of active ubiquitin: roles of E1 and E2 enzymes / chromosome segregation / Peroxisomal protein import / Downregulation of SMAD2/3:SMAD4 transcriptional activity / regulation of circadian rhythm / regulation of protein stability / neuron migration / rhythmic process / intracellular protein localization / cell migration / growth cone / amyloid fibril formation / ubiquitinyl hydrolase 1 / cysteine-type deubiquitinase activity / Ub-specific processing proteases / protein stabilization / protein ubiquitination / cilium / Amyloid fiber formation / cell division / cysteine-type endopeptidase activity / centrosome / negative regulation of transcription by RNA polymerase II / nucleus / membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
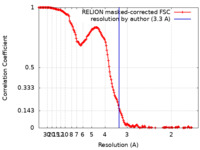
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Deme JC / Halabelian L | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of USP9X 著者: Halabelian L / Deme JC / Lea SM / Arrowsmith CH / Structural Genomics Consortium (SGC) | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14368.map.gz emd_14368.map.gz | 171.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14368-v30.xml emd-14368-v30.xml emd-14368.xml emd-14368.xml | 20.5 KB 20.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14368_fsc.xml emd_14368_fsc.xml | 15.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14368.png emd_14368.png | 119.8 KB | ||
| マスクデータ |  emd_14368_msk_1.map emd_14368_msk_1.map | 343 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14368.cif.gz emd-14368.cif.gz | 7.4 KB | ||
| その他 |  emd_14368_additional_1.map.gz emd_14368_additional_1.map.gz emd_14368_half_map_1.map.gz emd_14368_half_map_1.map.gz emd_14368_half_map_2.map.gz emd_14368_half_map_2.map.gz | 17.4 MB 318.2 MB 318.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14368 http://ftp.pdbj.org/pub/emdb/structures/EMD-14368 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14368 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14368 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14368_validation.pdf.gz emd_14368_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14368_full_validation.pdf.gz emd_14368_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_14368_validation.xml.gz emd_14368_validation.xml.gz | 23.8 KB | 表示 | |
| CIF形式データ |  emd_14368_validation.cif.gz emd_14368_validation.cif.gz | 31.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14368 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14368 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14368 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14368 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14368.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14368.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map used for model refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.832 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
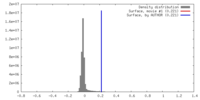
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_14368_msk_1.map emd_14368_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
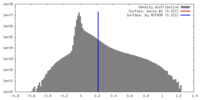
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_14368_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
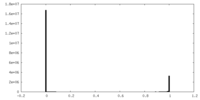
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_14368_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
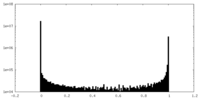
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_14368_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : trimer of USP9X
| 全体 | 名称: trimer of USP9X |
|---|---|
| 要素 |
|
-超分子 #1: trimer of USP9X
| 超分子 | 名称: trimer of USP9X / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Probable ubiquitin carboxyl-terminal hydrolase FAF-X
| 分子 | 名称: Probable ubiquitin carboxyl-terminal hydrolase FAF-X タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO / EC番号: ubiquitinyl hydrolase 1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 293.878844 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHHSSG RENLYFQGMT ATTRGSPVGG NDNQGQAPDG QSQPPLQQNQ TSSPDSSNEN SPATPPDEQG QGDAPPQLED EEPAFPHTD LAKLDDMINR PRWVVPVLPK GELEVLLEAA IDLSKKGLDV KSEACQRFFR DGLTISFTKI LTDEAVSGWK F EIHRCIIN ...文字列: MHHHHHHSSG RENLYFQGMT ATTRGSPVGG NDNQGQAPDG QSQPPLQQNQ TSSPDSSNEN SPATPPDEQG QGDAPPQLED EEPAFPHTD LAKLDDMINR PRWVVPVLPK GELEVLLEAA IDLSKKGLDV KSEACQRFFR DGLTISFTKI LTDEAVSGWK F EIHRCIIN NTHRLVELCV AKLSQDWFPL LELLAMALNP HCKFHIYNGT RPCESVSSSV QLPEDELFAR SPDPRSPKGW LV DLLNKFG TLNGFQILHD RFINGSALNV QIIAALIKPF GQCYEFLTLH TVKKYFLPII EMVPQFLENL TDEELKKEAK NEA KNDALS MIIKSLKNLA SRVPGQEETV KNLEIFRLKM ILRLLQISSF NGKMNALNEV NKVISSVSYY THRHGNPEEE EWLT AERMA EWIQQNNILS IVLRDSLHQP QYVEKLEKIL RFVIKEKALT LQDLDNIWAA QAGKHEAIVK NVHDLLAKLA WDFSP EQLD HLFDCFKASW TNASKKQREK LLELIRRLAE DDKDGVMAHK VLNLLWNLAH SDDVPVDIMD LALSAHIKIL DYSCSQ DRD TQKIQWIDRF IEELRTNDKW VIPALKQIRE ICSLFGEAPQ NLSQTQRSPH VFYRHDLINQ LQHNHALVTL VAENLAT YM ESMRLYARDH EDYDPQTVRL GSRYSHVQEV QERLNFLRFL LKDGQLWLCA PQAKQIWKCL AENAVYLCDR EACFKWYS K LMGDEPDLDP DINKDFFESN VLQLDPSLLT ENGMKCFERF FKAVNCREGK LVAKRRAYMM DDLELIGLDY LWRVVIQSN DDIASRAIDL LKEIYTNLGP RLQVNQVVIH EDFIQSCFDR LKASYDTLCV LDGDKDSVNC ARQEAVRMVR VLTVLREYIN ECDSDYHEE RTILPMSRAF RGKHLSFVVR FPNQGRQVDD LEVWSHTNDT IGSVRRCILN RIKANVAHTK IELFVGGELI D PADDRKLI GQLNLKDKSL ITAKLTQISS NMPSSPDSSS DSSTGSPGNH GNHYSDGPNP EVESCLPGVI MSLHPRYISF LW QVADLGS SLNMPPLRDG ARVLMKLMPP DSTTIEKLRA ICLDHAKLGE SSLSPSLDSL FFGPSASQVL YLTEVVYALL MPA GAPLAD DSSDFQFHFL KSGGLPLVLS MLTRNNFLPN ADMETRRGAY LNALKIAKLL LTAIGYGHVR AVAEACQPGV EGVN PMTQI NQVTHDQAVV LQSALQSIPN PSSECMLRNV SVRLAQQISD EASRYMPDIC VIRAIQKIIW ASGCGSLQLV FSPNE EITK IYEKTNAGNE PDLEDEQVCC EALEVMTLCF ALIPTALDAL SKEKAWQTFI IDLLLHCHSK TVRQVAQEQF FLMCTR CCM GHRPLLFFIT LLFTVLGSTA RERAKHSGDY FTLLRHLLNY AYNSNINVPN AEVLLNNEID WLKRIRDDVK RTGETGI EE TILEGHLGVT KELLAFQTSE KKFHIGCEKG GANLIKELID DFIFPASNVY LQYMRNGELP AEQAIPVCGS PPTINAGF E LLVALAVGCV RNLKQIVDSL TEMYYIGTAI TTCEALTEWE YLPPVGPRPP KGFVGLKNAG ATCYMNSVIQ QLYMIPSIR NGILAIEGTG SDVDDDMSGD EKQDNESNVD PRDDVFGYPQ QFEDKPALSK TEDRKEYNIG VLRHLQVIFG HLAASRLQYY VPRGFWKQF RLWGEPVNLR EQHDALEFFN SLVDSLDEAL KALGHPAMLS KVLGGSFADQ KICQGCPHRY ECEESFTTLN V DIRNHQNL LDSLEQYVKG DLLEGANAYH CEKCNKKVDT VKRLLIKKLP PVLAIQLKRF DYDWERECAI KFNDYFEFPR EL DMEPYTV AGVAKLEGDN VNPESQLIQQ SEQSESETAG STKYRLVGVL VHSGQASGGH YYSYIIQRNG GDGERNRWYK FDD GDVTEC KMDDDEEMKN QCFGGEYMGE VFDHMMKRMS YRRQKRWWNA YILFYERMDT IDQDDELIRY ISELAITTRP HQII MPSAI ERSVRKQNVQ FMHNRMQYSM EYFQFMKKLL TCNGVYLNPP PGQDHLLPEA EEITMISIQL AARFLFTTGF HTKKV VRGS ASDWYDALCI LLRHSKNVRF WFAHNVLFNV SNRFSEYLLE CPSAEVRGAF AKLIVFIAHF SLQDGPCPSP FASPGP SSQ AYDNLSLSDH LLRAVLNLLR REVSEHGRHL QQYFNLFVMY ANLGVAEKTQ LLKLSVPATF MLVSLDEGPG PPIKYQY AE LGKLYSVVSQ LIRCCNVSSR MQSSINGNPP LPNPFGDPNL SQPIMPIQQN VADILFVRTS YVKKIIEDCS NSEETVKL L RFCCWENPQF SSTVLSELLW QVAYSYTYEL RPYLDLLLQI LLIEDSWQTH RIHNALKGIP DDRDGLFDTI QRSKNHYQK RAYQCIKCMV ALFSNCPVAY QILQGNGDLK RKWTWAVEWL GDELERRPYT GNPQYTYNNW SPPVQSNETS NGYFLERSHS ARMTLAKAC ELCPEEEPDD QDAPDEHESP PPEDAPLYPH SPGSQYQQNN HVHGQPYTGP AAHHMNNPQR TGQRAQENYE G SEEVSPPQ TKDQDYKDDD K UniProtKB: Ubiquitin carboxyl-terminal hydrolase 9X |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 56.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)