+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13985 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
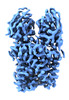
| タイトル | Cryo-EM map of apo-EleNRMT in complex with two nanobodies at 3.5A | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SLC11 / Magnesium / LeuT fold / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 | NRAMP family / Natural resistance-associated macrophage protein-like / cadmium ion transmembrane transporter activity / manganese ion transmembrane transporter activity / iron ion transmembrane transport / plasma membrane / Divalent metal cation transporter 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Eggerthella lenta (バクテリア) / synthetic construct (人工物) Eggerthella lenta (バクテリア) / synthetic construct (人工物) | |||||||||
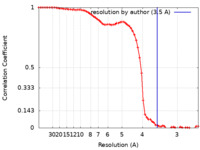
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Ramanadane K / Straub MS | |||||||||
| 資金援助 |  スイス, 1件 スイス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2022 ジャーナル: Elife / 年: 2022タイトル: Structural and functional properties of a magnesium transporter of the SLC11/NRAMP family. 著者: Karthik Ramanadane / Monique S Straub / Raimund Dutzler / Cristina Manatschal /  要旨: Members of the ubiquitous SLC11/NRAMP family catalyze the uptake of divalent transition metal ions into cells. They have evolved to efficiently select these trace elements from a large pool of Ca and ...Members of the ubiquitous SLC11/NRAMP family catalyze the uptake of divalent transition metal ions into cells. They have evolved to efficiently select these trace elements from a large pool of Ca and Mg, which are both orders of magnitude more abundant, and to concentrate them in the cytoplasm aided by the cotransport of H serving as energy source. In the present study, we have characterized a member of a distant clade of the family found in prokaryotes, termed NRMTs, that were proposed to function as transporters of Mg. The protein transports Mg and Mn but not Ca by a mechanism that is not coupled to H. Structures determined by cryo-EM and X-ray crystallography revealed a generally similar protein architecture compared to classical NRAMPs, with a restructured ion binding site whose increased volume provides suitable interactions with ions that likely have retained much of their hydration shell. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13985.map.gz emd_13985.map.gz | 11.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13985-v30.xml emd-13985-v30.xml emd-13985.xml emd-13985.xml | 20.7 KB 20.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13985_fsc.xml emd_13985_fsc.xml | 8.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13985.png emd_13985.png | 135.8 KB | ||
| マスクデータ |  emd_13985_msk_1.map emd_13985_msk_1.map | 22.2 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-13985.cif.gz emd-13985.cif.gz | 6.2 KB | ||
| その他 |  emd_13985_half_map_1.map.gz emd_13985_half_map_1.map.gz emd_13985_half_map_2.map.gz emd_13985_half_map_2.map.gz | 20.7 MB 20.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13985 http://ftp.pdbj.org/pub/emdb/structures/EMD-13985 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13985 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13985 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13985_validation.pdf.gz emd_13985_validation.pdf.gz | 751.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13985_full_validation.pdf.gz emd_13985_full_validation.pdf.gz | 751.4 KB | 表示 | |
| XML形式データ |  emd_13985_validation.xml.gz emd_13985_validation.xml.gz | 12 KB | 表示 | |
| CIF形式データ |  emd_13985_validation.cif.gz emd_13985_validation.cif.gz | 16.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13985 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13985 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13985 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13985 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13985.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13985.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.302 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
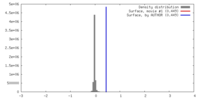
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_13985_msk_1.map emd_13985_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
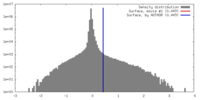
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_13985_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
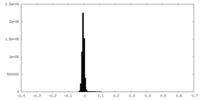
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_13985_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : EleNRMT in complex with two nanobodies
| 全体 | 名称: EleNRMT in complex with two nanobodies |
|---|---|
| 要素 |
|
-超分子 #1: EleNRMT in complex with two nanobodies
| 超分子 | 名称: EleNRMT in complex with two nanobodies / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 分子量 | 理論値: 0.013 kDa/nm |
-超分子 #2: EleNRMT
| 超分子 | 名称: EleNRMT / タイプ: organelle_or_cellular_component / ID: 2 / 親要素: 1 / 含まれる分子: #1 / 詳細: thermostabilized version |
|---|---|
| 由来(天然) | 生物種:  Eggerthella lenta (バクテリア) Eggerthella lenta (バクテリア) |
-超分子 #3: Nanobody 1
| 超分子 | 名称: Nanobody 1 / タイプ: organelle_or_cellular_component / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-超分子 #4: Nanobody 2
| 超分子 | 名称: Nanobody 2 / タイプ: organelle_or_cellular_component / ID: 4 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: Divalent metal cation transporter
| 分子 | 名称: Divalent metal cation transporter / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Eggerthella lenta (バクテリア) Eggerthella lenta (バクテリア) |
| 分子量 | 理論値: 46.848828 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MMMKKNEKLE LRDVAVDAAE STELVEVPEK KQPKKIWLLL AALGPGIVTA MAGNDAGGIS TYSTVGAKFG FATLWVIPIM CVLLIVVQM TAARMGAVTG KGFAALIRER FGIRLTALAM LALLIGNVAT TFSEFAGIAS GMEMFGVSKY LSVPVAAVAV W LLVVGGSY ...文字列: MMMKKNEKLE LRDVAVDAAE STELVEVPEK KQPKKIWLLL AALGPGIVTA MAGNDAGGIS TYSTVGAKFG FATLWVIPIM CVLLIVVQM TAARMGAVTG KGFAALIRER FGIRLTALAM LALLIGNVAT TFSEFAGIAS GMEMFGVSKY LSVPVAAVAV W LLVVGGSY KRVEKVFLIL SLVFVTYIVA AFMAQPNWEE ALTSTVVPHI VNDQSFVSLV IAMIGTTIAP WMMFFNQSNV VE KGVTVKD LFSQKVDVVA GTIAACLVAW FIIVTTGAVL FPQGIEIESA ADAARALAPF AGHYAEALFA IGLIAASFLA ACV LPLTTA FVICEAFGWE AGVSFKWKEA PLFKSIFTFV IAFSAVVVLI PNIDLMGVML TAQFVNGLIL PVLLVFMAII AADK RVMGA YRSRIVSRVL IWLTVGIVTV LTAALLVMQV LGI UniProtKB: Divalent metal cation transporter |
-分子 #2: Nanobody 1
| 分子 | 名称: Nanobody 1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 13.12676 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QLQLVESGGG LVQPGGSLRL SCEASGKVFM INAMGWYRQA PGKQRELVAF ISRRGNINYA DSVKGRFTIS RDNAKNTVYL QMNSLRPED TAIYYCSADP RSNLDDGRYW GKGTPVTVSS |
-分子 #3: Nanobody 2
| 分子 | 名称: Nanobody 2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 13.767339 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QLQLVESGGG LVLAGGSLRL SCAASVRTFS HYALGWFRQA PGKEREFVAA IRWTGSSANY ADSVKGRFTI SRDNAKNTVD LRMNSLKPE DTAVYYCAAR TVYRPGFEDP NEYAYWGQGT RVTVSS |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 3 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 3 / 実像数: 22117 / 平均露光時間: 1.01 sec. / 平均電子線量: 69.725 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)