+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1367 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Three-dimensional structure of a voltage-gated potassium channel at 2.5 nm resolution. | |||||||||
 マップデータ マップデータ | This is an image of surface rendered view of Shaker potassium voltage-gated channel | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報delayed rectifier potassium channel activity / action potential / voltage-gated potassium channel activity / voltage-gated potassium channel complex / protein homooligomerization / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 25.0 Å | |||||||||
 データ登録者 データ登録者 | Sokolova O / Kolmakova-Partensky L / Grigorieff N | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2001 ジャーナル: Structure / 年: 2001タイトル: Three-dimensional structure of a voltage-gated potassium channel at 2.5 nm resolution. 著者: O Sokolova / L Kolmakova-Partensky / N Grigorieff /  要旨: BACKGROUND: The voltage-gated potassium channel Shaker from Drosophila consists of a tetramer of identical subunits, each containing six transmembrane segments. The atomic structure of a bacterial ...BACKGROUND: The voltage-gated potassium channel Shaker from Drosophila consists of a tetramer of identical subunits, each containing six transmembrane segments. The atomic structure of a bacterial homolog, the potassium channel KcsA, is much smaller than Shaker. It does not have a voltage sensor and other important domains like the N-terminal tetramerization (T1) domain. The structure of these additional elements has to be studied in the more complex voltage-gated channels. RESULTS: We determined the three-dimensional structure of the entire Shaker channel at 2.5 nm resolution using electron microscopy. The four-fold symmetric structure shows a large and a small domain ...RESULTS: We determined the three-dimensional structure of the entire Shaker channel at 2.5 nm resolution using electron microscopy. The four-fold symmetric structure shows a large and a small domain linked by thin 2 nm long connectors. To interpret the structure, we used the crystal structures of the isolated T1 domain and the KcsA channel. A unique density assignment was made based on the symmetry and dimensions of the crystal structures and domains, identifying the smaller domain as the cytoplasmic mass of Shaker containing T1 and the larger domain as embedded in the membrane. CONCLUSIONS: The two-domain architecture of the Shaker channel is consistent with the recently proposed "hanging gondola" model for the T1 domain, putting the T1 domain at a distance from the ...CONCLUSIONS: The two-domain architecture of the Shaker channel is consistent with the recently proposed "hanging gondola" model for the T1 domain, putting the T1 domain at a distance from the membrane domain but attached to it by thin connectors. The space between the two domains is sufficient to permit cytoplasmic access of ions and the N-terminal inactivation domain to the pore region. A hanging gondola architecture has also been observed in the nicotinic acetylcholine receptor and the KcsA structure, suggesting that it is a common element of ion channels. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1367.map.gz emd_1367.map.gz | 908.6 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1367-v30.xml emd-1367-v30.xml emd-1367.xml emd-1367.xml | 9.8 KB 9.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1367.gif 1367.gif | 47.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1367 http://ftp.pdbj.org/pub/emdb/structures/EMD-1367 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1367 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1367 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1367_validation.pdf.gz emd_1367_validation.pdf.gz | 206.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1367_full_validation.pdf.gz emd_1367_full_validation.pdf.gz | 206 KB | 表示 | |
| XML形式データ |  emd_1367_validation.xml.gz emd_1367_validation.xml.gz | 4.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1367 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1367 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1367 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1367 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1367.map.gz / 形式: CCP4 / 大きさ: 1001 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1367.map.gz / 形式: CCP4 / 大きさ: 1001 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is an image of surface rendered view of Shaker potassium voltage-gated channel | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
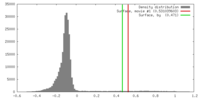
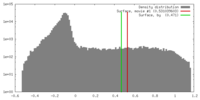
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Shaker B channel
| 全体 | 名称: Shaker B channel |
|---|---|
| 要素 |
|
-超分子 #1000: Shaker B channel
| 超分子 | 名称: Shaker B channel / タイプ: sample / ID: 1000 詳細: This is an inactivation-removed Shaker B channel construct, delta6-46/F425G, with a C-terminal 1D4 immunoaffinity tag. The molecular weight given above includes glycosylation. 集合状態: One homotetramer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 860 KDa / 理論値: 860 KDa / 手法: SDS PAGE electrophoresis |
-分子 #1: Kv1.1
| 分子 | 名称: Kv1.1 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Shaker Kv channel / コピー数: 4 / 集合状態: Tetramer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  別称: Drosophila / 組織: Muscule / 細胞: COS-1 / Organelle: Cell membranes / 細胞中の位置: Cell membranes |
| 分子量 | 実験値: 860 KDa / 理論値: 860 KDa |
| 組換発現 | 生物種: Mammalian (両生類) / 組換プラスミド: pmt3 |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 詳細: 80 mM KCl, 2 mM NaEDTA, 40 mM HEPES-KOH, 0.7% CHAPS, 1 mM DTT, 50 mM NaCl, 0.2 mM leupeptin/pepstatin,1 mM PMSF |
|---|---|
| 染色 | タイプ: NEGATIVE 詳細: Grids floated on drops of sample buffer twice with blotting inbetween, than floated on 1% solution of uranyl acetate twice (blot inbetween) for 10 seconds each |
| グリッド | 詳細: Continuous carbon on 400 mesh copper grid |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM120T |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 100,000 times magnification |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 3.5 µm / 実像数: 40 / 平均電子線量: 10 e/Å2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 1.9 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダー: Side-entry room temperature / 試料ホルダーモデル: OTHER |
- 画像解析
画像解析
| 詳細 | Manual selection of particles |
|---|---|
| CTF補正 | 詳細: Each particle |
| 最終 再構成 | 想定した対称性 - 点群: C4 (4回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 25.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: IMAGIC, FREALIGN / 使用した粒子像数: 6000 |
| 最終 2次元分類 | クラス数: 50 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)