+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12652 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Respiratory complex I from Escherichia coli - focused refinement of membrane arm | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NADH:ubiquinone reductase (H+-translocating) / oxidative phosphorylation / ELECTRON TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報トランスロカーゼ; ヒドロンの輸送の触媒; 酸化還元酵素反応を伴う / NADH dehydrogenase (quinone) (non-electrogenic) activity / NADH dehydrogenase complex / oxidoreductase activity, acting on NAD(P)H, quinone or similar compound as acceptor / ubiquinone binding / electron transport coupled proton transport / NADH dehydrogenase activity / respiratory chain complex I / NADH dehydrogenase (ubiquinone) activity / membrane => GO:0016020 ...トランスロカーゼ; ヒドロンの輸送の触媒; 酸化還元酵素反応を伴う / NADH dehydrogenase (quinone) (non-electrogenic) activity / NADH dehydrogenase complex / oxidoreductase activity, acting on NAD(P)H, quinone or similar compound as acceptor / ubiquinone binding / electron transport coupled proton transport / NADH dehydrogenase activity / respiratory chain complex I / NADH dehydrogenase (ubiquinone) activity / membrane => GO:0016020 / quinone binding / ATP synthesis coupled electron transport / endomembrane system / proton transmembrane transport / aerobic respiration / respiratory electron transport chain / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
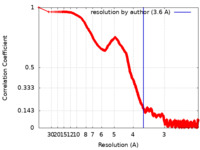
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Kolata P / Efremov RG | |||||||||
| 資金援助 |  ベルギー, 1件 ベルギー, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2021 ジャーナル: Elife / 年: 2021タイトル: Structure of respiratory complex I reconstituted into lipid nanodiscs reveals an uncoupled conformation. 著者: Piotr Kolata / Rouslan G Efremov /  要旨: Respiratory complex I is a multi-subunit membrane protein complex that reversibly couples NADH oxidation and ubiquinone reduction with proton translocation against transmembrane potential. Complex I ...Respiratory complex I is a multi-subunit membrane protein complex that reversibly couples NADH oxidation and ubiquinone reduction with proton translocation against transmembrane potential. Complex I from is among the best functionally characterized complexes, but its structure remains unknown, hindering further studies to understand the enzyme coupling mechanism. Here, we describe the single particle cryo-electron microscopy (cryo-EM) structure of the entire catalytically active complex I reconstituted into lipid nanodiscs. The structure of this mesophilic bacterial complex I displays highly dynamic connection between the peripheral and membrane domains. The peripheral domain assembly is stabilized by unique terminal extensions and an insertion loop. The membrane domain structure reveals novel dynamic features. Unusual conformation of the conserved interface between the peripheral and membrane domains suggests an uncoupled conformation of the complex. Considering constraints imposed by the structural data, we suggest a new simple hypothetical coupling mechanism for the molecular machine. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12652.map.gz emd_12652.map.gz | 4.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12652-v30.xml emd-12652-v30.xml emd-12652.xml emd-12652.xml | 27.2 KB 27.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12652_fsc.xml emd_12652_fsc.xml | 267.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12652.png emd_12652.png | 128.4 KB | ||
| Filedesc metadata |  emd-12652.cif.gz emd-12652.cif.gz | 7.7 KB | ||
| その他 |  emd_12652_half_map_1.map.gz emd_12652_half_map_1.map.gz emd_12652_half_map_2.map.gz emd_12652_half_map_2.map.gz | 98.6 MB 98.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12652 http://ftp.pdbj.org/pub/emdb/structures/EMD-12652 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12652 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12652 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12652.map.gz / 形式: CCP4 / 大きさ: 5.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12652.map.gz / 形式: CCP4 / 大きさ: 5.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
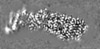
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1568 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #2
| ファイル | emd_12652_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
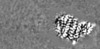
| 投影像・断面図 |
| ||||||||||||
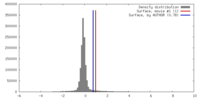
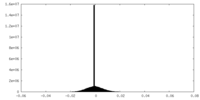
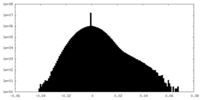
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_12652_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
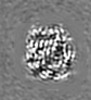
| 投影像・断面図 |
| ||||||||||||
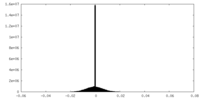
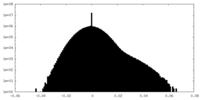
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Respiratory complex I from Escherichia coli - focused refinement ...
| 全体 | 名称: Respiratory complex I from Escherichia coli - focused refinement of membrane arm |
|---|---|
| 要素 |
|
-超分子 #1: Respiratory complex I from Escherichia coli - focused refinement ...
| 超分子 | 名称: Respiratory complex I from Escherichia coli - focused refinement of membrane arm タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 260 KDa |
-分子 #1: NADH-quinone oxidoreductase subunit A
| 分子 | 名称: NADH-quinone oxidoreductase subunit A / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号: トランスロカーゼ; ヒドロンの輸送の触媒; 酸化還元酵素反応を伴う |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 16.474283 KDa |
| 配列 | 文字列: MSMSTSTEVI AHHWAFAIFL IVAIGLCCLM LVGGWFLGGR ARARSKNVPF ESGIDSVGSA RLRLSAKFYL VAMFFVIFDV EALYLFAWS TSIRESGWVG FVEAAIFIFV LLAGLVYLVR IGALDWTPAR SRRERMNPET NSIANRQR UniProtKB: NADH-quinone oxidoreductase subunit A |
-分子 #2: NADH-quinone oxidoreductase subunit H
| 分子 | 名称: NADH-quinone oxidoreductase subunit H / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO EC番号: トランスロカーゼ; ヒドロンの輸送の触媒; 酸化還元酵素反応を伴う |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 36.240922 KDa |
| 配列 | 文字列: MSWISPELIE ILLTILKAVV ILLVVVTCGA FMSFGERRLL GLFQNRYGPN RVGWGGSLQL VADMIKMFFK EDWIPKFSDR VIFTLAPMI AFTSLLLAFA IVPVSPGWVV ADLNIGILFF LMMAGLAVYA VLFAGWSSNN KYSLLGAMRA SAQTLSYEVF L GLSLMGVV ...文字列: MSWISPELIE ILLTILKAVV ILLVVVTCGA FMSFGERRLL GLFQNRYGPN RVGWGGSLQL VADMIKMFFK EDWIPKFSDR VIFTLAPMI AFTSLLLAFA IVPVSPGWVV ADLNIGILFF LMMAGLAVYA VLFAGWSSNN KYSLLGAMRA SAQTLSYEVF L GLSLMGVV AQAGSFNMTD IVNSQAHVWN VIPQFFGFIT FAIAGVAVCH RHPFDQPEAE QELADGYHIE YSGMKFGLFF VG EYIGIVT ISALMVTLFF GGWQGPLLPP FIWFALKTAF FMMMFILIRA SLPRPRYDQV MSFGWKICLP LTLINLLVTA AVI LWQAQ UniProtKB: NADH-quinone oxidoreductase subunit H |
-分子 #3: NADH-quinone oxidoreductase subunit J
| 分子 | 名称: NADH-quinone oxidoreductase subunit J / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO EC番号: トランスロカーゼ; ヒドロンの輸送の触媒; 酸化還元酵素反応を伴う |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 19.889551 KDa |
| 配列 | 文字列: MEFAFYICGL IAILATLRVI THTNPVHALL YLIISLLAIS GVFFSLGAYF AGALEIIVYA GAIMVLFVFV VMMLNLGGSE IEQERQWLK PQVWIGPAIL SAIMLVVIVY AILGVNDQGI DGTPISAKAV GITLFGPYVL AVELASMLLL AGLVVAFHVG R EERAGEVL SNRKDDSAKR KTEEHA UniProtKB: NADH-quinone oxidoreductase subunit J |
-分子 #4: NADH-quinone oxidoreductase subunit K
| 分子 | 名称: NADH-quinone oxidoreductase subunit K / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO / EC番号: NADH:ubiquinone reductase (H+-translocating) |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 10.852961 KDa |
| 配列 | 文字列: MIPLQHGLIL AAILFVLGLT GLVIRRNLLF MLIGLEIMIN ASALAFVVAG SYWGQTDGQV MYILAISLAA AEASIGLALL LQLHRRRQN LNIDSVSEMR G UniProtKB: NADH-quinone oxidoreductase subunit K |
-分子 #5: NADH-quinone oxidoreductase subunit L
| 分子 | 名称: NADH-quinone oxidoreductase subunit L / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO / EC番号: NADH dehydrogenase (quinone) |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 66.513633 KDa |
| 配列 | 文字列: MNMLALTIIL PLIGFVLLAF SRGRWSENVS AIVGVGSVGL AALVTAFIGV DFFANGEQTY SQPLWTWMSV GDFNIGFNLV LDGLSLTML SVVTGVGFLI HMYASWYMRG EEGYSRFFAY TNLFIASMVV LVLADNLLLM YLGWEGVGLC SYLLIGFYYT D PKNGAAAM ...文字列: MNMLALTIIL PLIGFVLLAF SRGRWSENVS AIVGVGSVGL AALVTAFIGV DFFANGEQTY SQPLWTWMSV GDFNIGFNLV LDGLSLTML SVVTGVGFLI HMYASWYMRG EEGYSRFFAY TNLFIASMVV LVLADNLLLM YLGWEGVGLC SYLLIGFYYT D PKNGAAAM KAFVVTRVGD VFLAFALFIL YNELGTLNFR EMVELAPAHF ADGNNMLMWA TLMLLGGAVG KSAQLPLQTW LA DAMAGPT PVSALIHAAT MVTAGVYLIA RTHGLFLMTP EVLHLVGIVG AVTLLLAGFA ALVQTDIKRV LAYSTMSQIG YMF LALGVQ AWDAAIFHLM THAFFKALLF LASGSVILAC HHEQNIFKMG GLRKSIPLVY LCFLVGGAAL SALPLVTAGF FSKD EILAG AMANGHINLM VAGLVGAFMT SLYTFRMIFI VFHGKEQIHA HAVKGVTHSL PLIVLLILST FVGALIVPPL QGVLP QTTE LAHGSMLTLE ITSGVVAVVG ILLAAWLWLG KRTLVTSIAN SAPGRLLSTW WYNAWGFDWL YDKVFVKPFL GIAWLL KRD PLNSMMNIPA VLSRFAGKGL LLSENGYLRW YVASMSIGAV VVLALLMVLR UniProtKB: NADH-quinone oxidoreductase subunit L |
-分子 #6: NADH-quinone oxidoreductase subunit M
| 分子 | 名称: NADH-quinone oxidoreductase subunit M / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO EC番号: トランスロカーゼ; ヒドロンの輸送の触媒; 酸化還元酵素反応を伴う |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 56.56009 KDa |
| 配列 | 文字列: MLLPWLILIP FIGGFLCWQT ERFGVKVPRW IALITMGLTL ALSLQLWLQG GYSLTQSAGI PQWQSEFDMP WIPRFGISIH LAIDGLSLL MVVLTGLLGV LAVLCSWKEI EKYQGFFHLN LMWILGGVIG VFLAIDMFLF FFFWEMMLVP MYFLIALWGH K ASDGKTRI ...文字列: MLLPWLILIP FIGGFLCWQT ERFGVKVPRW IALITMGLTL ALSLQLWLQG GYSLTQSAGI PQWQSEFDMP WIPRFGISIH LAIDGLSLL MVVLTGLLGV LAVLCSWKEI EKYQGFFHLN LMWILGGVIG VFLAIDMFLF FFFWEMMLVP MYFLIALWGH K ASDGKTRI TAATKFFIYT QASGLVMLIA ILALVFVHYN ATGVWTFNYE ELLNTPMSSG VEYLLMLGFF IAFAVKMPVV PL HGWLPDA HSQAPTAGSV DLAGILLKTA AYGLLRFSLP LFPNASAEFA PIAMWLGVIG IFYGAWMAFA QTDIKRLIAY TSV SHMGFV LIAIYTGSQL AYQGAVIQMI AHGLSAAGLF ILCGQLYERI HTRDMRMMGG LWSKMKWLPA LSLFFAVATL GMPG TGNFV GEFMILFGSF QVVPVITVIS TFGLVFASVY SLAMLHRAYF GKAKSQIASQ ELPGMSLREL FMILLLVVLL VLLGF YPQP ILDTSHSAIG NIQQWFVNSV TTTRP UniProtKB: NADH-quinone oxidoreductase subunit M |
-分子 #7: NADH-quinone oxidoreductase subunit N
| 分子 | 名称: NADH-quinone oxidoreductase subunit N / タイプ: protein_or_peptide / ID: 7 / コピー数: 1 / 光学異性体: LEVO EC番号: トランスロカーゼ; ヒドロンの輸送の触媒; 酸化還元酵素反応を伴う |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 52.072672 KDa |
| 配列 | 文字列: MTITPQNLIA LLPLLIVGLT VVVVMLSIAW RRNHFLNATL SVIGLNAALV SLWFVGQAGA MDVTPLMRVD GFAMLYTGLV LLASLATCT FAYPWLEGYN DNKDEFYLLV LIAALGGILL ANANHLASLF LGIELISLPL FGLVGYAFRQ KRSLEASIKY T ILSAAASS ...文字列: MTITPQNLIA LLPLLIVGLT VVVVMLSIAW RRNHFLNATL SVIGLNAALV SLWFVGQAGA MDVTPLMRVD GFAMLYTGLV LLASLATCT FAYPWLEGYN DNKDEFYLLV LIAALGGILL ANANHLASLF LGIELISLPL FGLVGYAFRQ KRSLEASIKY T ILSAAASS FLLFGMALVY AQSGDLSFVA LGKNLGDGML NEPLLLAGFG LMIVGLGFKL SLVPFHLWTP DVYQGAPAPV ST FLATASK IAIFGVVMRL FLYAPVGDSE AIRVVLAIIA FASIIFGNLM ALSQTNIKRL LGYSSISHLG YLLVALIALQ TGE MSMEAV GVYLAGYLFS SLGAFGVVSL MSSPYRGPDA DSLFSYRGLF WHRPILAAVM TVMMLSLAGI PMTLGFIGKF YVLA VGVQA HLWWLVGAVV VGSAIGLYYY LRVAVSLYLH APEQPGRDAP SNWQYSAGGI VVLISALLVL VLGVWPQPLI SIVRL AMPL M UniProtKB: NADH-quinone oxidoreductase subunit N |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6.8 構成要素:
詳細: The buffer was used for gel filtration of protein reconstituted in lipid nanodiscs | ||||||||||||
| グリッド | モデル: Quantifoil R0.6/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 支持フィルム - Film thickness: 12 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 240 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.028 kPa | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 97 % / チャンバー内温度: 296 K / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 9122 / 平均露光時間: 3.0 sec. / 平均電子線量: 64.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.55 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 75 / 当てはまり具合の基準: Correlation coefficient | ||||||
| 得られたモデル |  PDB-7nyh: |
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)