+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12369 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of the human Separase-Securin complex | |||||||||
 マップデータ マップデータ | Postprocessed map of human Separase-Securin complex at 2.9A. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | pseudosubstrate HEAT repeat caspase cell cycle / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of sister chromatid cohesion / separase / meiotic chromosome separation / mitotic sister chromatid separation / homologous chromosome segregation / establishment of mitotic spindle localization / meiotic spindle organization / positive regulation of mitotic metaphase/anaphase transition / mitotic cytokinesis / mitotic sister chromatid segregation ...negative regulation of sister chromatid cohesion / separase / meiotic chromosome separation / mitotic sister chromatid separation / homologous chromosome segregation / establishment of mitotic spindle localization / meiotic spindle organization / positive regulation of mitotic metaphase/anaphase transition / mitotic cytokinesis / mitotic sister chromatid segregation / catalytic activity / cysteine-type endopeptidase inhibitor activity / chromosome organization / cysteine-type peptidase activity / APC/C:Cdc20 mediated degradation of Securin / molecular function activator activity / APC/C:Cdh1 mediated degradation of Cdc20 and other APC/C:Cdh1 targeted proteins in late mitosis/early G1 / SH3 domain binding / Separation of Sister Chromatids / mitotic spindle / spermatogenesis / cell division / cysteine-type endopeptidase activity / DNA repair / apoptotic process / centrosome / proteolysis / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Yu J / Raia P | |||||||||
| 資金援助 |  スイス, スイス,  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: Structural basis of human separase regulation by securin and CDK1-cyclin B1. 著者: Jun Yu / Pierre Raia / Chloe M Ghent / Tobias Raisch / Yashar Sadian / Simone Cavadini / Pramod M Sabale / David Barford / Stefan Raunser / David O Morgan / Andreas Boland /     要旨: In early mitosis, the duplicated chromosomes are held together by the ring-shaped cohesin complex. Separation of chromosomes during anaphase is triggered by separase-a large cysteine endopeptidase ...In early mitosis, the duplicated chromosomes are held together by the ring-shaped cohesin complex. Separation of chromosomes during anaphase is triggered by separase-a large cysteine endopeptidase that cleaves the cohesin subunit SCC1 (also known as RAD21). Separase is activated by degradation of its inhibitors, securin and cyclin B, but the molecular mechanisms of separase regulation are not clear. Here we used cryogenic electron microscopy to determine the structures of human separase in complex with either securin or CDK1-cyclin B1-CKS1. In both complexes, separase is inhibited by pseudosubstrate motifs that block substrate binding at the catalytic site and at nearby docking sites. As in Caenorhabditis elegans and yeast, human securin contains its own pseudosubstrate motifs. By contrast, CDK1-cyclin B1 inhibits separase by deploying pseudosubstrate motifs from intrinsically disordered loops in separase itself. One autoinhibitory loop is oriented by CDK1-cyclin B1 to block the catalytic sites of both separase and CDK1. Another autoinhibitory loop blocks substrate docking in a cleft adjacent to the separase catalytic site. A third separase loop contains a phosphoserine that promotes complex assembly by binding to a conserved phosphate-binding pocket in cyclin B1. Our study reveals the diverse array of mechanisms by which securin and CDK1-cyclin B1 bind and inhibit separase, providing the molecular basis for the robust control of chromosome segregation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12369.map.gz emd_12369.map.gz | 104.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12369-v30.xml emd-12369-v30.xml emd-12369.xml emd-12369.xml | 21.7 KB 21.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_12369.png emd_12369.png | 91 KB | ||
| Filedesc metadata |  emd-12369.cif.gz emd-12369.cif.gz | 7.3 KB | ||
| その他 |  emd_12369_additional_1.map.gz emd_12369_additional_1.map.gz emd_12369_additional_2.map.gz emd_12369_additional_2.map.gz emd_12369_additional_3.map.gz emd_12369_additional_3.map.gz | 96.1 MB 104.6 MB 96.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12369 http://ftp.pdbj.org/pub/emdb/structures/EMD-12369 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12369 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12369 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12369_validation.pdf.gz emd_12369_validation.pdf.gz | 344.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12369_full_validation.pdf.gz emd_12369_full_validation.pdf.gz | 344.4 KB | 表示 | |
| XML形式データ |  emd_12369_validation.xml.gz emd_12369_validation.xml.gz | 6.5 KB | 表示 | |
| CIF形式データ |  emd_12369_validation.cif.gz emd_12369_validation.cif.gz | 7.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12369 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12369 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12369 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12369 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12369.map.gz / 形式: CCP4 / 大きさ: 122.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12369.map.gz / 形式: CCP4 / 大きさ: 122.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Postprocessed map of human Separase-Securin complex at 2.9A. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
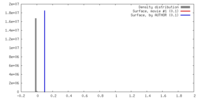
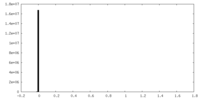
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Unsharpened map of human Separase-Securin complex at 2.9A.
| ファイル | emd_12369_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map of human Separase-Securin complex at 2.9A. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
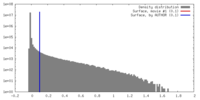
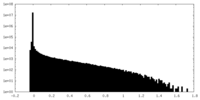
| 密度ヒストグラム |
-追加マップ: Focus-refined map (postprocessed) of human Separase-Securin complex at...
| ファイル | emd_12369_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focus-refined map (postprocessed) of human Separase-Securin complex at 2.9A. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
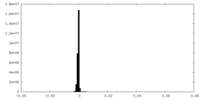
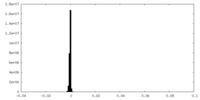
| 密度ヒストグラム |
-追加マップ: Focus-refined map (unsharpened) of human Separase-Securin complex at...
| ファイル | emd_12369_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focus-refined map (unsharpened) of human Separase-Securin complex at 2.9A. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
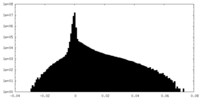
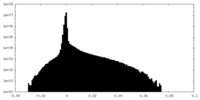
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Inhibitory complex of human separase bound to securin.
| 全体 | 名称: Inhibitory complex of human separase bound to securin. |
|---|---|
| 要素 |
|
-超分子 #1: Inhibitory complex of human separase bound to securin.
| 超分子 | 名称: Inhibitory complex of human separase bound to securin. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Separin
| 分子 | 名称: Separin / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: separase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 237.610469 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRSFKRVNFG TLLSSQKEAE ELLPDLKEFL SNPPAGFPSS RSDAERRQAC DAILRACNQQ LTAKLACPRH LGSLLELAEL ACDGYLVST PQRPPLYLER ILFVLLRNAA AQGSPEVTLR LAQPLHACLV QCSREAAPQD YEAVARGSFS LLWKGAEALL E RRAAFAAR ...文字列: MRSFKRVNFG TLLSSQKEAE ELLPDLKEFL SNPPAGFPSS RSDAERRQAC DAILRACNQQ LTAKLACPRH LGSLLELAEL ACDGYLVST PQRPPLYLER ILFVLLRNAA AQGSPEVTLR LAQPLHACLV QCSREAAPQD YEAVARGSFS LLWKGAEALL E RRAAFAAR LKALSFLVLL EDESTPCEVP HFASPTACRA VAAHQLFDAS GHGLNEADAD FLDDLLSRHV IRALVGERGS SS GLLSPQR ALCLLELTLE HCRRFCWSRH HDKAISAVEK AHSYLRNTNL APSLQLCQLG VKLLQVGEEG PQAVAKLLIK ASA VLSKSM EAPSPPLRAL YESCQFFLSG LERGTKRRYR LDAILSLFAF LGGYCSLLQQ LRDDGVYGGS SKQQQSFLQM YFQG LHLYT VVVYDFAQGC QIVDLADLTQ LVDSCKSTVV WMLEALEGLS GQELTDHMGM TASYTSNLAY SFYSHKLYAE ACAIS EPLC QHLGLVKPGT YPEVPPEKLH RCFRLQVESL KKLGKQAQGC KMVILWLAAL QPCSPEHMAE PVTFWVRVKM DAARAG DKE LQLKTLRDSL SGWDPETLAL LLREELQAYK AVRADTGQER FNIICDLLEL SPEETPAGAW ARATHLVELA QVLCYHD FT QQTNCSALDA IREALQLLDS VRPEAQARDQ LLDDKAQALL WLYICTLEAK IQEGIERDRR AQAPGNLEEF EVNDLNYE D KLQEDRFLYS NIAFNLAADA AQSKCLDQAL ALWKELLTKG QAPAVRCLQQ TAASLQILAA LYQLVAKPMQ ALEVLLLLR IVSERLKDHS KAAGSSCHIT QLLLTLGCPS YAQLHLEEAA SSLKHLDQTT DTYLLLSLTC DLLRSQLYWT HQKVTKGVSL LLSVLRDPA LQKSSKAWYL LRVQVLQLVA AYLSLPSNNL SHSLWEQLCA QGWQTPEIAL IDSHKLLRSI ILLLMGSDIL S TQKAAVET SFLDYGENLV QKWQVLSEVL SCSEKLVCHL GRLGSVSEAK AFCLEALKLT TKLQIPRQCA LFLVLKGELE LA RNDIDLC QSDLQQVLFL LESCTEFGGV TQHLDSVKKV HLQKGKQQAQ VPCPPQLPEE ELFLRGPALE LVATVAKEPG PIA PSTNSS PVLKTKPQPI PNFLSHSPTC DCSLCASPVL TAVCLRWVLV TAGVRLAMGH QAQGLDLLQV VLKGCPEAAE RLTQ ALQAS LNHKTPPSLV PSLLDEILAQ AYTLLALEGL NQPSNESLQK VLQSGLKFVA ARIPHLEPWR ASLLLIWALT KLGGL SCCT TQLFASSWGW QPPLIKSVPG SEPSKTQGQK RSGRGRQKLA SAPLSLNNTS QKGLEGRGLP CTPKPPDRIR QAGPHV PFT VFEEVCPTES KPEVPQAPRV QQRVQTRLKV NFSDDSDLED PVSAEAWLAE EPKRRGTASR GRGRARKGLS LKTDAVV AP GSAPGNPGLN GRSRRAKKVA SRHCEERRPQ RASDQARPGP EIMRTIPEEE LTDNWRKMSF EILRGSDGED SASGGKTP A PGPEAASGEW ELLRLDSSKK KLPSPCPDKE SDKDLGPRLQ LPSAPVATGL STLDSICDSL SVAFRGISHC PPSGLYAHL CRFLALCLGH RDPYATAFLV TESVSITCRH QLLTHLHRQL SKAQKHRGSL EIADQLQGLS LQEMPGDVPL ARIQRLFSFR ALESGHFPQ PEKESFQERL ALIPSGVTVC VLALATLQPG TVGNTLLLTR LEKDSPPVSV QIPTGQNKLH LRSVLNEFDA I QKAQKENS SCTDKREWWT GRLALDHRME VLIASLEKSV LGCWKGLLLP SSEEPGPAQE ASRLQELLQD CGWKYPDRTL LK IMLSGAG ALTPQDIQAL AYGLCPTQPE RAQELLNEAV GRLQGLTVPS NSHLVLVLDK DLQKLPWESM PSLQALPVTR LPS FRFLLS YSIIKEYGAS PVLSQGVDPR STFYVLNPHN NLSSTEEQFR ANFSSEAGWR GVVGEVPRPE QVQEALTKHD LYIY AGHGA GARFLDGQAV LRLSCRAVAL LFGCSSAALA VHGNLEGAGI VLKYIMAGCP LFLGNLWDVT DRDIDRYTEA LLQGW LGAG PGAPLLYYVN QARQAPRLKY LIGAAPIAYG LPVSLRSSLA EENLYFQSWS HPQFEKGGGS GGGSGGGSWS HPQFEK UniProtKB: Separin |
-分子 #2: Securin
| 分子 | 名称: Securin / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.05234 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MATLIYVDKE NGEPGTRVVA KDGLKLGSGP SIKALDGRSQ VSTPRFGKTF DAPPALPKAT RKALGTVNRA TEKSVKTKGP LKQKQPSFS AKKMTEKTVK AKSSVPASDD AYPEIEKFFP FNPLDFESFD LPEEHQIAHL PLSGVPLMIL DEERELEKLF Q LGPPSPVK ...文字列: MATLIYVDKE NGEPGTRVVA KDGLKLGSGP SIKALDGRSQ VSTPRFGKTF DAPPALPKAT RKALGTVNRA TEKSVKTKGP LKQKQPSFS AKKMTEKTVK AKSSVPASDD AYPEIEKFFP FNPLDFESFD LPEEHQIAHL PLSGVPLMIL DEERELEKLF Q LGPPSPVK MPSPPWESNL LQSPSSILST LDVELPPVCC DIDI UniProtKB: Securin |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.025 mg/mL |
|---|---|
| 緩衝液 | pH: 7.8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 293 K / 装置: LEICA EM GP |
| 詳細 | The sample was monodisperse. We use graphene oxide-coated EM grids. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 4 / 実像数: 16540 / 平均露光時間: 3.0 sec. / 平均電子線量: 67.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 1.3 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.3 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 2.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 使用した粒子像数: 205300 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7nj1: |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)