+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12291 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In situ structure of the myosin double-head bound to a thin filament in the rigor state from mouse psoas muscle | ||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 生物種 |  | ||||||||||||||||||
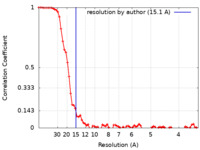
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 15.1 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Wang Z / Grange M / Wagner T / Kho AL / Gautel M / Raunser S | ||||||||||||||||||
| 資金援助 |  ドイツ, ドイツ,  英国, European Union, 5件 英国, European Union, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2021 ジャーナル: Cell / 年: 2021タイトル: The molecular basis for sarcomere organization in vertebrate skeletal muscle. 著者: Zhexin Wang / Michael Grange / Thorsten Wagner / Ay Lin Kho / Mathias Gautel / Stefan Raunser /   要旨: Sarcomeres are force-generating and load-bearing devices of muscles. A precise molecular picture of how sarcomeres are built underpins understanding their role in health and disease. Here, we ...Sarcomeres are force-generating and load-bearing devices of muscles. A precise molecular picture of how sarcomeres are built underpins understanding their role in health and disease. Here, we determine the molecular architecture of native vertebrate skeletal sarcomeres by electron cryo-tomography. Our reconstruction reveals molecular details of the three-dimensional organization and interaction of actin and myosin in the A-band, I-band, and Z-disc and demonstrates that α-actinin cross-links antiparallel actin filaments by forming doublets with 6-nm spacing. Structures of myosin, tropomyosin, and actin at ~10 Å further reveal two conformations of the "double-head" myosin, where the flexible orientation of the lever arm and light chains enable myosin not only to interact with the same actin filament, but also to split between two actin filaments. Our results provide unexpected insights into the fundamental organization of vertebrate skeletal muscle and serve as a strong foundation for future investigations of muscle diseases. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12291.map.gz emd_12291.map.gz | 4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12291-v30.xml emd-12291-v30.xml emd-12291.xml emd-12291.xml | 20.1 KB 20.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12291_fsc.xml emd_12291_fsc.xml | 9.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12291.png emd_12291.png | 104.9 KB | ||
| マスクデータ |  emd_12291_msk_1.map emd_12291_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| その他 |  emd_12291_half_map_1.map.gz emd_12291_half_map_1.map.gz emd_12291_half_map_2.map.gz emd_12291_half_map_2.map.gz | 23.4 MB 23.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12291 http://ftp.pdbj.org/pub/emdb/structures/EMD-12291 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12291 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12291 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12291_validation.pdf.gz emd_12291_validation.pdf.gz | 446.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12291_full_validation.pdf.gz emd_12291_full_validation.pdf.gz | 445.2 KB | 表示 | |
| XML形式データ |  emd_12291_validation.xml.gz emd_12291_validation.xml.gz | 13.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12291 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12291 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12291 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12291 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12291.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12291.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.755 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
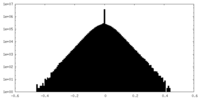
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_12291_msk_1.map emd_12291_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
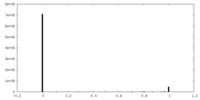
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_12291_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
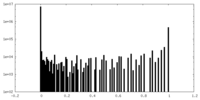
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_12291_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
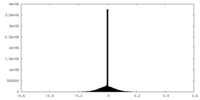
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : In situ myosin double head bound to a thin filament in the rigor ...
| 全体 | 名称: In situ myosin double head bound to a thin filament in the rigor state from mouse psoas muscle |
|---|---|
| 要素 |
|
-超分子 #1: In situ myosin double head bound to a thin filament in the rigor ...
| 超分子 | 名称: In situ myosin double head bound to a thin filament in the rigor state from mouse psoas muscle タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #2: actin
| 超分子 | 名称: actin / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: myosin double head
| 超分子 | 名称: myosin double head / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #4-#6 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #4: tropomyosin
| 超分子 | 名称: tropomyosin / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7 構成要素:
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 286 K / 装置: FEI VITROBOT MARK IV | ||||||||||||||||||
| 詳細 | The sample was myofibrils. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - 画像ごとのフレーム数: 1-8 / 平均電子線量: 3.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 28409 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)