+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12213 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of disease related M854K MDA5-dsRNA filament in complex with ATP | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | PROTEIN-RNA COMPLEX / HELICAL FILAMENT / ATPASE / INNATE IMMUNE RECEPTOR / IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報MDA-5 signaling pathway / positive regulation of response to cytokine stimulus / Ub-specific processing proteases / negative regulation of viral genome replication / type I interferon-mediated signaling pathway / pattern recognition receptor activity / cellular response to exogenous dsRNA / protein complex oligomerization / positive regulation of interferon-alpha production / protein sumoylation ...MDA-5 signaling pathway / positive regulation of response to cytokine stimulus / Ub-specific processing proteases / negative regulation of viral genome replication / type I interferon-mediated signaling pathway / pattern recognition receptor activity / cellular response to exogenous dsRNA / protein complex oligomerization / positive regulation of interferon-alpha production / protein sumoylation / ribonucleoprotein complex binding / antiviral innate immune response / positive regulation of interferon-beta production / cellular response to virus / positive regulation of interleukin-6 production / response to virus / positive regulation of tumor necrosis factor production / double-stranded RNA binding / defense response to virus / single-stranded RNA binding / RNA helicase activity / RNA helicase / protein domain specific binding / innate immune response / ATP hydrolysis activity / mitochondrion / DNA binding / zinc ion binding / ATP binding / identical protein binding / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   Pseudomonas virus phi6 (ウイルス) Pseudomonas virus phi6 (ウイルス) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Singh R / Herrero del Valle A | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: MDA5 disease variant M854K prevents ATP-dependent structural discrimination of viral and cellular RNA. 著者: Qin Yu / Alba Herrero Del Valle / Rahul Singh / Yorgo Modis /   要旨: Our innate immune responses to viral RNA are vital defenses. Long cytosolic double-stranded RNA (dsRNA) is recognized by MDA5. The ATPase activity of MDA5 contributes to its dsRNA binding selectivity. ...Our innate immune responses to viral RNA are vital defenses. Long cytosolic double-stranded RNA (dsRNA) is recognized by MDA5. The ATPase activity of MDA5 contributes to its dsRNA binding selectivity. Mutations that reduce RNA selectivity can cause autoinflammatory disease. Here, we show how the disease-associated MDA5 variant M854K perturbs MDA5-dsRNA recognition. M854K MDA5 constitutively activates interferon signaling in the absence of exogenous RNA. M854K MDA5 lacks ATPase activity and binds more stably to synthetic Alu:Alu dsRNA. CryoEM structures of MDA5-dsRNA filaments at different stages of ATP hydrolysis show that the K854 sidechain forms polar bonds that constrain the conformation of MDA5 subdomains, disrupting key steps in the ATPase cycle- RNA footprint expansion and helical twist modulation. The M854K mutation inhibits ATP-dependent RNA proofreading via an allosteric mechanism, allowing MDA5 to form signaling complexes on endogenous RNAs. This work provides insights on how MDA5 recognizes dsRNA in health and disease. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12213.map.gz emd_12213.map.gz | 8.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12213-v30.xml emd-12213-v30.xml emd-12213.xml emd-12213.xml | 23.2 KB 23.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12213_fsc.xml emd_12213_fsc.xml | 10 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12213.png emd_12213.png | 183.5 KB | ||
| マスクデータ |  emd_12213_msk_1.map emd_12213_msk_1.map | 83.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-12213.cif.gz emd-12213.cif.gz | 7.5 KB | ||
| その他 |  emd_12213_half_map_1.map.gz emd_12213_half_map_1.map.gz emd_12213_half_map_2.map.gz emd_12213_half_map_2.map.gz | 65.4 MB 65.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12213 http://ftp.pdbj.org/pub/emdb/structures/EMD-12213 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12213 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12213 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12213_validation.pdf.gz emd_12213_validation.pdf.gz | 797 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12213_full_validation.pdf.gz emd_12213_full_validation.pdf.gz | 796.5 KB | 表示 | |
| XML形式データ |  emd_12213_validation.xml.gz emd_12213_validation.xml.gz | 16.9 KB | 表示 | |
| CIF形式データ |  emd_12213_validation.cif.gz emd_12213_validation.cif.gz | 22.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12213 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12213 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12213 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12213 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7bkpMC  7bkqC  7ngaC  7nicC  7niqC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10630 (タイトル: CryoEM structure of disease related M854K MDA5-dsRNA filament in complex with ATP EMPIAR-10630 (タイトル: CryoEM structure of disease related M854K MDA5-dsRNA filament in complex with ATPData size: 1.2 TB Data #1: Unaligned multi frame micrographs for the Cryo-EM structure of disease related M854K MDA5-dsRNA filament in complex with ATP [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12213.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12213.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
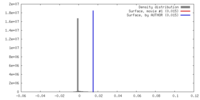
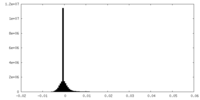
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_12213_msk_1.map emd_12213_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
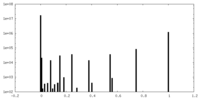
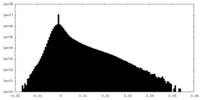
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_12213_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
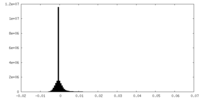
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_12213_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
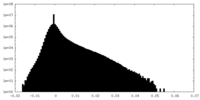
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : M854K MDA5-dsRNA filament in complex with ATP
| 全体 | 名称: M854K MDA5-dsRNA filament in complex with ATP |
|---|---|
| 要素 |
|
-超分子 #1: M854K MDA5-dsRNA filament in complex with ATP
| 超分子 | 名称: M854K MDA5-dsRNA filament in complex with ATP / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 分子量 | 理論値: 1.91 kDa/nm |
-超分子 #2: INTERFERON-INDUCED HELICASE C DOMAIN-CONTAINING PROTEIN
| 超分子 | 名称: INTERFERON-INDUCED HELICASE C DOMAIN-CONTAINING PROTEIN タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: RNA
| 超分子 | 名称: RNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas virus phi6 (ウイルス) Pseudomonas virus phi6 (ウイルス) |
-分子 #1: Interferon-induced helicase C domain-containing protein 1
| 分子 | 名称: Interferon-induced helicase C domain-containing protein 1 タイプ: protein_or_peptide / ID: 1 / 詳細: ATP / コピー数: 1 / 光学異性体: LEVO / EC番号: RNA helicase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 116.122359 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSIVCSAEDS FRNLILFFRP RLKMYIQVEP VLDHLIFLSA ETKEQILKKI NTCGNTSAAE LLLSTLEQGQ WPLGWTQMFV EALEHSGNP LAARYVKPTL TDLPSPSSET AHDECLHLLT LLQPTLVDKL LINDVLDTCF EKGLLTVEDR NRISAAGNSG N ESGVRELL ...文字列: MSIVCSAEDS FRNLILFFRP RLKMYIQVEP VLDHLIFLSA ETKEQILKKI NTCGNTSAAE LLLSTLEQGQ WPLGWTQMFV EALEHSGNP LAARYVKPTL TDLPSPSSET AHDECLHLLT LLQPTLVDKL LINDVLDTCF EKGLLTVEDR NRISAAGNSG N ESGVRELL RRIVQKENWF STFLDVLRQT GNDALFQELT GGGCPEDNTD LANSSHRDGP AANECLLPAV DESSLETEAW NV DDILPEA SCTDSSVTTE SDTSLAEGSV SCFDESLGHN SNMGRDSGTM GSDSDESVIQ TKRVSPEPEL QLRPYQMEVA QPA LDGKNI IICLPTGSGK TRVAVYITKD HLDKKKQASE SGKVIVLVNK VMLAEQLFRK EFNPYLKKWY RIIGLSGDTQ LKIS FPEVV KSYDVIISTA QILENSLLNL ESGDDDGVQL SDFSLIIIDE CHHTNKEAVY NNIMRRYLKQ KLRNNDLKKQ NKPAI PLPQ ILGLTASPGV GAAKKQSEAE KHILNICANL DAFTIKTVKE NLGQLKHQIK EPCKKFVIAD DTRENPFKEK LLEIMA SIQ TYCQKSPMSD FGTQHYEQWA IQMEKKAAKD GNRKDRVCAE HLRKYNEALQ INDTIRMIDA YSHLETFYTD EKEKKFA VL NDSDKSDDEA SSCNDQLKGD VKKSLKLDET DEFLMNLFFD NKKMLKKLAE NPKYENEKLI KLRNTILEQF TRSEESSR G IIFTKTRQST YALSQWIMEN AKFAEVGVKA HHLIGAGHSS EVKPMTQTEQ KEVISKFRTG EINLLIATTV AEEGLDIKE CNIVIRYGLV TNEIAMVQAR GRARADESTY VLVTSSGSGV TEREIVNDFR EKMKYKAINR VQNMKPEEYA HKILELQVQS ILEKKMKVK RSIAKQYNDN PSLITLLCKN CSMLVCSGEN IHVIEKMHHV NMTPEFKGLY IVRENKALQK KFADYQTNGE I ICKCGQAW GTMMVHKGLD LPCLKIRNFV VNFKNNSPKK QYKKWVELPI RFPDLDYSEY CLYSDED UniProtKB: Interferon-induced helicase C domain-containing protein 1 |
-分子 #2: RNA (5'-R(P*CP*UP*CP*UP*CP*CP*UP*CP*GP*GP*CP*UP*UP*G)-3')
| 分子 | 名称: RNA (5'-R(P*CP*UP*CP*UP*CP*CP*UP*CP*GP*GP*CP*UP*UP*G)-3') タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas virus phi6 (ウイルス) Pseudomonas virus phi6 (ウイルス) |
| 分子量 | 理論値: 4.35258 KDa |
| 配列 | 文字列: CUCUCCUCGG CUUG |
-分子 #3: RNA (5'-R(P*CP*AP*AP*GP*CP*CP*GP*AP*GP*GP*AP*GP*AP*G)-3')
| 分子 | 名称: RNA (5'-R(P*CP*AP*AP*GP*CP*CP*GP*AP*GP*GP*AP*GP*AP*G)-3') タイプ: rna / ID: 3 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas virus phi6 (ウイルス) Pseudomonas virus phi6 (ウイルス) |
| 分子量 | 理論値: 4.587852 KDa |
| 配列 | 文字列: CAAGCCGAGG AGAG |
-分子 #4: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 4 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #5: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.8 構成要素:
| |||||||||||||||
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 詳細: machine step 6 | |||||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.2 K / 装置: FEI VITROBOT MARK IV 詳細: Grids were blotted for 3 s; blot force 10, 5, 0, -5, -10. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 位相板: OTHER / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 実像数: 7147 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): -4.0 µm / 最小 デフォーカス(補正後): -0.25 µm / 照射モード: FLOOD BEAM / 撮影モード: DARK FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): -2.5 µm / 最小 デフォーカス(公称値): -1.2 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)