+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12138 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of fatty acid synthase (FAS) from Pichia pastoris | ||||||||||||
 マップデータ マップデータ | Map filtered according to local resolution. | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Multienzyme / Complex / Fatty acid / Synthase / BIOSYNTHETIC PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mitotic nuclear membrane biogenesis / palmitic acid biosynthetic process / fatty-acyl-CoA synthase system / fatty acid synthase complex / fatty-acyl-CoA synthase activity / [acyl-carrier-protein] S-acetyltransferase / [acyl-carrier-protein] S-acetyltransferase activity / holo-[acyl-carrier-protein] synthase activity / [acyl-carrier-protein] S-malonyltransferase / [acyl-carrier-protein] S-malonyltransferase activity ...mitotic nuclear membrane biogenesis / palmitic acid biosynthetic process / fatty-acyl-CoA synthase system / fatty acid synthase complex / fatty-acyl-CoA synthase activity / [acyl-carrier-protein] S-acetyltransferase / [acyl-carrier-protein] S-acetyltransferase activity / holo-[acyl-carrier-protein] synthase activity / [acyl-carrier-protein] S-malonyltransferase / [acyl-carrier-protein] S-malonyltransferase activity / 3-hydroxyacyl-[acyl-carrier-protein] dehydratase / beta-ketoacyl-[acyl-carrier-protein] synthase I / (3R)-hydroxyacyl-[acyl-carrier-protein] dehydratase activity / 3-oxoacyl-[acyl-carrier-protein] reductase / 3-oxoacyl-[acyl-carrier-protein] reductase (NADPH) activity / oleoyl-[acyl-carrier-protein] hydrolase / fatty acyl-[ACP] hydrolase activity / enoyl-[acyl-carrier-protein] reductase (NADH) / enoyl-[acyl-carrier-protein] reductase (NADH) activity / fatty acid synthase activity / 3-oxoacyl-[acyl-carrier-protein] synthase activity / fatty acid biosynthetic process / magnesium ion binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Komagataella phaffii GS115 (菌類) / Komagataella phaffii GS115 (菌類) /  Komagataella phaffii (strain GS115 / ATCC 20864) (菌類) Komagataella phaffii (strain GS115 / ATCC 20864) (菌類) | ||||||||||||
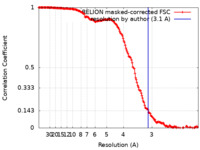
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | ||||||||||||
 データ登録者 データ登録者 | Snowden JS / Alzahrani J | ||||||||||||
| 資金援助 |  英国, 英国,  サウジアラビア, 3件 サウジアラビア, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Sci Rep / 年: 2021 ジャーナル: Sci Rep / 年: 2021タイトル: Structural insight into Pichia pastoris fatty acid synthase. 著者: Joseph S Snowden / Jehad Alzahrani / Lee Sherry / Martin Stacey / David J Rowlands / Neil A Ranson / Nicola J Stonehouse /  要旨: Type I fatty acid synthases (FASs) are critical metabolic enzymes which are common targets for bioengineering in the production of biofuels and other products. Serendipitously, we identified FAS as a ...Type I fatty acid synthases (FASs) are critical metabolic enzymes which are common targets for bioengineering in the production of biofuels and other products. Serendipitously, we identified FAS as a contaminant in a cryoEM dataset of virus-like particles (VLPs) purified from P. pastoris, an important model organism and common expression system used in protein production. From these data, we determined the structure of P. pastoris FAS to 3.1 Å resolution. While the overall organisation of the complex was typical of type I FASs, we identified several differences in both structural and enzymatic domains through comparison with the prototypical yeast FAS from S. cerevisiae. Using focussed classification, we were also able to resolve and model the mobile acyl-carrier protein (ACP) domain, which is key for function. Ultimately, the structure reported here will be a useful resource for further efforts to engineer yeast FAS for synthesis of alternate products. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12138.map.gz emd_12138.map.gz | 211.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12138-v30.xml emd-12138-v30.xml emd-12138.xml emd-12138.xml | 33 KB 33 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12138_fsc.xml emd_12138_fsc.xml | 16.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12138.png emd_12138.png | 216.3 KB | ||
| マスクデータ |  emd_12138_msk_1.map emd_12138_msk_1.map | 371.3 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-12138.cif.gz emd-12138.cif.gz | 8.8 KB | ||
| その他 |  emd_12138_additional_1.map.gz emd_12138_additional_1.map.gz emd_12138_additional_2.map.gz emd_12138_additional_2.map.gz emd_12138_additional_3.map.gz emd_12138_additional_3.map.gz emd_12138_additional_4.map.gz emd_12138_additional_4.map.gz emd_12138_half_map_1.map.gz emd_12138_half_map_1.map.gz emd_12138_half_map_2.map.gz emd_12138_half_map_2.map.gz | 295.8 MB 186 MB 348.6 MB 48 MB 296.9 MB 298.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12138 http://ftp.pdbj.org/pub/emdb/structures/EMD-12138 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12138 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12138 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12138_validation.pdf.gz emd_12138_validation.pdf.gz | 918.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12138_full_validation.pdf.gz emd_12138_full_validation.pdf.gz | 918.5 KB | 表示 | |
| XML形式データ |  emd_12138_validation.xml.gz emd_12138_validation.xml.gz | 23.8 KB | 表示 | |
| CIF形式データ |  emd_12138_validation.cif.gz emd_12138_validation.cif.gz | 31.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12138 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12138 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12138 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12138 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12138.map.gz / 形式: CCP4 / 大きさ: 371.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12138.map.gz / 形式: CCP4 / 大きさ: 371.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map filtered according to local resolution. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.065 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
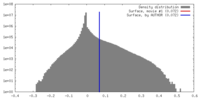
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_12138_msk_1.map emd_12138_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
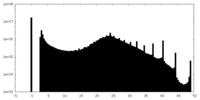
| 密度ヒストグラム |
-追加マップ: Unsharpened map.
| ファイル | emd_12138_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map. | ||||||||||||
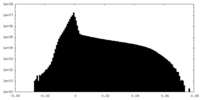
| 投影像・断面図 |
| ||||||||||||
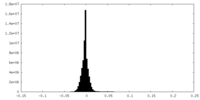
| 密度ヒストグラム |
-追加マップ: Map of local resolution values, for local resolution...
| ファイル | emd_12138_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map of local resolution values, for local resolution colouring of maps. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Sharpened map without solvent mask applied.
| ファイル | emd_12138_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map without solvent mask applied. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
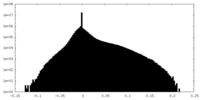
| 密度ヒストグラム |
-追加マップ: Sharpened map with solvent mask applied.
| ファイル | emd_12138_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map with solvent mask applied. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
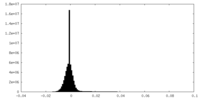
| 密度ヒストグラム |
-ハーフマップ: Half map 1.
| ファイル | emd_12138_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2.
| ファイル | emd_12138_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Fatty acid synthase
| 全体 | 名称: Fatty acid synthase |
|---|---|
| 要素 |
|
-超分子 #1: Fatty acid synthase
| 超分子 | 名称: Fatty acid synthase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Komagataella phaffii GS115 (菌類) Komagataella phaffii GS115 (菌類) |
| 分子量 | 理論値: 2.6 MDa |
-分子 #1: Fatty acid synthase subunit alpha
| 分子 | 名称: Fatty acid synthase subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: fatty-acyl-CoA synthase system |
|---|---|
| 由来(天然) | 生物種:  Komagataella phaffii (strain GS115 / ATCC 20864) (菌類) Komagataella phaffii (strain GS115 / ATCC 20864) (菌類)株: GS115 / ATCC 20864 |
| 分子量 | 理論値: 206.375734 KDa |
| 配列 | 文字列: MRPEVEQELS HVLLTELLAY QFASPVRWIE TQDVFLKDYN TERVVEIGPS PTLAGMASRT IKAKYESYDA ALSLQRQVLC YAKDTKEIY YTPDPADIAP PIKEEAETSA AATSSSAPAA AAPVSAAPAA APSGPVAEIP DEPVKAALVL HVLVAHKLKK S LDAVPLSK ...文字列: MRPEVEQELS HVLLTELLAY QFASPVRWIE TQDVFLKDYN TERVVEIGPS PTLAGMASRT IKAKYESYDA ALSLQRQVLC YAKDTKEIY YTPDPADIAP PIKEEAETSA AATSSSAPAA AAPVSAAPAA APSGPVAEIP DEPVKAALVL HVLVAHKLKK S LDAVPLSK AIKDLVGGKS TVQNEILGDL GKEFGSTPEK PEDTPLQELA EQFQDTFPGS LGKQTGSLVN RLMSSKMPGG FS LSVARKY LQTRWGLGPG RQDSVLLVAL VNEPGARLSS DGEAKEFLDS CAQKYASGAG ITLAQAAAGG AGSSGAGGAV IDA EAFEEL TKDNRVLARQ QLEVLARYLK YDLTKGEKSL VKEKEASSLL QQELDLWAEE HGEIYAQGIK PVFSHLKART YDSY WNWAR QDALSMYFDI IFGKLTDVDR ETVSQCIQLM NRSNPTLIKF MQYHIDHCPE YKGETYQLAK SLGQQLIDNC IQVAN QDPV YKDISYPTGP HTEVDSKGNI VYKEVNRKSV RKLEQYVFEM SQGGELTKEV QPTIQEDLAK IYEALNKQAS TESQLE FNK LYNSLIEFVE KSKEIEVSKS INAVLASKSS DSDRSAEISS LSEKTSIVDP VSGGIPPETV PFLHLKKKLP SGEWVFD RD TSALFLDGLQ KGAVNGISYK GKNVLITGAG AGSIGAEVLQ GLISGGAKVI VTTSRFSKKV TEYYQDIYAR FGAAGSCL I VVPFNQGSKQ DVEALIDYIY RDVKDEGLGW DLDAVIPFAA IPEAGIEIDE LGSKSELAHR IMLTNLLRLL GEVKKQKFT RAINTRPAQI ILPLSPNHGT FGSDGLYSES KLGLETLFNR WYSESWSEQL TVCGAIIGWT RGTGLMSGNN IIAEGLEKLG VRTFSQKEM AFNILGLMTP ELTEMCQNGP VVADLNGGLQ FIENLREYTA QLRNEIYETS EVRRAVSIET GIETRVVNGE N ADAPYQKA RIEPRANLKF EFPPLKSHKE IQNKAPGLEG LLDLERVIVV TGFGEVSPWG NTRTRWEMEA FGEFSIEGCL EM AWIMGFI KYHNGNLKGK PYTGWIDAKT NEPVEDKDIK KKYEEEILAH AGIRLIEPEL FRGYNPEKKE LIQEVIIEQD MAP FVTDES TAQQYKLQHE DAVDILKSEE SDEYTVTFKK GARLFVPKAL RFDRLVAGQI PTGWDAKRYG ISEDTISQVD PVTL YALVS TIEALLSAGI TDPYEFYKYV HVSEVGNCSG SGMGGVSALR GMFRDRYSDK PVQNDILQES FINTMSAWVN MLLLS SSGP IKTPVGACAT AVESVDIGVE TILSGKAKIC LVGGYDDFQE EGSYEFANMN ATSNSLDEFD HGRTPQEMSR PATTTR NGF MEAQGSGTQV IMNAELAIKM GVPIYAIVAL TATATDKIGR SVPAPGKGIL TTAREHHGSL KTKSPKLDIK YRTRQLN KR KDQIKQWVED ELEYIREEAA ELANSDAKFD AVSFVSERTE EVYREATKQV KMAQQEWGNE FWKNDPRIAP LRGALATF N LTVDDLGVAS FHGTSTKAND KNESITINKM MQHLGRSEGN PVFGVFQKYL TGHPKGAAGA WMLNGAIQIL QTGIVPGNR NADNVDKILE DFEYVLYPSR SIQTDGIKAC SVTSFGFGQK GGQAIVVHPD YLFASLDSET FEEYKTKVEA RYKSTYRYMH NAIIRNTMF VAKSDPPYTD ELEQPVYLDP LARVNNCKKN PSKLVFVNAD VQSKQNFVGK SANDTAKVIS SLTSDVTSGG K GVGVDVEL ISAINNENHT FIERNFTENE ISYCASAPSS KSSLAGTWSA KEAVFKALGV ESKGAGASLK DIEIVRDSKG AP TVVLHGD AKSAASAAGV KNVKVSISHD DVQSVAVAIS EF UniProtKB: Fatty acid synthase subunit alpha |
-分子 #2: Fatty acid synthase subunit beta
| 分子 | 名称: Fatty acid synthase subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: fatty-acyl-CoA synthase system |
|---|---|
| 由来(天然) | 生物種:  Komagataella phaffii (strain GS115 / ATCC 20864) (菌類) Komagataella phaffii (strain GS115 / ATCC 20864) (菌類)株: GS115 / ATCC 20864 |
| 分子量 | 理論値: 230.000625 KDa |
| 配列 | 文字列: MSATSGVVNR PLVLNHGSIE STILIPTTEY HFYQTLLEGF RKSLPQVTEG FADDDEPSSK AELLMKFLGY IVQSGVSNQQ EQLAAAKLV LNEFESRFLQ GLNLHSYAAI LLKSETFPTT LLKIKENLIK NYYLGRALVY LPGQRGLVYP PSALLNAGKS G SAQIYAIF ...文字列: MSATSGVVNR PLVLNHGSIE STILIPTTEY HFYQTLLEGF RKSLPQVTEG FADDDEPSSK AELLMKFLGY IVQSGVSNQQ EQLAAAKLV LNEFESRFLQ GLNLHSYAAI LLKSETFPTT LLKIKENLIK NYYLGRALVY LPGQRGLVYP PSALLNAGKS G SAQIYAIF GGQGNTDDYF EELRDIYHIY QGLVSDFVTK AQLKLQELIR TTPETDRIYT QGLDLINWLE NKDKTPDQQQ LL SIPMSCP LICVIQLCHY IVTCRILGIT PGQLRDSLKG TTGHSQGLVT AVVVSSADSW ESFEKLALQA VEFMFYIGVR GLQ TYPNTS LPPSIVQDSE ENAEGTPSPM LSVRDLSYDQ LVKFVNETNQ HLPEAKHIDI SLINGPRNVV LTGPPQSLYG LNLN LRKAK APSGLDQARI PFSERKLRFS NRFLPIMSPF HSHLLSPSTE KIVADLKKAG VEFSQSSMKL PVFDTYDGKD LRSYS GSIA ARLVECITKL RVNWELSTEF NSTHVLDFGP GGASGLGVLT HRNKEGTGSR VIVAGVLDAE SEDSEFGYKQ EIFESN EKA IKYAPNWLKE YKPKLVKTSA GKIFVDTKFS RLLGRAPLMV PGMTPTTVSP DFVAATLNAG FHTEIAGGGY FAPSIMK AA LQRVIDQVTP GTGVGINLIY VNPRMLQWGI PMIKELREQG FPIQSLSIGA GVPSLEVATE YIETLGLAHL GLKPGSID A VNQVITIAKA HPNFPIVLQW TGGRGGGHHS FEDFHQPILQ MYSKIRKCKN IILIAGSGFG SAEDTYPYLT GSWSHQFSY PSMPFDGVLF GSRVMTAKEA KTSPAAKQAI ADCTGVDNSQ WENTYKKPTG GIITVRSEMG EPIHKIATRG VMLWKELDDT IFTLPKNKM LEAIAKKKDY IIKKLNADYQ KPWFAKNEKG TCDLEDMTYK QIAERLVELM YVRKSQRWID VTLRNFTGKF L RRIEERFA TKVGTISLIQ NFSQLEEPEK AIDSVFKAYP EAASQLINEE DCDWFLLEAQ SPTQKPVPFI PVLDERFEFF FK KDSLWQS EDLEAVVGED VQRTCILHGP VAAQFSNKVD EPIKDILENI HKGHIKSLVK EVYNGDESKI PVVEYFSSVD SFS DTAIEG VKIERSRNTE TFTVTSGNVD NQQWFDLLAG KELSWRRAFI TAARLVQGTN FVSNPAHSVL APSKDLVVKI ENGS DAKKT VLTAFQRVRG KYVPAVSLKS IGDLKIKLEL IETRTADKSA VALELFYNYK PTDGFAPILE VMEGRNTSIK NFYWK LWFG SSVPVDLDFD ANKPISGGEA SVSSQAIAEF THAVGNSCED FVPRAGRPQL APMDFAIVLG WKAIMKAIFP KTVDGD ILK LVHLSNGYKM FPGADPLKKG DVVSTVAHIR SVVNGETGKT VEVVGVISRD GKPVLEVNSQ FFYRGKYQDF GNSFKKT TE TPVQVAFKSA KDIAVLKSKE WFHLEKDIDL LNQTLTFRCE SYVKFKSSTV FASVKTTGQA LLELPSKEII QVAEINYE S GSSYGNPVLD YLTRHGSTIE QPIMFENAIP LAQGTELTSK APGTNETYAA VSGDYNPIHV NKVFASYANL PGTITHGMY SSAAVRALVE QWAAQNVATR VRAFKADFVG MVLPNDELVT HLEHVGMING RKIIKVETKK VETEEVVLIG EAEIEQPVST FVFTGQGSQ EQGMGMDLYN SSEVAKSVWD RADVHFINNY GFSILDIVKN NPTELTVHFG GAKGRSIRNN YISMMFETVA A DGQLKSEK IFKEINEDTI SFTFKSPTGL LSATQFTQPA LTLMEKASFE DMKSKGLVPS ESMFAGHSLG EYSALTSLGD VM PIESLVD VVFYRGMTMQ VAVPRDEQGR SNYGMIAVNP SRVSSTFNDS ALRFVVEHIA QQTGWLLEIV NYNVENTQYV AAG DLRGLD TLSNVLNVFK IQKIDIVKLQ ETISLDEVKV HLSEIVDEVS KKSSSKPQPI DLERGFACIP LKGISVPFHS SYLR SGVKP FQTFLCKKIP KSAVKPANLI GKYIPNLTAK PFQLTKEYFE DVYELTKSEK IKHILDHWEE YESS UniProtKB: Fatty acid synthase subunit beta |
-分子 #3: FLAVIN MONONUCLEOTIDE
| 分子 | 名称: FLAVIN MONONUCLEOTIDE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: FMN |
|---|---|
| 分子量 | 理論値: 456.344 Da |
| Chemical component information |  ChemComp-FMN: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: PBS |
|---|---|
| グリッド | 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 281 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 実像数: 3643 / 平均電子線量: 60.1 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)