+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11740 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human RNA Polymerase III Elongation Complex, map F | |||||||||
 マップデータ マップデータ | Cryo-EM map of human RNA Polymerase III Elongation Complex, map F, unsharpened | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報snRNA transcription by RNA polymerase III / RNA Polymerase III Chain Elongation / RNA Polymerase III Transcription Termination / calcitonin gene-related peptide receptor activity / DNA/RNA hybrid binding / regulation of transcription by RNA polymerase I / regulation of transcription by RNA polymerase III / RPAP3/R2TP/prefoldin-like complex / DNA polymerase III complex / RNA Polymerase III Transcription Initiation From Type 1 Promoter ...snRNA transcription by RNA polymerase III / RNA Polymerase III Chain Elongation / RNA Polymerase III Transcription Termination / calcitonin gene-related peptide receptor activity / DNA/RNA hybrid binding / regulation of transcription by RNA polymerase I / regulation of transcription by RNA polymerase III / RPAP3/R2TP/prefoldin-like complex / DNA polymerase III complex / RNA Polymerase III Transcription Initiation From Type 1 Promoter / RNA Polymerase III Transcription Initiation From Type 2 Promoter / RNA Polymerase III Transcription Initiation From Type 3 Promoter / Cytosolic sensors of pathogen-associated DNA / RNA Polymerase III Abortive And Retractive Initiation / positive regulation of innate immune response / nucleobase-containing compound metabolic process / Abortive elongation of HIV-1 transcript in the absence of Tat / FGFR2 alternative splicing / RNA Polymerase I Transcription Termination / MicroRNA (miRNA) biogenesis / Viral Messenger RNA Synthesis / Signaling by FGFR2 IIIa TM / RNA Pol II CTD phosphorylation and interaction with CE during HIV infection / RNA Pol II CTD phosphorylation and interaction with CE / Formation of the Early Elongation Complex / Formation of the HIV-1 Early Elongation Complex / HIV Transcription Initiation / RNA Polymerase II HIV Promoter Escape / Transcription of the HIV genome / RNA Polymerase II Promoter Escape / RNA Polymerase II Transcription Pre-Initiation And Promoter Opening / RNA Polymerase II Transcription Initiation / RNA Polymerase II Transcription Initiation And Promoter Clearance / mRNA Capping / transcription initiation at RNA polymerase III promoter / mRNA Splicing - Minor Pathway / PIWI-interacting RNA (piRNA) biogenesis / RNA Polymerase I Transcription Initiation / Processing of Capped Intron-Containing Pre-mRNA / Pausing and recovery of Tat-mediated HIV elongation / Tat-mediated HIV elongation arrest and recovery / transcription by RNA polymerase III / RNA polymerase II transcribes snRNA genes / HIV elongation arrest and recovery / Pausing and recovery of HIV elongation / neuropeptide signaling pathway / Tat-mediated elongation of the HIV-1 transcript / Formation of HIV-1 elongation complex containing HIV-1 Tat / RNA polymerase I complex / transcription elongation by RNA polymerase I / RNA polymerase III complex / Formation of HIV elongation complex in the absence of HIV Tat / RNA polymerase II, core complex / tRNA transcription by RNA polymerase III / RNA Polymerase II Transcription Elongation / transcription by RNA polymerase I / Formation of RNA Pol II elongation complex / RNA Polymerase II Pre-transcription Events / acrosomal vesicle / Inhibition of DNA recombination at telomere / mRNA Splicing - Major Pathway / positive regulation of interferon-beta production / TP53 Regulates Transcription of DNA Repair Genes / RNA Polymerase I Promoter Escape / Transcription-Coupled Nucleotide Excision Repair (TC-NER) / Transcriptional regulation by small RNAs / Formation of TC-NER Pre-Incision Complex / protein-DNA complex / NoRC negatively regulates rRNA expression / B-WICH complex positively regulates rRNA expression / Dual incision in TC-NER / Gap-filling DNA repair synthesis and ligation in TC-NER / ribonucleoside binding / Activation of anterior HOX genes in hindbrain development during early embryogenesis / fibrillar center / DNA-directed RNA polymerase / DNA-directed RNA polymerase activity / single-stranded DNA binding / 4 iron, 4 sulfur cluster binding / double-stranded DNA binding / defense response to virus / Estrogen-dependent gene expression / nucleic acid binding / transcription by RNA polymerase II / cell population proliferation / protein dimerization activity / protein stabilization / nuclear body / innate immune response / nucleotide binding / intracellular membrane-bounded organelle / DNA-templated transcription / centrosome / chromatin binding / magnesium ion binding / mitochondrion / DNA binding / zinc ion binding / nucleoplasm / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
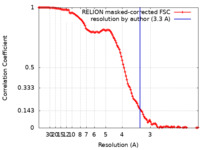
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Girbig M / Misiaszek AD / Vorlaender MK / Mueller CW | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2021 ジャーナル: Nat Struct Mol Biol / 年: 2021タイトル: Cryo-EM structures of human RNA polymerase III in its unbound and transcribing states. 著者: Mathias Girbig / Agata D Misiaszek / Matthias K Vorländer / Aleix Lafita / Helga Grötsch / Florence Baudin / Alex Bateman / Christoph W Müller /   要旨: RNA polymerase III (Pol III) synthesizes transfer RNAs and other short, essential RNAs. Human Pol III misregulation is linked to tumor transformation, neurodegenerative and developmental disorders, ...RNA polymerase III (Pol III) synthesizes transfer RNAs and other short, essential RNAs. Human Pol III misregulation is linked to tumor transformation, neurodegenerative and developmental disorders, and increased sensitivity to viral infections. Here, we present cryo-electron microscopy structures at 2.8 to 3.3 Å resolution of transcribing and unbound human Pol III. We observe insertion of the TFIIS-like subunit RPC10 into the polymerase funnel, providing insights into how RPC10 triggers transcription termination. Our structures resolve elements absent from Saccharomyces cerevisiae Pol III such as the winged-helix domains of RPC5 and an iron-sulfur cluster, which tethers the heterotrimer subcomplex to the core. The cancer-associated RPC7α isoform binds the polymerase clamp, potentially interfering with Pol III inhibition by tumor suppressor MAF1, which may explain why overexpressed RPC7α enhances tumor transformation. Finally, the human Pol III structure allows mapping of disease-related mutations and may contribute to the development of inhibitors that selectively target Pol III for therapeutic interventions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11740.map.gz emd_11740.map.gz | 113.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11740-v30.xml emd-11740-v30.xml emd-11740.xml emd-11740.xml | 20 KB 20 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11740_fsc.xml emd_11740_fsc.xml | 12 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11740.png emd_11740.png | 72.9 KB | ||
| マスクデータ |  emd_11740_msk_1.map emd_11740_msk_1.map | 144.7 MB |  マスクマップ マスクマップ | |
| その他 |  emd_11740_additional_1.map.gz emd_11740_additional_1.map.gz emd_11740_half_map_1.map.gz emd_11740_half_map_1.map.gz emd_11740_half_map_2.map.gz emd_11740_half_map_2.map.gz | 113.6 MB 114 MB 113.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11740 http://ftp.pdbj.org/pub/emdb/structures/EMD-11740 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11740 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11740 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11740_validation.pdf.gz emd_11740_validation.pdf.gz | 514.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11740_full_validation.pdf.gz emd_11740_full_validation.pdf.gz | 513.9 KB | 表示 | |
| XML形式データ |  emd_11740_validation.xml.gz emd_11740_validation.xml.gz | 17.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11740 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11740 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11740 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11740 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7a6hC  7ae1C  7ae3C  7aeaC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10697 (タイトル: Cryo-EM structures of human RNA Polymerase III EMPIAR-10697 (タイトル: Cryo-EM structures of human RNA Polymerase IIIData size: 3.3 TB Data #1: LZW-TIFF compressed multiframe micrographs of human RNA Pol III EC [micrographs - multiframe] Data #2: Initially picked particles of human RNA Pol III EC [picked particles - single frame - processed] Data #3: Polished particles of human RNA Pol III EC (final reconstruction) [picked particles - single frame - processed] Data #4: LZW-TIFF compressed multiframe micrographs of human RNA Pol III apo [micrographs - multiframe] Data #5: Initially picked particles of human RNA Pol III apo [picked particles - single frame - processed] Data #6: Polished particles of human RNA Pol III apo (final reconstruction) [picked particles - single frame - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11740.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11740.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of human RNA Polymerase III Elongation Complex, map F, unsharpened | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11740_msk_1.map emd_11740_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Cryo-EM map of human RNA Polymerase III Elongation...
| ファイル | emd_11740_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of human RNA Polymerase III Elongation Complex, map F, locally sharpened via LocalDeblur | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_11740_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_11740_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human RNA Polymerase III Elongation Complex
| 全体 | 名称: Human RNA Polymerase III Elongation Complex |
|---|---|
| 要素 |
|
-超分子 #1: Human RNA Polymerase III Elongation Complex
| 超分子 | 名称: Human RNA Polymerase III Elongation Complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#20 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 株: HEK293F Homo sapiens (ヒト) / 株: HEK293F |
| 分子量 | 理論値: 776 kDa/nm |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: Wait time 10 s Blot force 4 Blot time 4 s. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 40.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.75 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)