+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1174 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural analysis of the anaphase-promoting complex reveals multiple active sites and insights into polyubiquitylation. | |||||||||
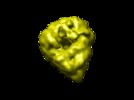
 マップデータ マップデータ | Structure of Saccharomyces cerevisiae Anaphase-Promoting Complex/Cyclosome (APC) | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | anaphase-promoting complex 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 20.0 Å | |||||||||
 データ登録者 データ登録者 | Passmore LA / Booth CR / Venien-Bryan C / Ludtke SJ / Fioretto C / Johnson LN / Chiu W / Barford D | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2005 ジャーナル: Mol Cell / 年: 2005タイトル: Structural analysis of the anaphase-promoting complex reveals multiple active sites and insights into polyubiquitylation. 著者: Lori A Passmore / Christopher R Booth / Catherine Vénien-Bryan / Steven J Ludtke / Céline Fioretto / Louise N Johnson / Wah Chiu / David Barford /  要旨: The anaphase-promoting complex/cyclosome (APC/C) is an E3 ubiquitin ligase composed of approximately 13 distinct subunits required for progression through meiosis, mitosis, and the G1 phase of the ...The anaphase-promoting complex/cyclosome (APC/C) is an E3 ubiquitin ligase composed of approximately 13 distinct subunits required for progression through meiosis, mitosis, and the G1 phase of the cell cycle. Despite its central role in these processes, information concerning its composition and structure is limited. Here, we determined the structure of yeast APC/C by cryo-electron microscopy (cryo-EM). Docking of tetratricopeptide repeat (TPR)-containing subunits indicates that they likely form a scaffold-like outer shell, mediating assembly of the complex and providing potential binding sites for regulators and substrates. Quantitative determination of subunit stoichiometry indicates multiple copies of specific subunits, consistent with a total APC/C mass of approximately 1.7 MDa. Moreover, yeast APC/C forms both monomeric and dimeric species. Dimeric APC/C is a more active E3 ligase than the monomer, with greatly enhanced processivity. Our data suggest that multimerisation and/or the presence of multiple active sites facilitates the APC/C's ability to elongate polyubiquitin chains. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1174.map.gz emd_1174.map.gz | 10.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1174-v30.xml emd-1174-v30.xml emd-1174.xml emd-1174.xml | 10.4 KB 10.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1174.gif 1174.gif | 7.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1174 http://ftp.pdbj.org/pub/emdb/structures/EMD-1174 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1174 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1174 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1174_validation.pdf.gz emd_1174_validation.pdf.gz | 202.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1174_full_validation.pdf.gz emd_1174_full_validation.pdf.gz | 201.6 KB | 表示 | |
| XML形式データ |  emd_1174_validation.xml.gz emd_1174_validation.xml.gz | 5.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1174 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1174 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1174 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1174 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1174.map.gz / 形式: CCP4 / 大きさ: 11.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1174.map.gz / 形式: CCP4 / 大きさ: 11.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of Saccharomyces cerevisiae Anaphase-Promoting Complex/Cyclosome (APC) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.168 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Anaphase-Promoting Complex
| 全体 | 名称: Anaphase-Promoting Complex |
|---|---|
| 要素 |
|
-超分子 #1000: Anaphase-Promoting Complex
| 超分子 | 名称: Anaphase-Promoting Complex / タイプ: sample / ID: 1000 詳細: The APC was freshly purified before grid preparation. 集合状態: monomer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 1.7 MDa / 理論値: 1.7 MDa |
-分子 #1: Anaphase-Promoting Complex, Cyclosome
| 分子 | 名称: Anaphase-Promoting Complex, Cyclosome / タイプ: protein_or_peptide / ID: 1 / Name.synonym: APC 詳細: Endogenous APC was purified using a TAP tag present at the C-terminus of the Cdc16 subunit. The APC has 13 different protein subunits. There are multiple copies of specific APC subunits, ...詳細: Endogenous APC was purified using a TAP tag present at the C-terminus of the Cdc16 subunit. The APC has 13 different protein subunits. There are multiple copies of specific APC subunits, consistent with a total mass of 1.7 MDa. 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 1.7 MDa / 理論値: 1.7 MDa |
| 組換発現 | 生物種:  |
| 配列 | GO: anaphase-promoting complex |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.125 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 20 mM HEPES, 150 mM NaCl, 2 mM EGTA, 3 mM DTT, 1 mM Magnesium acetate, 0.015% (w/v) n-Dodecyl B-D-maltoside (DDM) |
| グリッド | 詳細: Quantifoil R2/2, 200 mesh with an additional layer of freshly floated carbon |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / 装置: OTHER / 詳細: Vitrification instrument: Vitrobot 手法: 4 ul sample was applied to the grid then blotted two times one second on both sides before plunging into liquid ethane. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2010F |
|---|---|
| 温度 | 平均: 92 K |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サンプリング間隔: 15 µm / 実像数: 650 / 平均電子線量: 15 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 69200 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 1 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Side entry liquid-nitrogen cooled cryo specimen holder 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle was phase flipped. Amplitude corrections were applied during class averaging. |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 20.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: eman / 使用した粒子像数: 19384 |
-原子モデル構築 1
| ソフトウェア | 名称: Foldhunter, DockEM |
|---|---|
| 詳細 | 15 or 18 consecutive TPR motifs modelled from PP5 (1A17) were docked as rigid structures into the APC density map using Foldhunter or DockEM. Both programs produced similar results. |
| 精密化 | プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Cross-correlation |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















