+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11233 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
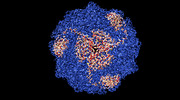
| タイトル | Helicobacter pylori urease with inhibitor bound in the active site | |||||||||
 マップデータ マップデータ | Relion unmasked map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | dodecamer / bi nickel center / enzyme / cytoplasm / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報urease complex / urease / urease activity / urea catabolic process / nickel cation binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
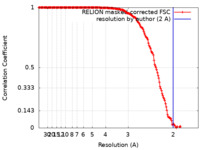
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.0 Å | |||||||||
 データ登録者 データ登録者 | Luecke H / Cunha E | |||||||||
| 資金援助 |  ノルウェー, ノルウェー,  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Cryo-EM structure of Helicobacter pylori urease with an inhibitor in the active site at 2.0 Å resolution. 著者: Eva S Cunha / Xiaorui Chen / Marta Sanz-Gaitero / Deryck J Mills / Hartmut Luecke /     要旨: Infection of the human stomach by Helicobacter pylori remains a worldwide problem and greatly contributes to peptic ulcer disease and gastric cancer. Without active intervention approximately 50% of ...Infection of the human stomach by Helicobacter pylori remains a worldwide problem and greatly contributes to peptic ulcer disease and gastric cancer. Without active intervention approximately 50% of the world population will continue to be infected with this gastric pathogen. Current eradication, called triple therapy, entails a proton-pump inhibitor and two broadband antibiotics, however resistance to either clarithromycin or metronidazole is greater than 25% and rising. Therefore, there is an urgent need for a targeted, high-specificity eradication drug. Gastric infection by H. pylori depends on the expression of a nickel-dependent urease in the cytoplasm of the bacteria. Here, we report the 2.0 Å resolution structure of the 1.1 MDa urease in complex with an inhibitor by cryo-electron microscopy and compare it to a β-mercaptoethanol-inhibited structure at 2.5 Å resolution. The structural information is of sufficient detail to aid in the development of inhibitors with high specificity and affinity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11233.map.gz emd_11233.map.gz | 370.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11233-v30.xml emd-11233-v30.xml emd-11233.xml emd-11233.xml | 26.5 KB 26.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11233_fsc.xml emd_11233_fsc.xml | 16.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11233.png emd_11233.png | 274 KB | ||
| マスクデータ |  emd_11233_msk_1.map emd_11233_msk_1.map | 396.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-11233.cif.gz emd-11233.cif.gz | 8 KB | ||
| その他 |  emd_11233_additional_1.map.gz emd_11233_additional_1.map.gz emd_11233_half_map_1.map.gz emd_11233_half_map_1.map.gz emd_11233_half_map_2.map.gz emd_11233_half_map_2.map.gz | 70.9 MB 314.8 MB 314.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11233 http://ftp.pdbj.org/pub/emdb/structures/EMD-11233 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11233 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11233 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11233_validation.pdf.gz emd_11233_validation.pdf.gz | 952.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11233_full_validation.pdf.gz emd_11233_full_validation.pdf.gz | 952.3 KB | 表示 | |
| XML形式データ |  emd_11233_validation.xml.gz emd_11233_validation.xml.gz | 24 KB | 表示 | |
| CIF形式データ |  emd_11233_validation.cif.gz emd_11233_validation.cif.gz | 31.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11233 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11233 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11233 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11233 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11233.map.gz / 形式: CCP4 / 大きさ: 396.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11233.map.gz / 形式: CCP4 / 大きさ: 396.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion unmasked map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8426 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11233_msk_1.map emd_11233_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Density modified map
| ファイル | emd_11233_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Density modified map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_11233_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_11233_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 1.1 MDa Helicobacter pylori Urease
| 全体 | 名称: 1.1 MDa Helicobacter pylori Urease |
|---|---|
| 要素 |
|
-超分子 #1: 1.1 MDa Helicobacter pylori Urease
| 超分子 | 名称: 1.1 MDa Helicobacter pylori Urease / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 1.1 MDa |
-分子 #1: Urease subunit alpha
| 分子 | 名称: Urease subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO / EC番号: urease |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 26.645703 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKLTPKELDK LMLHYAGELA RKRKEKGIKL NYVEAVALIS AHIMEEARAG KKTAAELMQE GRTLLKPDDV MDGVASMIHE VGIEAMFPD GTKLVTVHTP IEANGKLVPG ELFLKNEDIT INEGKKAVSV KVKNVGDRPV QIGSHFHFFE VNRCLDFDRE K TFGKRLDI ...文字列: MKLTPKELDK LMLHYAGELA RKRKEKGIKL NYVEAVALIS AHIMEEARAG KKTAAELMQE GRTLLKPDDV MDGVASMIHE VGIEAMFPD GTKLVTVHTP IEANGKLVPG ELFLKNEDIT INEGKKAVSV KVKNVGDRPV QIGSHFHFFE VNRCLDFDRE K TFGKRLDI ASGTAVRFEP GEEKSVELID IGGNRRIFGF NALVDRQADN ESKKIALHRA KERGFHGTKS DDNYVKTIKE UniProtKB: Urease subunit alpha |
-分子 #2: Urease subunit beta
| 分子 | 名称: Urease subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 12 / 光学異性体: LEVO / EC番号: urease |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 61.832531 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKKISRKEYV SMYGPTTGDK VRLGDTDLIA EVEHDYTIYG EELKFGGGKT LREGMSQSNN PSKEELDLII TNALIVDYTG IYKADIGIK DGKIAGIGKG GNKDMQDGVK NNLSVGPATE ALAGEGLIVT AGGIDTHIHF ISPQQIPTAF ASGVTTMIGG G TGPADGTN ...文字列: MKKISRKEYV SMYGPTTGDK VRLGDTDLIA EVEHDYTIYG EELKFGGGKT LREGMSQSNN PSKEELDLII TNALIVDYTG IYKADIGIK DGKIAGIGKG GNKDMQDGVK NNLSVGPATE ALAGEGLIVT AGGIDTHIHF ISPQQIPTAF ASGVTTMIGG G TGPADGTN ATTITPGRRN LKWMLRAAEE YSMNLGFLAK GNTSNDASLA DQIEAGAIGF (KCX)IHEDWGTTP SAINHALD V ADKYDVQVAI HTDTLNEAGC VEDTMAAIAG RTMHTFHTEG AGGGHAPDII KVAGEHNILP ASTNPTIPFT VNTEAEHMD MLMVCHHLDK SIKEDVQFAD SRIRPQTIAA EDTLHDMGIF SITSSDSQAM GRVGEVITRT WQTADKNKKE FGRLKEEKGD NDNFRIKRY LSKYTINPAI AHGISEYVGS VEVGKVADLV LWSPAFFGVK PNMIIKGGFI ALSQMGDANA SIPTPQPVYY R EMFAHHGK AKYDANITFV SQAAYDKGIK EELGLERQVL PVKNCRNITK KDMQFNDTTA HIEVNPETYH VFVDGKEVTS KP ANKVSLA QLFSIF UniProtKB: Urease subunit beta |
-分子 #3: NICKEL (II) ION
| 分子 | 名称: NICKEL (II) ION / タイプ: ligand / ID: 3 / コピー数: 24 / 式: NI |
|---|---|
| 分子量 | 理論値: 58.693 Da |
| Chemical component information |  ChemComp-NI: |
-分子 #4: 2-{[1-(3,5-dimethylphenyl)-1H-imidazol-2-yl]sulfanyl}-N-hydroxyac...
| 分子 | 名称: 2-{[1-(3,5-dimethylphenyl)-1H-imidazol-2-yl]sulfanyl}-N-hydroxyacetamide タイプ: ligand / ID: 4 / コピー数: 12 / 式: DJM |
|---|---|
| 分子量 | 理論値: 277.342 Da |
| Chemical component information | 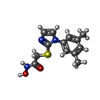 ChemComp-DJM: |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 3191 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 / 構成要素:
| ||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 70 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 1 / 実像数: 956 / 平均電子線量: 50.0 e/Å2 / 詳細: Used 718 movies |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)