+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11203 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Uncleavable Spike Protein of SARS-CoV-2 in Closed Conformation | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SARS-CoV-2 / Spike / Virus Glycoprotein / Coronavirus / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated disruption of host tissue / Maturation of spike protein / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / viral translation / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion ...symbiont-mediated disruption of host tissue / Maturation of spike protein / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / viral translation / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / membrane fusion / Attachment and Entry / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / host cell surface receptor binding / symbiont-mediated suppression of host innate immune response / receptor ligand activity / endocytosis involved in viral entry into host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / symbiont entry into host cell / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
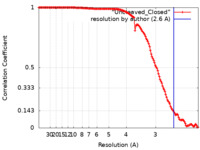
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||
 データ登録者 データ登録者 | Wrobel AG / Benton DJ | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2020 ジャーナル: Nat Struct Mol Biol / 年: 2020タイトル: SARS-CoV-2 and bat RaTG13 spike glycoprotein structures inform on virus evolution and furin-cleavage effects. 著者: Antoni G Wrobel / Donald J Benton / Pengqi Xu / Chloë Roustan / Stephen R Martin / Peter B Rosenthal / John J Skehel / Steven J Gamblin /   要旨: SARS-CoV-2 is thought to have emerged from bats, possibly via a secondary host. Here, we investigate the relationship of spike (S) glycoprotein from SARS-CoV-2 with the S protein of a closely related ...SARS-CoV-2 is thought to have emerged from bats, possibly via a secondary host. Here, we investigate the relationship of spike (S) glycoprotein from SARS-CoV-2 with the S protein of a closely related bat virus, RaTG13. We determined cryo-EM structures for RaTG13 S and for both furin-cleaved and uncleaved SARS-CoV-2 S; we compared these with recently reported structures for uncleaved SARS-CoV-2 S. We also biochemically characterized their relative stabilities and affinities for the SARS-CoV-2 receptor ACE2. Although the overall structures of human and bat virus S proteins are similar, there are key differences in their properties, including a more stable precleavage form of human S and about 1,000-fold tighter binding of SARS-CoV-2 to human receptor. These observations suggest that cleavage at the furin-cleavage site decreases the overall stability of SARS-CoV-2 S and facilitates the adoption of the open conformation that is required for S to bind to the ACE2 receptor. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11203.map.gz emd_11203.map.gz | 5.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11203-v30.xml emd-11203-v30.xml emd-11203.xml emd-11203.xml | 17.7 KB 17.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11203_fsc.xml emd_11203_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11203.png emd_11203.png | 160.4 KB | ||
| マスクデータ |  emd_11203_msk_1.map emd_11203_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-11203.cif.gz emd-11203.cif.gz | 6.6 KB | ||
| その他 |  emd_11203_half_map_1.map.gz emd_11203_half_map_1.map.gz emd_11203_half_map_2.map.gz emd_11203_half_map_2.map.gz | 226.6 MB 226.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11203 http://ftp.pdbj.org/pub/emdb/structures/EMD-11203 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11203 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11203 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11203_validation.pdf.gz emd_11203_validation.pdf.gz | 741.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11203_full_validation.pdf.gz emd_11203_full_validation.pdf.gz | 741.3 KB | 表示 | |
| XML形式データ |  emd_11203_validation.xml.gz emd_11203_validation.xml.gz | 22.4 KB | 表示 | |
| CIF形式データ |  emd_11203_validation.cif.gz emd_11203_validation.cif.gz | 28.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11203 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11203 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11203 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11203 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6zgeMC  6zgfC  6zggC  6zghC  6zgiC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10469 (タイトル: Single particle cryo EM dataset of SARS-CoV-2 Spike protein in uncleavable form, which generated a 2.6Å reconstruction of the closed trimer. EMPIAR-10469 (タイトル: Single particle cryo EM dataset of SARS-CoV-2 Spike protein in uncleavable form, which generated a 2.6Å reconstruction of the closed trimer.Data size: 8.4 TB Data #1: Unaligned Movies of uncleavable SARS-CoV-2 Spike protein ectodomain [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11203.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11203.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.087 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
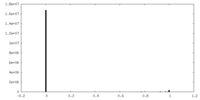
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11203_msk_1.map emd_11203_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
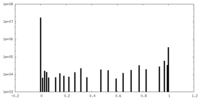
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_11203_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
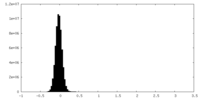
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_11203_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
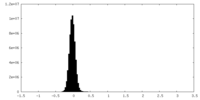
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 Spike Glycoprotein Ectodomain
| 全体 | 名称: SARS-CoV-2 Spike Glycoprotein Ectodomain |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 Spike Glycoprotein Ectodomain
| 超分子 | 名称: SARS-CoV-2 Spike Glycoprotein Ectodomain / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 500 KDa |
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 142.269859 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGILPSPGMP ALLSLVSLLS VLLMGCVAET GMFVFLVLLP LVSSQCVNLT TRTQLPPAYT NSFTRGVYYP DKVFRSSVLH STQDLFLPF FSNVTWFHAI HVSGTNGTKR FDNPVLPFND GVYFASTEKS NIIRGWIFGT TLDSKTQSLL IVNNATNVVI K VCEFQFCN ...文字列: MGILPSPGMP ALLSLVSLLS VLLMGCVAET GMFVFLVLLP LVSSQCVNLT TRTQLPPAYT NSFTRGVYYP DKVFRSSVLH STQDLFLPF FSNVTWFHAI HVSGTNGTKR FDNPVLPFND GVYFASTEKS NIIRGWIFGT TLDSKTQSLL IVNNATNVVI K VCEFQFCN DPFLGVYYHK NNKSWMESEF RVYSSANNCT FEYVSQPFLM DLEGKQGNFK NLREFVFKNI DGYFKIYSKH TP INLVRDL PQGFSALEPL VDLPIGINIT RFQTLLALHR SYLTPGDSSS GWTAGAAAYY VGYLQPRTFL LKYNENGTIT DAV DCALDP LSETKCTLKS FTVEKGIYQT SNFRVQPTES IVRFPNITNL CPFGEVFNAT RFASVYAWNR KRISNCVADY SVLY NSASF STFKCYGVSP TKLNDLCFTN VYADSFVIRG DEVRQIAPGQ TGKIADYNYK LPDDFTGCVI AWNSNNLDSK VGGNY NYLY RLFRKSNLKP FERDISTEIY QAGSTPCNGV EGFNCYFPLQ SYGFQPTNGV GYQPYRVVVL SFELLHAPAT VCGPKK STN LVKNKCVNFN FNGLTGTGVL TESNKKFLPF QQFGRDIADT TDAVRDPQTL EILDITPCSF GGVSVITPGT NTSNQVA VL YQDVNCTEVP VAIHADQLTP TWRVYSTGSN VFQTRAGCLI GAEHVNNSYE CDIPIGAGIC ASYQTQTNSP SRASSVAS Q SIIAYTMSLG AENSVAYSNN SIAIPTNFTI SVTTEILPVS MTKTSVDCTM YICGDSTECS NLLLQYGSFC TQLNRALTG IAVEQDKNTQ EVFAQVKQIY KTPPIKDFGG FNFSQILPDP SKPSKRSFIE DLLFNKVTLA DAGFIKQYGD CLGDIAARDL ICAQKFNGL TVLPPLLTDE MIAQYTSALL AGTITSGWTF GAGAALQIPF AMQMAYRFNG IGVTQNVLYE NQKLIANQFN S AIGKIQDS LSSTASALGK LQDVVNQNAQ ALNTLVKQLS SNFGAISSVL NDILSRLDPP EAEVQIDRLI TGRLQSLQTY VT QQLIRAA EIRASANLAA TKMSECVLGQ SKRVDFCGKG YHLMSFPQSA PHGVVFLHVT YVPAQEKNFT TAPAICHDGK AHF PREGVF VSNGTHWFVT QRNFYEPQII TTDNTFVSGN CDVVIGIVNN TVYDPLQPEL DSFKEELDKY FKNHTSPDVD LGDI SGINA SVVNIQKEID RLNEVAKNLN ESLIDLQELG KYEQSGRENL YFQGGGGSGY IPEAPRDGQA YVRKDGEWVL LSTFL GHHH HHH UniProtKB: Spike glycoprotein |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 21 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 48 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均露光時間: 60.0 sec. / 平均電子線量: 33.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)