+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1110 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural basis for the function of the ribosomal L7/12 stalk in factor binding and GTPase activation. | |||||||||
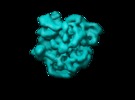
 マップデータ マップデータ | This is a map of the E.coli Ribosome in complex with elongation factor G in the presence of the antibiotic fusidic acid. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 18.0 Å | |||||||||
 データ登録者 データ登録者 | Diaconu M / Kothe U / Schlunzen F / Harms JM / Fischer N / Stark H / Rodnina MV / Wahl MC | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2005 ジャーナル: Cell / 年: 2005タイトル: Structural basis for the function of the ribosomal L7/12 stalk in factor binding and GTPase activation. 著者: Mihaela Diaconu / Ute Kothe / Frank Schlünzen / Niels Fischer / Jörg M Harms / Alexander G Tonevitsky / Holger Stark / Marina V Rodnina / Markus C Wahl /  要旨: The L7/12 stalk of the large subunit of bacterial ribosomes encompasses protein L10 and multiple copies of L7/12. We present crystal structures of Thermotoga maritima L10 in complex with three L7/12 ...The L7/12 stalk of the large subunit of bacterial ribosomes encompasses protein L10 and multiple copies of L7/12. We present crystal structures of Thermotoga maritima L10 in complex with three L7/12 N-terminal-domain dimers, refine the structure of an archaeal L10E N-terminal domain on the 50S subunit, and identify these elements in cryo-electron-microscopic reconstructions of Escherichia coli ribosomes. The mobile C-terminal helix alpha8 of L10 carries three L7/12 dimers in T. maritima and two in E. coli, in concordance with the different length of helix alpha8 of L10 in these organisms. The stalk is organized into three elements (stalk base, L10 helix alpha8-L7/12 N-terminal-domain complex, and L7/12 C-terminal domains) linked by flexible connections. Highly mobile L7/12 C-terminal domains promote recruitment of translation factors to the ribosome and stimulate GTP hydrolysis by the ribosome bound factors through stabilization of their active GTPase conformation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1110.map.gz emd_1110.map.gz | 4.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1110-v30.xml emd-1110-v30.xml emd-1110.xml emd-1110.xml | 9.6 KB 9.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1110.gif 1110.gif | 46.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1110 http://ftp.pdbj.org/pub/emdb/structures/EMD-1110 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1110 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1110 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1110_validation.pdf.gz emd_1110_validation.pdf.gz | 244.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1110_full_validation.pdf.gz emd_1110_full_validation.pdf.gz | 243.4 KB | 表示 | |
| XML形式データ |  emd_1110_validation.xml.gz emd_1110_validation.xml.gz | 5.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1110 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1110 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1110 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1110 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1110.map.gz / 形式: CCP4 / 大きさ: 4.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1110.map.gz / 形式: CCP4 / 大きさ: 4.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a map of the E.coli Ribosome in complex with elongation factor G in the presence of the antibiotic fusidic acid. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : E coli ribosome in complex with elongation factor G
| 全体 | 名称: E coli ribosome in complex with elongation factor G |
|---|---|
| 要素 |
|
-超分子 #1000: E coli ribosome in complex with elongation factor G
| 超分子 | 名称: E coli ribosome in complex with elongation factor G / タイプ: sample / ID: 1000 / Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 2.5 MDa |
-超分子 #1: 70S
| 超分子 | 名称: 70S / タイプ: complex / ID: 1 / 組換発現: No / Ribosome-details: ribosome-prokaryote: ALL |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 2.5 MDa |
-分子 #1: Elongation Factor G
| 分子 | 名称: Elongation Factor G / タイプ: protein_or_peptide / ID: 1 / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 50 mM Tris HCl, 70 mM NH4Cl, 30 mM KCl, 7 mM MgCl2, 1 mM DTT |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 40 % / チャンバー内温度: 93 K / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: home made / 手法: Blotted manually for two seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200FEG |
|---|---|
| 温度 | 平均: 93 K |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: PRIMESCAN / デジタル化 - サンプリング間隔: 4.0 µm / 実像数: 12 / 平均電子線量: 15 e/Å2 / Od range: 1.3 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 詳細 | The particles were selected manually. |
|---|---|
| CTF補正 | 詳細: Each particle |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 18.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: IMAGIC / 使用した粒子像数: 8000 |
-原子モデル構築 1
| ソフトウェア | 名称: Amira 3.1, TGS Software |
|---|---|
| 詳細 | Protocol: Rigid Body. Manual fitting |
| 精密化 | プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















