+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10795 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain map of S. cerevisiae Brf1 and TBP bound to the TFIIIC-subcomplex tauA | |||||||||
 マップデータ マップデータ | Refined map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 32.36 Å | |||||||||
 データ登録者 データ登録者 | Vorlaender MK / Muller CW | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Structure of the TFIIIC subcomplex τA provides insights into RNA polymerase III pre-initiation complex formation. 著者: Matthias K Vorländer / Anna Jungblut / Kai Karius / Florence Baudin / Helga Grötsch / Jan Kosinski / Christoph W Müller /  要旨: Transcription factor (TF) IIIC is a conserved eukaryotic six-subunit protein complex with dual function. It serves as a general TF for most RNA polymerase (Pol) III genes by recruiting TFIIIB, but it ...Transcription factor (TF) IIIC is a conserved eukaryotic six-subunit protein complex with dual function. It serves as a general TF for most RNA polymerase (Pol) III genes by recruiting TFIIIB, but it is also involved in chromatin organization and regulation of Pol II genes through interaction with CTCF and condensin II. Here, we report the structure of the S. cerevisiae TFIIIC subcomplex τA, which contains the most conserved subunits of TFIIIC and is responsible for recruitment of TFIIIB and transcription start site (TSS) selection at Pol III genes. We show that τA binding to its promoter is auto-inhibited by a disordered acidic tail of subunit τ95. We further provide a negative-stain reconstruction of τA bound to the TFIIIB subunits Brf1 and TBP. This shows that a ruler element in τA achieves positioning of TFIIIB upstream of the TSS, and suggests remodeling of the complex during assembly of TFIIIB by TFIIIC. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10795.map.gz emd_10795.map.gz | 23.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10795-v30.xml emd-10795-v30.xml emd-10795.xml emd-10795.xml | 21.8 KB 21.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10795_fsc.xml emd_10795_fsc.xml | 9.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10795.png emd_10795.png | 78.7 KB | ||
| その他 |  emd_10795_half_map_1.map.gz emd_10795_half_map_1.map.gz emd_10795_half_map_2.map.gz emd_10795_half_map_2.map.gz | 23.4 MB 23.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10795 http://ftp.pdbj.org/pub/emdb/structures/EMD-10795 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10795 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10795 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10795_validation.pdf.gz emd_10795_validation.pdf.gz | 295.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10795_full_validation.pdf.gz emd_10795_full_validation.pdf.gz | 294.8 KB | 表示 | |
| XML形式データ |  emd_10795_validation.xml.gz emd_10795_validation.xml.gz | 13 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10795 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10795 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10795 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10795 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10795.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10795.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refined map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.292 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
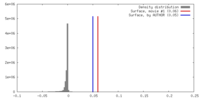
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half map 2
| ファイル | emd_10795_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
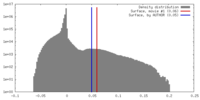
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_10795_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
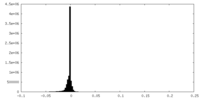
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex between Brf1, TBP, and the tauA subcomplex of TFIIIC
| 全体 | 名称: Complex between Brf1, TBP, and the tauA subcomplex of TFIIIC |
|---|---|
| 要素 |
|
-超分子 #1: Complex between Brf1, TBP, and the tauA subcomplex of TFIIIC
| 超分子 | 名称: Complex between Brf1, TBP, and the tauA subcomplex of TFIIIC タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Crosslinked with Glutaraldehyde in absence of DNA |
|---|---|
| 分子量 | 理論値: 300 KDa |
-超分子 #2: Transcription factor tau 131 kDa subunit, Transcription factor ta...
| 超分子 | 名称: Transcription factor tau 131 kDa subunit, Transcription factor tau 95 kDa subunit and Transcription factor tau 55 kDa subunit タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
-超分子 #3: Transcription factor IIIB 70 kDa subunit and TATA-box-binding protein
| 超分子 | 名称: Transcription factor IIIB 70 kDa subunit and TATA-box-binding protein タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #4-#5 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-分子 #1: Transcription factor tau 131 kDa subunit
| 分子 | 名称: Transcription factor tau 131 kDa subunit / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAAGKLKKEQ QNQSAERESA DTGKVNDEDE EHLYGNIDDY KHLIQDEEYD DEDVPHDLQL SEDEYNSER DSSLLAEFSD YGEISEDDEE DFMNAIREAS NFKVKKKKKN DKGKSYGRQR K ERVLDPEV AQLLSQANEA FVRNDLQVAE RLFNEVIKKD ARNFAAYETL ...文字列: MAAGKLKKEQ QNQSAERESA DTGKVNDEDE EHLYGNIDDY KHLIQDEEYD DEDVPHDLQL SEDEYNSER DSSLLAEFSD YGEISEDDEE DFMNAIREAS NFKVKKKKKN DKGKSYGRQR K ERVLDPEV AQLLSQANEA FVRNDLQVAE RLFNEVIKKD ARNFAAYETL GDIYQLQGRL ND CCNSWFL AAHLNASDWE FWKIVAILSA DLDHVRQAIY CFSRVISLNP MEWESIYRRS MLY KKTGQL ARALDGFQRL YMYNPYDANI LRELAILYVD YDRIEDSIEL YMKVFNANVE RREA ILAAL ENALDSSDEE SAAEGEDADE KEPLEQDEDR QMFPDINWKK IDAKYKCIPF DWSSL NILA ELFLKLAVSE VDGIKTIKKC ARWIQRRESQ TFWDHVPDDS EFDNRRFKNS TFDSLL AAE KEKSYNIPID IRVRLGLLRL NTDNLVEALN HFQCLYDETF SDVADLYFEA ATALTRA EK YKEAIDFFTP LLSLEEWRTT DVFKPLARCY KEIESYETAK EFYELAIKSE PDDLDIRV S LAEVYYRLND PETFKHMLVD VVEMRKHQVD ETLHRISNEK SSNDTSDISS KPLLEDSKF RTFRKKKRTP YDAERERIER ERRITAKVVD KYEKMKKFEL NSGLNEAKQA SIWINTVSEL VDIFSSVKN FFMKSRSRKF VGILRRTKKF NTELDFQIER LSKLAEGDSV FEGPLMEERV T LTSATELR GLSYEQWFEL FMELSLVIAK YQSVEDGLSV VETAQEVNVF FQDPERVKMM KF VKLAIVL QMDDEEELAE NLRGLLNQFQ FNRKVLQVFM YSLCRGPSSL NILSSTIQQK FFL RQLKAF DSCRYNTEVN GQASITNKEV YNPNKKSSPY LYYIYAVLLY SSRGFLSALQ YLTR LEEDI PDDPMVNLLM GLSHIHRAMQ RLTAQRHFQI FHGLRYLYRY HKIRKSLYTD LEKQE ADYN LGRAFHLIGL VSIAIEYYNR VLENYDDGKL KKHAAYNSII IYQQSGNVEL ADHLME KYL SIRSGG |
-分子 #2: Transcription factor tau 95 kDa subunit
| 分子 | 名称: Transcription factor tau 95 kDa subunit / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MHHHHHHENL YFQSMPVEEP LATLSSIPDS SADQAPPLIA DEFTLDLPRI PSLELPLNVS TKHSSIQKA IKMCGGIEKV KEAFKEHGPI ESQHGLQLYL NDDTDSDGSK SYFNEHPVIG K RVPFRDES VILKVTMPKG TLSKNNNSVK DSIKSLKDSN KLRVTPVSIV ...文字列: MHHHHHHENL YFQSMPVEEP LATLSSIPDS SADQAPPLIA DEFTLDLPRI PSLELPLNVS TKHSSIQKA IKMCGGIEKV KEAFKEHGPI ESQHGLQLYL NDDTDSDGSK SYFNEHPVIG K RVPFRDES VILKVTMPKG TLSKNNNSVK DSIKSLKDSN KLRVTPVSIV DNTIKFREMS DF QIKLDNV PSAREFKSSF GSLEWNNFKS FVNSVPDNDS QPQENIGNLI LDRSVKIPST DFQ LPPPPK LSMVGFPLLY KYKANPFAKK KKNGVTEVKG TYIKNYQLFV HDLSDKTVIP SQAH EQVLY DFEVAKKTKV YPGTKSDSKF YESLEECLKI LRELFARRPI WVKRHLDGIV PKKIH HTMK IALALISYRF TMGPWRNTYI KFGIDPRSSV EYAQYQTEYF KIERKLLSSP IVKKNV PKP PPLVFESDTP GGIDSRFKFD GKRIPWYLML QIDLLIGEPN IAEVFHNVEY LDKANEL TG WFKELDLVKI RRIVKYELGC MVQGNYEYNK YKLKYFKTML FVKESMVPEN KNSEEGMG V NTNKDADGDI NMDAGSQMSS NAIEEDKGIA AGDDFDDNGA ITEEPDDAAL ENEEMDTDQ NLKVPASIDD DVDDVDADEE EQESFDVKTA SFQDIINKIA KLDPKTAETM KSELKGFVDE VDL |
-分子 #3: Transcription factor tau 55 kDa subunit
| 分子 | 名称: Transcription factor tau 55 kDa subunit / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MVVNTIYIAR HGYRSNWLPE GPYPDPLTGI DSDVPLAEHG VQQAKELAHY LLSLDNQPEA AFASPFYRC LETVQPIAKL LEIPVYLERG IGEWYRPDRK PVIPVPAGYE ILSKFFPGVI S QEWDSTLT PNEKGETEQE MYMRFKKFWP LFIERVEKEY PNVECILLVT ...文字列: MVVNTIYIAR HGYRSNWLPE GPYPDPLTGI DSDVPLAEHG VQQAKELAHY LLSLDNQPEA AFASPFYRC LETVQPIAKL LEIPVYLERG IGEWYRPDRK PVIPVPAGYE ILSKFFPGVI S QEWDSTLT PNEKGETEQE MYMRFKKFWP LFIERVEKEY PNVECILLVT HAASKIALGM SL LGYDNPR MSLNENGDKI RSGSCSLDKY EILKKSYDTI DETDDQTSFT YIPFSDRKWV LTM NGNTEF LSSGEEMNWN FDCVAEAGSD ADIKKRQMTK KTSSPIPEAD DQTEVETVYI SVDI PSGNY KERTEIAKSA ILQYSGLETD APLFRIGNRL YEGSWERLVG TELAFPNAAH VHKKT AGLL SPTEENETTN AGQSKGSSTA NDPNIQIQEE DVGLPDSTNT SRDHTGDKEE VQSEKI YRI KERIVLSNVR PM |
-分子 #4: Transcription factor IIIB 70 kDa subunit
| 分子 | 名称: Transcription factor IIIB 70 kDa subunit / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSALESREAT LNNARRKLRA VSYALHIPEY ITDAAFQWYK LALANNFVQG R RSQNVIAS CLYVACRKEK THHMLIDFSS RLQVSVYSIG ATFLKMVKKL HITELPLADP SL FIQHFAE KLDLADKKIK VVKDAVKLAQ RMSKDWMFEG RRPAGIAGAC ILLACRMNNL ...文字列: MSALESREAT LNNARRKLRA VSYALHIPEY ITDAAFQWYK LALANNFVQG R RSQNVIAS CLYVACRKEK THHMLIDFSS RLQVSVYSIG ATFLKMVKKL HITELPLADP SL FIQHFAE KLDLADKKIK VVKDAVKLAQ RMSKDWMFEG RRPAGIAGAC ILLACRMNNL RRT HTEIVA VSHVAEETLQ QRLNEFKNTK AAKLSVQKFR ENDVEDGEAR PPSFVKNRKK ERKI KDSLD KEEMFQTSEE ALNKNPILTQ VLGEQELSSK EVLFYLKQFS ERRARVVERI KATNG IDGE NIYHEGSENE TRKRKLSEVS IQNEHVEGED KETEGTEEKV KKVKTKTSEE KKENES GHF QDAIDGYSLE TDPYCPRNLH LLPTTDTYLS KVSDDPDNLE DVDDEELNAH LLNEEAS KL KERIWIGLNA DFLLEQESKR LKQEADIATG NTSVKKKRTR RRNNTRSDEP TKTVDAAA A IGLMSDLQDK SGLHAALKAA EESGDFTTAD SVKNMLQKAS FSKKINYDAI DGLFRLEHHH HHH |
-分子 #5: TATA-box-binding protein
| 分子 | 名称: TATA-box-binding protein / タイプ: protein_or_peptide / ID: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGIVPTLQNI VATVTLGCRL DLKTVALHAR NAEYNPKRFA AVIMRIREPK TTALIFASGK MVVTGAKSE DDSKLASRKY ARIIQKIGFA AKFTDFKIQN IVGSCDVKFP IRLEGLAFSH G TFSSYEPE LFPGLIYRMV KPKIVLLIFV SGKIVLTGAK QREEIYQAFE AIYPVLSEFR KM |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Acetate |
| 詳細 | Crosslinked sample |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 平均電子線量: 20.0 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Envelope score |
|---|
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)