+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10687 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Multibody refinement of 50S ribosome body of pseudouridimycin-stalled Mycoplasma pneumoniae in-cell expressome | |||||||||
 マップデータ マップデータ | 50S ribosome body of stalled expressome from pseudouridimycin-treated Mycoplasma pneumoniae cells, multibody refinement | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Mycoplasma pneumoniae M129 (バクテリア) Mycoplasma pneumoniae M129 (バクテリア) | |||||||||
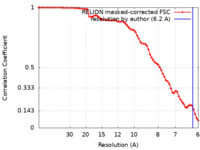
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 6.2 Å | |||||||||
 データ登録者 データ登録者 | Mahamid J / Xue L | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2020 ジャーナル: Science / 年: 2020タイトル: In-cell architecture of an actively transcribing-translating expressome. 著者: Francis J O'Reilly / Liang Xue / Andrea Graziadei / Ludwig Sinn / Swantje Lenz / Dimitry Tegunov / Cedric Blötz / Neil Singh / Wim J H Hagen / Patrick Cramer / Jörg Stülke / Julia Mahamid / Juri Rappsilber /   要旨: Structural biology studies performed inside cells can capture molecular machines in action within their native context. In this work, we developed an integrative in-cell structural approach using the ...Structural biology studies performed inside cells can capture molecular machines in action within their native context. In this work, we developed an integrative in-cell structural approach using the genome-reduced human pathogen We combined whole-cell cross-linking mass spectrometry, cellular cryo-electron tomography, and integrative modeling to determine an in-cell architecture of a transcribing and translating expressome at subnanometer resolution. The expressome comprises RNA polymerase (RNAP), the ribosome, and the transcription elongation factors NusG and NusA. We pinpointed NusA at the interface between a NusG-bound elongating RNAP and the ribosome and propose that it can mediate transcription-translation coupling. Translation inhibition dissociated the expressome, whereas transcription inhibition stalled and rearranged it. Thus, the active expressome architecture requires both translation and transcription elongation within the cell. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10687.map.gz emd_10687.map.gz | 2.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10687-v30.xml emd-10687-v30.xml emd-10687.xml emd-10687.xml | 15.5 KB 15.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10687_fsc.xml emd_10687_fsc.xml | 7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10687.png emd_10687.png | 59.3 KB | ||
| マスクデータ |  emd_10687_msk_1.map emd_10687_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10687_half_map_1.map.gz emd_10687_half_map_1.map.gz emd_10687_half_map_2.map.gz emd_10687_half_map_2.map.gz | 22.1 MB 22.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10687 http://ftp.pdbj.org/pub/emdb/structures/EMD-10687 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10687 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10687 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10687_validation.pdf.gz emd_10687_validation.pdf.gz | 428.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10687_full_validation.pdf.gz emd_10687_full_validation.pdf.gz | 427.8 KB | 表示 | |
| XML形式データ |  emd_10687_validation.xml.gz emd_10687_validation.xml.gz | 12.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10687 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10687 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10687 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10687 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10687.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10687.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 50S ribosome body of stalled expressome from pseudouridimycin-treated Mycoplasma pneumoniae cells, multibody refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10687_msk_1.map emd_10687_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: 50S ribosome body of stalled expressome from pseudouridimycin-treated...
| ファイル | emd_10687_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 50S ribosome body of stalled expressome from pseudouridimycin-treated Mycoplasma pneumoniae cells, multibody refinement, half 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: 50S ribosome body of stalled expressome from pseudouridimycin-treated...
| ファイル | emd_10687_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 50S ribosome body of stalled expressome from pseudouridimycin-treated Mycoplasma pneumoniae cells, multibody refinement, half 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : wild-type Mycoplasma pneumoniae M129 cells were treated with 0.4 ...
| 全体 | 名称: wild-type Mycoplasma pneumoniae M129 cells were treated with 0.4 mg/ml pseudouridimycin (PUM), 15-20 minutes prior to blotting and vitrification. |
|---|---|
| 要素 |
|
-超分子 #1: wild-type Mycoplasma pneumoniae M129 cells were treated with 0.4 ...
| 超分子 | 名称: wild-type Mycoplasma pneumoniae M129 cells were treated with 0.4 mg/ml pseudouridimycin (PUM), 15-20 minutes prior to blotting and vitrification. タイプ: cell / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  Mycoplasma pneumoniae M129 (バクテリア) Mycoplasma pneumoniae M129 (バクテリア) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 詳細: modified Hayflick medium as described in Halbedel, Hames, and Stulke 2004, with 0.4 mg/ml pseudouridimycin (PUM) |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: GOLD / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 45 % / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 2.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 4.5 µm / 最小 デフォーカス(補正後): 1.5 µm / 倍率(補正後): 81000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)