登録情報 データベース : EMDB / ID : EMD-10414タイトル SAGA Core module local resolution filtered map / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
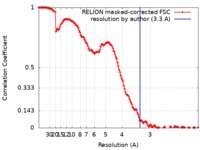
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Saccharomyces cerevisiae S288C (パン酵母) / Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母)手法 / / 解像度 : 3.3 Å Wang H / Cheung A 資金援助 Organization Grant number 国 German Research Foundation SFB860, SPP1935, EXC 2067/1-390729940 European Research Council 693023
ジャーナル : Nature / 年 : 2020タイトル : Structure of the transcription coactivator SAGA.著者 : Haibo Wang / Christian Dienemann / Alexandra Stützer / Henning Urlaub / Alan C M Cheung / Patrick Cramer / 要旨 : Gene transcription by RNA polymerase II is regulated by activator proteins that recruit the coactivator complexes SAGA (Spt-Ada-Gcn5-acetyltransferase) and transcription factor IID (TFIID). SAGA is ... Gene transcription by RNA polymerase II is regulated by activator proteins that recruit the coactivator complexes SAGA (Spt-Ada-Gcn5-acetyltransferase) and transcription factor IID (TFIID). SAGA is required for all regulated transcription and is conserved among eukaryotes. SAGA contains four modules: the activator-binding Tra1 module, the core module, the histone acetyltransferase (HAT) module and the histone deubiquitination (DUB) module. Previous studies provided partial structures, but the structure of the central core module is unknown. Here we present the cryo-electron microscopy structure of SAGA from the yeast Saccharomyces cerevisiae and resolve the core module at 3.3 Å resolution. The core module consists of subunits Taf5, Sgf73 and Spt20, and a histone octamer-like fold. The octamer-like fold comprises the heterodimers Taf6-Taf9, Taf10-Spt7 and Taf12-Ada1, and two histone-fold domains in Spt3. Spt3 and the adjacent subunit Spt8 interact with the TATA box-binding protein (TBP). The octamer-like fold and its TBP-interacting region are similar in TFIID, whereas Taf5 and the Taf6 HEAT domain adopt distinct conformations. Taf12 and Spt20 form flexible connections to the Tra1 module, whereas Sgf73 tethers the DUB module. Binding of a nucleosome to SAGA displaces the HAT and DUB modules from the core-module surface, allowing the DUB module to bind one face of an ubiquitinated nucleosome. 履歴 登録 2019年10月28日 - ヘッダ(付随情報) 公開 2019年11月27日 - マップ公開 2020年1月29日 - 更新 2024年5月22日 - 現状 2024年5月22日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報

 データ登録者
データ登録者 ドイツ, 2件
ドイツ, 2件  引用
引用 ジャーナル: Nature / 年: 2020
ジャーナル: Nature / 年: 2020

 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_10414.map.gz
emd_10414.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-10414-v30.xml
emd-10414-v30.xml emd-10414.xml
emd-10414.xml EMDBヘッダ
EMDBヘッダ emd_10414_fsc.xml
emd_10414_fsc.xml FSCデータファイル
FSCデータファイル emd_10414.png
emd_10414.png emd-10414.cif.gz
emd-10414.cif.gz emd_10414_half_map_1.map.gz
emd_10414_half_map_1.map.gz emd_10414_half_map_2.map.gz
emd_10414_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-10414
http://ftp.pdbj.org/pub/emdb/structures/EMD-10414 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10414
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10414 emd_10414_validation.pdf.gz
emd_10414_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_10414_full_validation.pdf.gz
emd_10414_full_validation.pdf.gz emd_10414_validation.xml.gz
emd_10414_validation.xml.gz emd_10414_validation.cif.gz
emd_10414_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10414
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10414 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10414
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10414 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_10414.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_10414.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー




























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)