+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10338 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | XPF-ERCC1 Cryo-EM Structure, DNA-Bound form | |||||||||
 マップデータ マップデータ | Sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA Repair enzyme. Nucleotide excision repair / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of t-circle formation / pyrimidine dimer repair by nucleotide-excision repair / telomeric DNA-containing double minutes formation / ERCC4-ERCC1 complex / negative regulation of protection from non-homologous end joining at telomere / syncytium formation / nucleotide-excision repair factor 1 complex / nucleotide-excision repair involved in interstrand cross-link repair / nucleotide-excision repair complex / t-circle formation ...positive regulation of t-circle formation / pyrimidine dimer repair by nucleotide-excision repair / telomeric DNA-containing double minutes formation / ERCC4-ERCC1 complex / negative regulation of protection from non-homologous end joining at telomere / syncytium formation / nucleotide-excision repair factor 1 complex / nucleotide-excision repair involved in interstrand cross-link repair / nucleotide-excision repair complex / t-circle formation / negative regulation of telomere maintenance / single-stranded DNA endodeoxyribonuclease activity / resolution of meiotic recombination intermediates / mitotic recombination / UV protection / post-embryonic hemopoiesis / isotype switching / UV-damage excision repair / negative regulation of telomere maintenance via telomere lengthening / HDR through Single Strand Annealing (SSA) / oogenesis / TFIID-class transcription factor complex binding / replicative senescence / response to X-ray / positive regulation of transcription initiation by RNA polymerase II / interstrand cross-link repair / response to UV / telomere maintenance / insulin-like growth factor receptor signaling pathway / Fanconi Anemia Pathway / DNA endonuclease activity / determination of adult lifespan / double-strand break repair via homologous recombination / nucleotide-excision repair / promoter-specific chromatin binding / Dual Incision in GG-NER / Formation of Incision Complex in GG-NER / Dual incision in TC-NER / double-strand break repair via nonhomologous end joining / multicellular organism growth / male gonad development / cellular response to UV / single-stranded DNA binding / response to oxidative stress / spermatogenesis / damaged DNA binding / 加水分解酵素; エステル加水分解酵素 / chromosome, telomeric region / cell population proliferation / regulation of autophagy / DNA repair / nucleoplasm / identical protein binding / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.9 Å | |||||||||
 データ登録者 データ登録者 | Jones ML / Briggs DC | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Cryo-EM structures of the XPF-ERCC1 endonuclease reveal how DNA-junction engagement disrupts an auto-inhibited conformation. 著者: Morgan Jones / Fabienne Beuron / Aaron Borg / Andrea Nans / Christopher P Earl / David C Briggs / Ambrosius P Snijders / Maureen Bowles / Edward P Morris / Mark Linch / Neil Q McDonald /  要旨: The structure-specific endonuclease XPF-ERCC1 participates in multiple DNA damage repair pathways including nucleotide excision repair (NER) and inter-strand crosslink repair (ICLR). How XPF-ERCC1 is ...The structure-specific endonuclease XPF-ERCC1 participates in multiple DNA damage repair pathways including nucleotide excision repair (NER) and inter-strand crosslink repair (ICLR). How XPF-ERCC1 is catalytically activated by DNA junction substrates is not currently understood. Here we report cryo-electron microscopy structures of both DNA-free and DNA-bound human XPF-ERCC1. DNA-free XPF-ERCC1 adopts an auto-inhibited conformation in which the XPF helical domain masks the ERCC1 (HhH) domain and restricts access to the XPF catalytic site. DNA junction engagement releases the ERCC1 (HhH) domain to couple with the XPF-ERCC1 nuclease/nuclease-like domains. Structure-function data indicate xeroderma pigmentosum patient mutations frequently compromise the structural integrity of XPF-ERCC1. Fanconi anaemia patient mutations in XPF often display substantial in-vitro activity but are resistant to activation by ICLR recruitment factor SLX4. Our data provide insights into XPF-ERCC1 architecture and catalytic activation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10338.map.gz emd_10338.map.gz | 20.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10338-v30.xml emd-10338-v30.xml emd-10338.xml emd-10338.xml | 15.6 KB 15.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10338.png emd_10338.png | 57.3 KB | ||
| Filedesc metadata |  emd-10338.cif.gz emd-10338.cif.gz | 6.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10338 http://ftp.pdbj.org/pub/emdb/structures/EMD-10338 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10338 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10338 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10338_validation.pdf.gz emd_10338_validation.pdf.gz | 493.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10338_full_validation.pdf.gz emd_10338_full_validation.pdf.gz | 492.7 KB | 表示 | |
| XML形式データ |  emd_10338_validation.xml.gz emd_10338_validation.xml.gz | 5.5 KB | 表示 | |
| CIF形式データ |  emd_10338_validation.cif.gz emd_10338_validation.cif.gz | 6.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10338 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10338 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10338 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10338 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10338.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10338.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.38 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
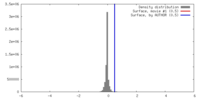
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : ERCC1/XPF/DNA complex
| 全体 | 名称: ERCC1/XPF/DNA complex |
|---|---|
| 要素 |
|
-超分子 #1: ERCC1/XPF/DNA complex
| 超分子 | 名称: ERCC1/XPF/DNA complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: ERCC1/XPF
| 超分子 | 名称: ERCC1/XPF / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: DNA
| 超分子 | 名称: DNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3-#4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: DNA repair endonuclease XPF
| 分子 | 名称: DNA repair endonuclease XPF / タイプ: protein_or_peptide / ID: 1 詳細: Some residues have been modelled as Alanine where sidechains were not visible. コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 104.636156 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MESGQPARRI AMAPLLEYER QLVLELLDTD GLVVCARGLG ADRLLYHFLQ LHCHPACLVL VLNTQPAEEE YFINQLKIEG VEHLPRRVT NEITSNSRYE VYTQGGVIFA TSRILVVDFL TDRIPSDLIT GILVYRAHRI IESCQEAFIL RLFRQKNKRG F IKAFTDNA ...文字列: MESGQPARRI AMAPLLEYER QLVLELLDTD GLVVCARGLG ADRLLYHFLQ LHCHPACLVL VLNTQPAEEE YFINQLKIEG VEHLPRRVT NEITSNSRYE VYTQGGVIFA TSRILVVDFL TDRIPSDLIT GILVYRAHRI IESCQEAFIL RLFRQKNKRG F IKAFTDNA VAFDTGFCHV ERVMRNLFVR KLYLWPRFHV AVNSFLEQHK PEVVEIHVSM TPTMLAIQTA ILDILNACLK EL KCHNPSL EVEDLSLENA IGKPFDKTIR HYLDPLWHQL GAKTKSLVQD LKILRTLLQY LSQYDCVTFL NLLESLRATE KAF GQNSGW LFLDSSTSMF INARARVYHL PDAKMSKKEK ISEKMEIKEG EETKKELVLE SNPKWEALTE VLKEIEAENK ESEA LGGPG QVLICASDDR TCSQLRDYIT LGAEAFLLRL YRKTFEKDSK AEEVWMKFRK EDSSKRIRKS HKRPKDPQNK ERAST KERT LKKKKRKLTL TQMVGKPEEL EEEGDVEEGY RREISSSPES CPEEIKHEEF DVNLSSDAAF GILKEPLTII HPLLGC SDP YALTRVLHEV EPRYVVLYDA ELTFVRQLEI YRASRPGKPL RVYFLIYGGS TEEQRYLTAL RKEKEAFEKL IREKASM VV PEEREGRDET NLDLVRGTAS ADVSTDTRKA GGQEQNGTQQ SIVVDMREFR SELPSLIHRR GIDIEPVTLE VGDYILTP E MCVERKSISD LIGSLNNGRL YSQCISMSRY YKRPVLLIEF DPSKPFSLTS RGALFQEISS NDISSKLTLL TLHFPRLRI LWCPSPHATA ELFEELKQSK PQPDAATALA ITADSETLPE SEKYNPGPQD FLLKMPGVNA KNCRSLMHHV KNIAELAALS QDELTSILG NAANAKQLYD FIHTSFAEVV SKGKGKK UniProtKB: DNA repair endonuclease XPF |
-分子 #2: DNA excision repair protein ERCC-1
| 分子 | 名称: DNA excision repair protein ERCC-1 / タイプ: protein_or_peptide / ID: 2 詳細: Some residues modelled as Alanine where no sidechain information available. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 32.598301 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDPGKDKEGV PQPSGPPARK KFVIPLDEDE VPPGVAKPLF RSTQSLPTVD TSAQAAPQTY AEYAISQPLE GAGATCPTGS EPLAGETPN QALKPGAKSN SIIVSPRQRG NPVLKFVRNV PWEFGDVIPD YVLGQSTCAL FLSLRYHNLH PDYIHGRLQS L GKNFALRV ...文字列: MDPGKDKEGV PQPSGPPARK KFVIPLDEDE VPPGVAKPLF RSTQSLPTVD TSAQAAPQTY AEYAISQPLE GAGATCPTGS EPLAGETPN QALKPGAKSN SIIVSPRQRG NPVLKFVRNV PWEFGDVIPD YVLGQSTCAL FLSLRYHNLH PDYIHGRLQS L GKNFALRV LLVQVDVKDP QQALKELAKM CILADCTLIL AWSPEEAGRY LETYKAYEQK PADLLMEKLE QDFVSRVTEC LT TVKSVNK TDSQTLLTTF GSLEQLIAAS REDLALCPGL GPQKARRLFD VLHEPFLKVP UniProtKB: DNA excision repair protein ERCC-1 |
-分子 #3: DNA (5'-D(P*CP*AP*GP*AP*TP*GP*CP*TP*GP*A)-3')
| 分子 | 名称: DNA (5'-D(P*CP*AP*GP*AP*TP*GP*CP*TP*GP*A)-3') / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 3.06903 KDa |
| 配列 | 文字列: (DC)(DA)(DG)(DA)(DT)(DG)(DC)(DT)(DG)(DA) |
-分子 #4: DNA (5'-D(*TP*CP*AP*GP*CP*AP*TP*CP*TP*G)-3')
| 分子 | 名称: DNA (5'-D(*TP*CP*AP*GP*CP*AP*TP*CP*TP*G)-3') / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 3.019992 KDa |
| 配列 | 文字列: (DT)(DC)(DA)(DG)(DC)(DA)(DT)(DC)(DT)(DG) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 詳細: 20 mM HEPES pH 7.8, 150 mM NaCl, 1 mM TCEP, 0.01% CHAPS |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 2-17 / 実像数: 15315 / 平均電子線量: 63.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 4.5 µm / 最小 デフォーカス(補正後): 0.9 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.2 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 7.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 2) / 使用した粒子像数: 198212 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)