+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1016 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
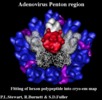
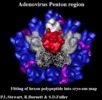
| タイトル | Difference imaging of adenovirus: bridging the resolution gap between X-ray crystallography and electron microscopy. | |||||||||
 マップデータ マップデータ | Adeno-virus type 2. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Human adenovirus 2 (ヒトアデノウイルス) Human adenovirus 2 (ヒトアデノウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 25.0 Å | |||||||||
 データ登録者 データ登録者 | Stewart PL / Fuller SD / Burnett RM | |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 1993 ジャーナル: EMBO J / 年: 1993タイトル: Difference imaging of adenovirus: bridging the resolution gap between X-ray crystallography and electron microscopy. 著者: P L Stewart / S D Fuller / R M Burnett /  要旨: While X-ray crystallography provides atomic resolution structures of proteins and small viruses, electron microscopy provides complementary structural information on the organization of larger ...While X-ray crystallography provides atomic resolution structures of proteins and small viruses, electron microscopy provides complementary structural information on the organization of larger assemblies at lower resolution. A novel combination of these two techniques has bridged this resolution gap and revealed the various structural components forming the capsid of human type 2 adenovirus. An image reconstruction of the intact virus, derived from cryo-electron micrographs, was deconvolved with an approximate contrast transfer function to mitigate microscope distortions. A model capsid was calculated from 240 copies of the crystallographic structure of the major capsid protein and filtered to the correct resolution. Subtraction of the calculated capsid from the corrected reconstruction gave a three-dimensional difference map revealing the minor proteins that stabilize the virion. Elongated density penetrating the hexon capsid at the facet edges was ascribed to polypeptide IIIa, a component required for virion assembly. Density on the inner surface of the capsid, connecting the ring of peripentonal hexons, was assigned as polypeptide VI, a component that binds DNA. Identification of the regions of hexon that contact the penton base suggests a structural mechanism for previously proposed events during cell entry. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1016.map.gz emd_1016.map.gz | 2.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1016-v30.xml emd-1016-v30.xml emd-1016.xml emd-1016.xml | 15.8 KB 15.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1016.gif 1016.gif emd_1016.tif emd_1016.tif emd_emd_1016.tif emd_emd_1016.tif | 28.8 KB 108 KB 108 KB | ||
| その他 |  emd_1016_additional_1.map.gz emd_1016_additional_1.map.gz emd_1016_additional_2.map.gz emd_1016_additional_2.map.gz emd_1016_additional_3.map.gz emd_1016_additional_3.map.gz | 23.1 KB 22.6 KB 24.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1016 http://ftp.pdbj.org/pub/emdb/structures/EMD-1016 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1016 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1016 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1016_validation.pdf.gz emd_1016_validation.pdf.gz | 248.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1016_full_validation.pdf.gz emd_1016_full_validation.pdf.gz | 247.3 KB | 表示 | |
| XML形式データ |  emd_1016_validation.xml.gz emd_1016_validation.xml.gz | 6.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1016 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1016 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1016 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1016 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1016.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) ダウンロード / ファイル: emd_1016.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Adeno-virus type 2. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 7.3 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: emd 1016 additional 1.map
| ファイル | emd_1016_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-添付マップデータ: emd 1016 additional 2.map
| ファイル | emd_1016_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-添付マップデータ: emd 1016 additional 3.map
| ファイル | emd_1016_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : adenovirus type 2
| 全体 | 名称: adenovirus type 2 |
|---|---|
| 要素 |
|
-超分子 #1000: adenovirus type 2
| 超分子 | 名称: adenovirus type 2 / タイプ: sample / ID: 1000 / 集合状態: T=25 particle / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 150 MDa |
-超分子 #1: Human adenovirus 2
| 超分子 | 名称: Human adenovirus 2 / タイプ: virus / ID: 1 / NCBI-ID: 10515 / 生物種: Human adenovirus 2 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) / 別称: VERTEBRATES Homo sapiens (ヒト) / 別称: VERTEBRATES |
| ウイルス殻 | Shell ID: 1 / 名称: fibres (5f positions) 1090A / T番号(三角分割数): 25 |
| ウイルス殻 | Shell ID: 2 / 名称: capsid (along 2f) 884A / T番号(三角分割数): 1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 10 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 詳細: Sample stored in CsCl was diluted in Tris (10mM) NaCl (100 mM) pH 7.4 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 60 % / チャンバー内温度: 23 K / 装置: HOMEMADE PLUNGER 詳細: Vitrification instrument: EMBL plunger. plunging at ambient temperature and humidity 手法: Blot for 2 sec |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS EM400 |
|---|---|
| 温度 | 平均: 105 K |
| 詳細 | Details of the microscopy are in Stewart, P. L., Burnett, R. M., Cyrklaff, M. and Fuller, S. D. (1991). Image reconstruction reveals the complex molecular organization of adenovirus. Cell 67, 145-54. Images were taken in triples at 1, 2.6 and 8.8 microns underfocus. Some images were taken at 12 degrees tilt to compensate for the preference for the virus to orient with the fibres away from the water layer. For image reconstruction see: Stewart, P. L., Burnett, R. M.,Cyrklaff, M. and Fuller, S. D. (1991). Image reconstruction reveals the complex molecular organization of adenovirus. Cell 67, 145-54. For reconstruction details see: P.L. Stewart, S.D. Fuller, R. M. Burnett, (1993). Difference imaging of adenovirus: bridging the resolution gap between X- ray crystallography and electron microscopy EMBO J.Stewart PL, Burnett RM. Adenovirus structure by X-ray crystallography and electron microscopy. Curr Top Microbiol Immunol. 1995, 199 (1):25-38. The PDB code for the Ad2 hexon model is 1DHX, and the reference Athappilly, F. K., Murali, R., Rux, J. J., Cai, Z., Burnett, R. M.: The refined crystal structure of hexon, the major coat protein of adenovirus type 2, at 2.9 A resolution. J Mol Biol 242 pp. 430 (1994) |
| 日付 | 1990年1月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: OPTRONICS / デジタル化 - サンプリング間隔: 25 µm / 実像数: 50 / 平均電子線量: 8 e/Å2 / Od range: 1 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 80 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 8.8 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 34000 |
| 試料ステージ | 試料ホルダー: eucentric - Philips EM400 / 試料ホルダーモデル: GATAN LIQUID NITROGEN / Tilt angle max: 12 |
- 画像解析
画像解析
| 詳細 | Purified by centrifugation in CsCl |
|---|---|
| CTF補正 | 詳細: initially different defocuses were combined by summing in resolution bands |
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 25.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMBL-ICOS / 使用した粒子像数: 29 |
| 最終 角度割当 | 詳細: sufficient to give maximum inverse eigenvalue of 0.1 |
-原子モデル構築 1
| ソフトウェア | 名称: maximizing cross correlation |
|---|---|
| 詳細 | Protocol: rigid body |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 3000 / 当てはまり具合の基準: max cross correlation |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)













































