+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0903 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Heteromeric amino acid transporter b0,+AT-rBAT complex bound with Arginine | ||||||||||||||||||||||||
 マップデータ マップデータ | cryo EM map of b0, AT-rBAT complex bound with Arginine | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
 キーワード キーワード | transporter / MEMBRANE PROTEIN | ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報broad specificity neutral L-amino acid:basic L-amino acid antiporter activity / Defective SLC3A1 causes cystinuria (CSNU) / Defective SLC7A9 causes cystinuria (CSNU) / basic amino acid transport / basic amino acid transmembrane transporter activity / L-cystine transmembrane transporter activity / L-cystine transport / amino acid transmembrane transport / aspartate transmembrane transport / L-glutamate transmembrane transport ...broad specificity neutral L-amino acid:basic L-amino acid antiporter activity / Defective SLC3A1 causes cystinuria (CSNU) / Defective SLC7A9 causes cystinuria (CSNU) / basic amino acid transport / basic amino acid transmembrane transporter activity / L-cystine transmembrane transporter activity / L-cystine transport / amino acid transmembrane transport / aspartate transmembrane transport / L-glutamate transmembrane transport / neutral amino acid transport / neutral L-amino acid transmembrane transporter activity / amino acid transmembrane transporter activity / Amino acid transport across the plasma membrane / vacuolar membrane / antiporter activity / Basigin interactions / amino acid transport / brush border membrane / peptide antigen binding / protein-containing complex assembly / gene expression / carbohydrate metabolic process / apical plasma membrane / protein heterodimerization activity / protein-containing complex binding / extracellular exosome / membrane / plasma membrane 類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.3 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Yan RH / Li YN | ||||||||||||||||||||||||
| 資金援助 |  中国, 7件 中国, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2020 ジャーナル: Sci Adv / 年: 2020タイトル: Cryo-EM structure of the human heteromeric amino acid transporter bAT-rBAT. 著者: Renhong Yan / Yaning Li / Yi Shi / Jiayao Zhou / Jianlin Lei / Jing Huang / Qiang Zhou /  要旨: Heteromeric amino acid transporters (HATs) catalyze the transmembrane movement of amino acids, comprising two subunits, a heavy chain and a light chain, linked by a disulfide bridge. The bAT (SLC7A9) ...Heteromeric amino acid transporters (HATs) catalyze the transmembrane movement of amino acids, comprising two subunits, a heavy chain and a light chain, linked by a disulfide bridge. The bAT (SLC7A9) is a representative light chain of HATs, forming heterodimer with rBAT, a heavy chain which mediates the membrane trafficking of bAT. The bAT-rBAT complex is an obligatory exchanger, which mediates the influx of cystine and cationic amino acids and the efflux of neutral amino acids in kidney and small intestine. Here, we report the cryo-EM structure of the human bAT-rBAT complex alone and in complex with arginine substrate at resolution of 2.7 and 2.3 Å, respectively. The overall structure of bAT-rBAT exists as a dimer of heterodimer consistent with the previous study. A ligand molecule is bound to the substrate binding pocket, near which an occluded pocket is identified, to which we found that it is important for substrate transport. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0903.map.gz emd_0903.map.gz | 116.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0903-v30.xml emd-0903-v30.xml emd-0903.xml emd-0903.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_0903.png emd_0903.png | 40.5 KB | ||
| Filedesc metadata |  emd-0903.cif.gz emd-0903.cif.gz | 6.4 KB | ||
| その他 |  emd_0903_additional_1.map.gz emd_0903_additional_1.map.gz emd_0903_additional_2.map.gz emd_0903_additional_2.map.gz | 116.9 MB 116.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0903 http://ftp.pdbj.org/pub/emdb/structures/EMD-0903 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0903 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0903 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0903.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0903.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryo EM map of b0, AT-rBAT complex bound with Arginine | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.091 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
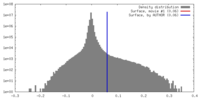
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: cryo EM map of b0, AT-rBAT complex bound...
| ファイル | emd_0903_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryo EM map of b0, AT-rBAT complex bound with Arginine, focused refined on TM domain | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
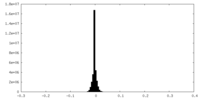
| 密度ヒストグラム |
-追加マップ: cryo EM map of b0, AT-rBAT complex bound...
| ファイル | emd_0903_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryo EM map of b0, AT-rBAT complex bound with Arginine, focused refined on soluble domain | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
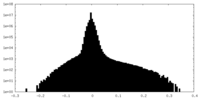
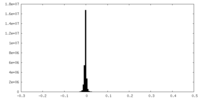
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : b0,+AT-rBAT complex bound with Arginine
| 全体 | 名称: b0,+AT-rBAT complex bound with Arginine |
|---|---|
| 要素 |
|
-超分子 #1: b0,+AT-rBAT complex bound with Arginine
| 超分子 | 名称: b0,+AT-rBAT complex bound with Arginine / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 120 KDa |
-分子 #1: Neutral and basic amino acid transport protein rBAT
| 分子 | 名称: Neutral and basic amino acid transport protein rBAT / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 80.691586 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAHHHHHHHH HHSGRAEDKS KRDSIEMSMK GCQTNNGFVH NEDILEQTPD PGSSTDNLKH STRGILGSQE PDFKGVQPYA GMPKEVLFQ FSGQARYRIP REILFWLTVA SVLVLIAATI AIIALSPKCL DWWQEGPMYQ IYPRSFKDSN KDGNGDLKGI Q DKLDYITA ...文字列: MAHHHHHHHH HHSGRAEDKS KRDSIEMSMK GCQTNNGFVH NEDILEQTPD PGSSTDNLKH STRGILGSQE PDFKGVQPYA GMPKEVLFQ FSGQARYRIP REILFWLTVA SVLVLIAATI AIIALSPKCL DWWQEGPMYQ IYPRSFKDSN KDGNGDLKGI Q DKLDYITA LNIKTVWITS FYKSSLKDFR YGVEDFREVD PIFGTMEDFE NLVAAIHDKG LKLIIDFIPN HTSDKHIWFQ LS RTRTGKY TDYYIWHDCT HENGKTIPPN NWLSVYGNSS WHFDEVRNQC YFHQFMKEQP DLNFRNPDVQ EEIKEILRFW LTK GVDGFS LDAVKFLLEA KHLRDEIQVN KTQIPDTVTQ YSELYHDFTT TQVGMHDIVR SFRQTMDQYS TEPGRYRFMG TEAY AESID RTVMYYGLPF IQEADFPFNN YLSMLDTVSG NSVYEVITSW MENMPEGKWP NWMIGGPDSS RLTSRLGNQY VNVMN MLLF TLPGTPITYY GEEIGMGNIV AANLNESYDI NTLRSKSPMQ WDNSSNAGFS EASNTWLPTN SDYHTVNVDV QKTQPR SAL KLYQDLSLLH ANELLLNRGW FCHLRNDSHY VVYTRELDGI DRIFIVVLNF GESTLLNLHN MISGLPAKMR IRLSTNS AD KGSKVDTSGI FLDKGEGLIF EHNTKNLLHR QTAFRDRCFV SNRACYSSVL NILYTSC UniProtKB: Amino acid transporter heavy chain SLC3A1 |
-分子 #2: b(0,+)-type amino acid transporter 1
| 分子 | 名称: b(0,+)-type amino acid transporter 1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 55.656105 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MADYKDDDDK SGPDEVDASG RGDTGLRKRR EDEKSIQSQE PKTTSLQKEL GLISGISIIV GTIIGSGIFV SPKSVLSNTE AVGPCLIIW AACGVLATLG ALCFAELGTM ITKSGGEYPY LMEAYGPIPA YLFSWASLIV IKPTSFAIIC LSFSEYVCAP F YVGCKPPQ ...文字列: MADYKDDDDK SGPDEVDASG RGDTGLRKRR EDEKSIQSQE PKTTSLQKEL GLISGISIIV GTIIGSGIFV SPKSVLSNTE AVGPCLIIW AACGVLATLG ALCFAELGTM ITKSGGEYPY LMEAYGPIPA YLFSWASLIV IKPTSFAIIC LSFSEYVCAP F YVGCKPPQ IVVKCLAAAA ILFISTVNSL SVRLGSYVQN IFTAAKLVIV AIIIISGLVL LAQGNTKNFD NSFEGAQLSV GA ISLAFYN GLWAYDGWNQ LNYITEELRN PYRNLPLAII IGIPLVTACY ILMNVSYFTV MTATELLQSQ AVAVTFGDRV LYP ASWIVP LFVAFSTIGA ANGTCFTAGR LIYVAGREGH MLKVLSYISV RRLTPAPAII FYGIIATIYI IPGDINSLVN YFSF AAWLF YGLTILGLIV MRFTRKELER PIKVPVVIPV LMTLISVFLV LAPIISKPTW EYLYCVLFIL SGLLFYFLFV HYKFG WAQK ISKPITMHLQ MLMEVVPPEE DPE UniProtKB: b(0,+)-type amino acid transporter 1 |
-分子 #4: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #5: ARGININE
| 分子 | 名称: ARGININE / タイプ: ligand / ID: 5 / コピー数: 2 / 式: ARG |
|---|---|
| 分子量 | 理論値: 175.209 Da |
| Chemical component information |  ChemComp-ARG: |
-分子 #6: 1,2-DIACYL-GLYCEROL-3-SN-PHOSPHATE
| 分子 | 名称: 1,2-DIACYL-GLYCEROL-3-SN-PHOSPHATE / タイプ: ligand / ID: 6 / コピー数: 4 / 式: 3PH |
|---|---|
| 分子量 | 理論値: 704.998 Da |
| Chemical component information |  ChemComp-3PH: |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 246 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C2 (2回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 2.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3) / 使用した粒子像数: 665827 |
| 初期 角度割当 | タイプ: OTHER |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)