+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0523 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Yeast Ctf19 complex | |||||||||
 マップデータ マップデータ | Biological protomer from reconstruction of real space-subtracted images | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Kinetochore / CELL CYCLE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of meiotic DNA double-strand break formation involved in reciprocal meiotic recombination / COMA complex / maintenance of meiotic sister chromatid cohesion / Mis6-Sim4 complex / : / meiotic sister chromatid segregation / establishment of meiotic sister chromatid cohesion / ascospore formation / attachment of spindle microtubules to kinetochore / CENP-A containing chromatin assembly ...negative regulation of meiotic DNA double-strand break formation involved in reciprocal meiotic recombination / COMA complex / maintenance of meiotic sister chromatid cohesion / Mis6-Sim4 complex / : / meiotic sister chromatid segregation / establishment of meiotic sister chromatid cohesion / ascospore formation / attachment of spindle microtubules to kinetochore / CENP-A containing chromatin assembly / kinetochore assembly / outer kinetochore / protein localization to chromosome, centromeric region / establishment of mitotic sister chromatid cohesion / protein localization to kinetochore / spindle pole body / mitotic spindle assembly checkpoint signaling / DNA replication initiation / meiotic cell cycle / chromosome segregation / kinetochore / cell division / structural molecule activity / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
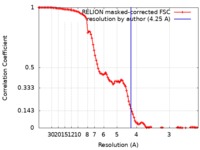
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.25 Å | |||||||||
 データ登録者 データ登録者 | Hinshaw SM / Harrison SC | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2019 ジャーナル: Elife / 年: 2019タイトル: The structure of the Ctf19c/CCAN from budding yeast. 著者: Stephen M Hinshaw / Stephen C Harrison /  要旨: Eukaryotic kinetochores connect spindlemicrotubules to chromosomal centromeres. A group of proteins called the Ctf19 complex (Ctf19c) in yeast and the constitutive centromere associated network (CCAN) ...Eukaryotic kinetochores connect spindlemicrotubules to chromosomal centromeres. A group of proteins called the Ctf19 complex (Ctf19c) in yeast and the constitutive centromere associated network (CCAN) in other organisms creates the foundation of a kinetochore. The Ctf19c/CCAN influences the timing of kinetochore assembly, sets its location by associating with a specialized nucleosome containing the histone H3 variant Cse4/CENP-A, and determines the organization of the microtubule attachment apparatus. We present here the structure of a reconstituted 13-subunit Ctf19c determined by cryo-electron microscopy at ~4 Å resolution. The structure accounts for known and inferred contacts with the Cse4 nucleosome and for an observed assembly hierarchy. We describe its implications for establishment of kinetochores and for their regulation by kinases throughout the cell cycle. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0523.map.gz emd_0523.map.gz | 6.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0523-v30.xml emd-0523-v30.xml emd-0523.xml emd-0523.xml | 27.8 KB 27.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0523_fsc.xml emd_0523_fsc.xml | 10.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0523.png emd_0523.png | 58.3 KB | ||
| Filedesc metadata |  emd-0523.cif.gz emd-0523.cif.gz | 8.2 KB | ||
| その他 |  emd_0523_additional.map.gz emd_0523_additional.map.gz | 97.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0523 http://ftp.pdbj.org/pub/emdb/structures/EMD-0523 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0523 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0523 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0523_validation.pdf.gz emd_0523_validation.pdf.gz | 382.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0523_full_validation.pdf.gz emd_0523_full_validation.pdf.gz | 381.9 KB | 表示 | |
| XML形式データ |  emd_0523_validation.xml.gz emd_0523_validation.xml.gz | 11.9 KB | 表示 | |
| CIF形式データ |  emd_0523_validation.cif.gz emd_0523_validation.cif.gz | 15.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0523 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0523 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0523 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0523 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0523.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0523.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Biological protomer from reconstruction of real space-subtracted images | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.23 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Dimeric assembly. Twofold symmetry enforced during reconstruction.
| ファイル | emd_0523_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Dimeric assembly. Twofold symmetry enforced during reconstruction. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Ctf19 complex
+超分子 #1: Ctf19 complex
+分子 #1: Inner kinetochore subunit IML3
+分子 #2: Inner kinetochore subunit MCM21
+分子 #3: Inner kinetochore subunit CTF19
+分子 #4: Inner kinetochore subunit CHL4
+分子 #5: Inner kinetochore subunit OKP1
+分子 #6: Inner kinetochore subunit NKP1
+分子 #7: Inner kinetochore subunit AME1
+分子 #8: Inner kinetochore subunit CTF3,Inner kinetochore subunit CTF3
+分子 #9: Inner kinetochore subunit NKP2
+分子 #10: Inner kinetochore subunit Mcm22
+分子 #11: Inner kinetochore subunit Mcm16
+分子 #12: Unknown (unassigned)
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8.5 |
|---|---|
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 47.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)