+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0488 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the TRPM8 ion channel in complex with high occupancy icilin, PI(4,5)P2, and calcium | |||||||||
 マップデータ マップデータ | em-volume_P1 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ion channel / TRP channel / TRPM channel / TRPM8 channel / cold sensing / lipid sensing / menthol / icilin / WS-12 / PI(4 / 5)P2 / cooling agent / MEMBRANE PROTEIN / calcium-permeable ion channel / TRANSPORT PROTEIN | |||||||||
| 生物種 |  Ficedula albicollis (シロエリヒタキ) Ficedula albicollis (シロエリヒタキ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Yin Y / Le SC | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2019 ジャーナル: Science / 年: 2019タイトル: Structural basis of cooling agent and lipid sensing by the cold-activated TRPM8 channel. 著者: Ying Yin / Son C Le / Allen L Hsu / Mario J Borgnia / Huanghe Yang / Seok-Yong Lee /  要旨: Transient receptor potential melastatin member 8 (TRPM8) is a calcium ion (Ca)-permeable cation channel that serves as the primary cold and menthol sensor in humans. Activation of TRPM8 by cooling ...Transient receptor potential melastatin member 8 (TRPM8) is a calcium ion (Ca)-permeable cation channel that serves as the primary cold and menthol sensor in humans. Activation of TRPM8 by cooling compounds relies on allosteric actions of agonist and membrane lipid phosphatidylinositol 4,5-bisphosphate (PIP), but lack of structural information has thus far precluded a mechanistic understanding of ligand and lipid sensing by TRPM8. Using cryo-electron microscopy, we determined the structures of TRPM8 in complex with the synthetic cooling compound icilin, PIP, and Ca, as well as in complex with the menthol analog WS-12 and PIP Our structures reveal the binding sites for cooling agonists and PIP in TRPM8. Notably, PIP binds to TRPM8 in two different modes, which illustrate the mechanism of allosteric coupling between PIP and agonists. This study provides a platform for understanding the molecular mechanism of TRPM8 activation by cooling agents. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0488.map.gz emd_0488.map.gz | 58.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0488-v30.xml emd-0488-v30.xml emd-0488.xml emd-0488.xml | 19.6 KB 19.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_0488.png emd_0488.png | 265.2 KB | ||
| Filedesc metadata |  emd-0488.cif.gz emd-0488.cif.gz | 7.1 KB | ||
| その他 |  emd_0488_half_map_1.map.gz emd_0488_half_map_1.map.gz emd_0488_half_map_2.map.gz emd_0488_half_map_2.map.gz | 45.6 MB 45.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0488 http://ftp.pdbj.org/pub/emdb/structures/EMD-0488 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0488 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0488 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0488_validation.pdf.gz emd_0488_validation.pdf.gz | 881.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0488_full_validation.pdf.gz emd_0488_full_validation.pdf.gz | 881.4 KB | 表示 | |
| XML形式データ |  emd_0488_validation.xml.gz emd_0488_validation.xml.gz | 12 KB | 表示 | |
| CIF形式データ |  emd_0488_validation.cif.gz emd_0488_validation.cif.gz | 14.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0488 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0488 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0488 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0488 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0488.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0488.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | em-volume_P1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: em-half-volume P1
| ファイル | emd_0488_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | em-half-volume_P1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: em-half-volume P2
| ファイル | emd_0488_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | em-half-volume_P2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Transient receptor potential melastatin member 8
| 全体 | 名称: Transient receptor potential melastatin member 8 |
|---|---|
| 要素 |
|
-超分子 #1: Transient receptor potential melastatin member 8
| 超分子 | 名称: Transient receptor potential melastatin member 8 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Ficedula albicollis (シロエリヒタキ) Ficedula albicollis (シロエリヒタキ) |
-分子 #1: Transient receptor potential cation channel subfamily M member 8
| 分子 | 名称: Transient receptor potential cation channel subfamily M member 8 タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Ficedula albicollis (シロエリヒタキ) Ficedula albicollis (シロエリヒタキ) |
| 分子量 | 理論値: 130.011391 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MATLFQVSMG SMRHRRNGNF ESSRLLYSSM SRSIDVACSD ADLANFIQEN FKKRECVFFT KDTKSMGNLC KCGYPENQHI EGTQVNTTE KWNYKK(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) ...文字列: MATLFQVSMG SMRHRRNGNF ESSRLLYSSM SRSIDVACSD ADLANFIQEN FKKRECVFFT KDTKSMGNLC KCGYPENQHI EGTQVNTTE KWNYKK(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)K YIRLSCDTDS ETLYDLMTQH WHLKTPNLVI SVTG GAKNF ALKPRMRKIF SRLIYIAQSK GAWIFTGGTH YGLMKYIGEV VRDNTISRSS EENVVAIGIA AWGMISNRET LIRTA DSDG SYLAHYIMDD LKRDPLYCLD NNHTHLLLVD NGTHGHPTIE AKVRTQLEKY ISERVIPESN YGGKIPIVCF AQGGGK ETL KSINVAIKSK IPCVVVEGSG RIADVIASLV EAEGTLASSC VKESLLRFLP RTISRLSEEE TESWIKWIKE VLESPHL LT VIKIEEAGDE IVSNAISFAL YKAFSTNEHD RDNWNGQLKL LLEWNQLDLA SDEIFTNDRN WESADLQDVM FTALVKDR P KFVRLFLENG LNLRKFLTTE VLRELYTNNF SSLVFKNLQI AKNSYNDALL TFVWKMVEDF RRGAKRDDKN SKDEMEIEL SEECPITRHP LQALFIWSVL QNKKELSKVI WEQTRGCTLA ALGASKLLKS MAKVKNDINA AGESEELANE YETRAVELFT ECYSNDEDL AEQLLTYSCE AWGGSNCLEL AVEARDQQFI AQPGVQNFLS KQWYGEISRD TKNWKIILCL FFFPLIGCGF I SFRKKPVE KTKKLFLYYV SFFTSPFVVF SWNVIFYIAF LLLFAYVLLM DFQKEPTALE IILYVLVFIL LCDEVRQWYM NG SKYFSDL WNVMDTLGIF YFIAGIVFRL HSDESSWYSG RVIFCLDYIV FTLRLIHIFT VSRNLGPKII MLQRMMIDVF FFL FLFAVW MVAFGVARQG ILRKNEHRWE WIFRSVIYEP YLAMFGQYPD DIDGTTYNFD HCTFSGNESK PLCVELDANN QPRF PEWIT IPLVCIYMLS TNILLVNLLV AMFGYTVGSV QENNDQVWKF QRFFLVQEYC SRLTIPFPFV IFAYIFMVMR KCFKC CCKK ESKEPSVCCS RNEDNEILAW EAVMKENYLV KINTKASDSS EE(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)KI KSNSLEVLFQ GPDYKDDDDK AHHHHHHHHH H |
-分子 #2: Icilin
| 分子 | 名称: Icilin / タイプ: ligand / ID: 2 / コピー数: 4 / 式: KX7 |
|---|---|
| 分子量 | 理論値: 311.292 Da |
| Chemical component information | 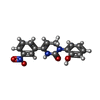 ChemComp-KX7: |
-分子 #3: (2S)-1-{[(R)-hydroxy{[(1R,2R,3S,4R,5R,6S)-2,3,6-trihydroxy-4,5-bi...
| 分子 | 名称: (2S)-1-{[(R)-hydroxy{[(1R,2R,3S,4R,5R,6S)-2,3,6-trihydroxy-4,5-bis(phosphonooxy)cyclohexyl]oxy}phosphoryl]oxy}-3-(octadecanoyloxy)propan-2-yl icosa-5,8,11,14-tetraenoate タイプ: ligand / ID: 3 / コピー数: 4 / 式: KXP |
|---|---|
| 分子量 | 理論値: 1.047088 KDa |
| Chemical component information | 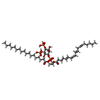 ChemComp-KXP: |
-分子 #4: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 2 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 26 sec. / 詳細: 15 mA |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 3705 / 平均露光時間: 60.0 sec. / 平均電子線量: 42.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.25 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)







































