+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0442 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Type III-A CRISPR Effector Subcomplex from Staphylococcus epidermidis RP62a | |||||||||
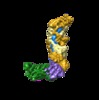
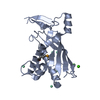
 マップデータ マップデータ | Type III-A CRISPR Effector Subcomplex from Staphylococcus epidermidis RP62a, primary map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌) | |||||||||
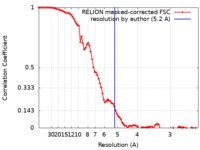
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.2 Å | |||||||||
 データ登録者 データ登録者 | Dorsey BW / Mondragon A | |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2019 ジャーナル: Nucleic Acids Res / 年: 2019タイトル: Structural organization of a Type III-A CRISPR effector subcomplex determined by X-ray crystallography and cryo-EM. 著者: Bryan W Dorsey / Lei Huang / Alfonso Mondragón /  要旨: Clustered regularly interspaced short palindromic repeats (CRISPR) and their associated Cas proteins provide an immune-like response in many prokaryotes against extraneous nucleic acids. CRISPR-Cas ...Clustered regularly interspaced short palindromic repeats (CRISPR) and their associated Cas proteins provide an immune-like response in many prokaryotes against extraneous nucleic acids. CRISPR-Cas systems are classified into different classes and types. Class 1 CRISPR-Cas systems form multi-protein effector complexes that includes a guide RNA (crRNA) used to identify the target for destruction. Here we present crystal structures of Staphylococcus epidermidis Type III-A CRISPR subunits Csm2 and Csm3 and a 5.2 Å resolution single-particle cryo-electron microscopy (cryo-EM) reconstruction of an in vivo assembled effector subcomplex including the crRNA. The structures help to clarify the quaternary architecture of Type III-A effector complexes, and provide details on crRNA binding, target RNA binding and cleavage, and intermolecular interactions essential for effector complex assembly. The structures allow a better understanding of the organization of Type III-A CRISPR effector complexes as well as highlighting the overall similarities and differences with other Class 1 effector complexes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0442.map.gz emd_0442.map.gz | 51.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0442-v30.xml emd-0442-v30.xml emd-0442.xml emd-0442.xml | 13.1 KB 13.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0442_fsc.xml emd_0442_fsc.xml | 8.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0442.png emd_0442.png | 64.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0442 http://ftp.pdbj.org/pub/emdb/structures/EMD-0442 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0442 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0442 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0442_validation.pdf.gz emd_0442_validation.pdf.gz | 78.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0442_full_validation.pdf.gz emd_0442_full_validation.pdf.gz | 77.8 KB | 表示 | |
| XML形式データ |  emd_0442_validation.xml.gz emd_0442_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0442 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0442 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0442 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0442 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0442.map.gz / 形式: CCP4 / 大きさ: 55.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0442.map.gz / 形式: CCP4 / 大きさ: 55.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Type III-A CRISPR Effector Subcomplex from Staphylococcus epidermidis RP62a, primary map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.24 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Type III-A CRISPR Effector Subcomplex from Staphylococcus epiderm...
| 全体 | 名称: Type III-A CRISPR Effector Subcomplex from Staphylococcus epidermidis RP62a |
|---|---|
| 要素 |
|
-超分子 #1: Type III-A CRISPR Effector Subcomplex from Staphylococcus epiderm...
| 超分子 | 名称: Type III-A CRISPR Effector Subcomplex from Staphylococcus epidermidis RP62a タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus epidermidis RP62A (表皮ブドウ球菌) Staphylococcus epidermidis RP62A (表皮ブドウ球菌) |
| 分子量 | 実験値: 255 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.20 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: C-flat-1.2/1.3 4C / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 詳細: Glow discharged with 15mA. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 99 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot for 4 seconds with +5 force before plunging.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FS |
|---|---|
| 温度 | 最低: 100.0 K / 最高: 100.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 7 / 実像数: 1037 / 平均露光時間: 8.0 sec. / 平均電子線量: 37.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 40323 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 30000 |
| 試料ステージ | 試料ホルダーモデル: GATAN 626 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)