+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0108 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-electron microscopic structure of the dihydrolipoamide succinyltransferase (E2) component of the human alpha-ketoglutarate (2-oxoglutarate) dehydrogenase complex [residues 218-453] | |||||||||||||||||||||
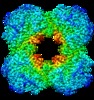
 マップデータ マップデータ | Cryo-electron microscopic map of the human dihydrolipoamide succinyltransferase [residues 218-453] | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | alpha-ketoglutarate dehydrogenase complex / 2-oxoglutarate dehydrogenase complex / dihydrolipoamide succinyltransferase / E2 component / TRANSFERASE | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報OGDH complex synthesizes succinyl-CoA from 2-OG / OADH complex synthesizes glutaryl-CoA from 2-OA / oxoadipate dehydrogenase complex / Glycine degradation / L-lysine catabolic process to acetyl-CoA via saccharopine / succinyl-CoA metabolic process / dihydrolipoyllysine-residue succinyltransferase / dihydrolipoyllysine-residue succinyltransferase activity / Protein lipoylation / oxoglutarate dehydrogenase complex ...OGDH complex synthesizes succinyl-CoA from 2-OG / OADH complex synthesizes glutaryl-CoA from 2-OA / oxoadipate dehydrogenase complex / Glycine degradation / L-lysine catabolic process to acetyl-CoA via saccharopine / succinyl-CoA metabolic process / dihydrolipoyllysine-residue succinyltransferase / dihydrolipoyllysine-residue succinyltransferase activity / Protein lipoylation / oxoglutarate dehydrogenase complex / 2-oxoglutarate metabolic process / acyltransferase activity / tricarboxylic acid cycle / generation of precursor metabolites and energy / mitochondrial matrix / mitochondrion / nucleoplasm / nucleus / membrane / cytosol 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Nagy B / Zambo Z | |||||||||||||||||||||
| 資金援助 |  ハンガリー, ハンガリー,  米国, 6件 米国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Biochim Biophys Acta Gen Subj / 年: 2021 ジャーナル: Biochim Biophys Acta Gen Subj / 年: 2021タイトル: Structure of the dihydrolipoamide succinyltransferase (E2) component of the human alpha-ketoglutarate dehydrogenase complex (hKGDHc) revealed by cryo-EM and cross-linking mass ...タイトル: Structure of the dihydrolipoamide succinyltransferase (E2) component of the human alpha-ketoglutarate dehydrogenase complex (hKGDHc) revealed by cryo-EM and cross-linking mass spectrometry: Implications for the overall hKGDHc structure. 著者: Balint Nagy / Martin Polak / Oliver Ozohanics / Zsofia Zambo / Eszter Szabo / Agnes Hubert / Frank Jordan / Jiří Novaček / Vera Adam-Vizi / Attila Ambrus /    要旨: BACKGROUND: The human mitochondrial alpha-ketoglutarate dehydrogenase complex (hKGDHc) converts KG to succinyl-CoA and NADH. Malfunction of and reactive oxygen species generation by the hKGDHc as ...BACKGROUND: The human mitochondrial alpha-ketoglutarate dehydrogenase complex (hKGDHc) converts KG to succinyl-CoA and NADH. Malfunction of and reactive oxygen species generation by the hKGDHc as well as its E1-E2 subcomplex are implicated in neurodegenerative disorders, ischemia-reperfusion injury, E3-deficiency and cancers. 手法: We performed cryo-EM, cross-linking mass spectrometry (CL-MS) and molecular modeling analyses to determine the structure of the E2 component of the hKGDHc (hE2k); hE2k transfers a succinyl ...手法: We performed cryo-EM, cross-linking mass spectrometry (CL-MS) and molecular modeling analyses to determine the structure of the E2 component of the hKGDHc (hE2k); hE2k transfers a succinyl group to CoA and forms the structural core of hKGDHc. We also assessed the overall structure of the hKGDHc by negative-stain EM and modeling. RESULTS: We report the 2.9 Å resolution cryo-EM structure of the hE2k component. The cryo-EM map comprises density for hE2k residues 151-386 - the entire (inner) core catalytic domain plus a few ...RESULTS: We report the 2.9 Å resolution cryo-EM structure of the hE2k component. The cryo-EM map comprises density for hE2k residues 151-386 - the entire (inner) core catalytic domain plus a few additional residues -, while residues 1-150 are not observed due to the inherent flexibility of the N-terminal region. The structure of the latter segment was also determined by CL-MS and homology modeling. Negative-stain EM on in vitro assembled hKGDHc and previous data were used to build a putative overall structural model of the hKGDHc. CONCLUSIONS: The E2 core of the hKGDHc is composed of 24 hE2k chains organized in octahedral (8 × 3 type) assembly. Each lipoyl domain is oriented towards the core domain of an adjacent chain in ...CONCLUSIONS: The E2 core of the hKGDHc is composed of 24 hE2k chains organized in octahedral (8 × 3 type) assembly. Each lipoyl domain is oriented towards the core domain of an adjacent chain in the hE2k homotrimer. hE1k and hE3 are most likely tethered at the edges and faces, respectively, of the cubic hE2k assembly. GENERAL SIGNIFICANCE: The revealed structural information will support the future pharmacologically targeting of the hKGDHc. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0108.map.gz emd_0108.map.gz | 78.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0108-v30.xml emd-0108-v30.xml emd-0108.xml emd-0108.xml | 14.8 KB 14.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_0108.png emd_0108.png | 264.6 KB | ||
| Filedesc metadata |  emd-0108.cif.gz emd-0108.cif.gz | 6.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0108 http://ftp.pdbj.org/pub/emdb/structures/EMD-0108 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0108 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0108 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0108_validation.pdf.gz emd_0108_validation.pdf.gz | 524.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0108_full_validation.pdf.gz emd_0108_full_validation.pdf.gz | 524.4 KB | 表示 | |
| XML形式データ |  emd_0108_validation.xml.gz emd_0108_validation.xml.gz | 6.4 KB | 表示 | |
| CIF形式データ |  emd_0108_validation.cif.gz emd_0108_validation.cif.gz | 7.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0108 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0108 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0108 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0108 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0108.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0108.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-electron microscopic map of the human dihydrolipoamide succinyltransferase [residues 218-453] | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.061 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Affinity purified human dihydrolipoamide succinyltransferase
| 全体 | 名称: Affinity purified human dihydrolipoamide succinyltransferase |
|---|---|
| 要素 |
|
-超分子 #1: Affinity purified human dihydrolipoamide succinyltransferase
| 超分子 | 名称: Affinity purified human dihydrolipoamide succinyltransferase タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Protein in 100 mM Tris-HCl, 150 mM NaCl, 1 mM EDTA, pH 8.0. |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Dihydrolipoyllysine-residue succinyltransferase component of 2-ox...
| 分子 | 名称: Dihydrolipoyllysine-residue succinyltransferase component of 2-oxoglutarate dehydrogenase complex, mitochondrial タイプ: protein_or_peptide / ID: 1 詳細: N-terminal Twin-Strep (affinity) tag with proteolytic cleavage site and linkers: MASWSHPQFEKGGGSGGGSGGSAWSHPQFEKLEVLFQGPG Density was found for 236 residues [218-453] コピー数: 1 / 光学異性体: LEVO / EC番号: dihydrolipoyllysine-residue succinyltransferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 45.543004 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASWSHPQFE KGGGSGGGSG GSAWSHPQFE KLEVLFQGPG DDLVTVKTPA FAESVTEGDV RWEKAVGDTV AEDEVVCEIE TDKTSVQVP SPANGVIEAL LVPDGGKVEG GTPLFTLRKT GAAPAKAKPA EAPAAAAPKA EPTAAAVPPP AAPIPTQMPP V PSPSQPPS ...文字列: MASWSHPQFE KGGGSGGGSG GSAWSHPQFE KLEVLFQGPG DDLVTVKTPA FAESVTEGDV RWEKAVGDTV AEDEVVCEIE TDKTSVQVP SPANGVIEAL LVPDGGKVEG GTPLFTLRKT GAAPAKAKPA EAPAAAAPKA EPTAAAVPPP AAPIPTQMPP V PSPSQPPS GKPVSAVKPT VAPPLAEPGA GKGLRSEHRE KMNRMRQRIA QRLKEAQNTC AMLTTFNEID MSNIQEMRAR HK EAFLKKH NLKLGFMSAF VKASAFALQE QPVVNAVIDD TTKEVVYRDY IDISVAVATP RGLVVPVIRN VEAMNFADIE RTI TELGEK ARKNELAIED MDGGTFTISN GGVFGSLFGT PIINPPQSAI LGMHGIFDRP VAIGGKVEVR PMMYVALTYD HRLI DGREA VTFLRKIKAA VEDPRVLLLD L UniProtKB: Dihydrolipoyllysine-residue succinyltransferase component of 2-oxoglutarate dehydrogenase complex, mitochondrial |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV | ||||||||||||
| 詳細 | This sample was highly purified and monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 1 / 平均露光時間: 1.0 sec. / 平均電子線量: 48.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | COOT was used for structure manipulations, while MOLPROBITY was applied for validation. |
|---|---|
| 精密化 | プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-6h05: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















