+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 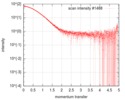 |
|---|---|
 試料 試料 | Glutamate/aspartate import solute-binding protein (DEBP), apo-form
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報aspartate binding / L-glutamate transmembrane transport / L-aspartate transmembrane transport / L-aspartate import across plasma membrane / L-glutamate import across plasma membrane / glutamate binding / amino acid transport / ATP-binding cassette (ABC) transporter complex, substrate-binding subunit-containing / outer membrane-bounded periplasmic space / extracellular region / membrane 類似検索 - 分子機能 |
| 生物種 |  |
 引用 引用 |  日付: 2019 Aug 1 日付: 2019 Aug 1タイトル: Structure-based screening of binding affinities via small-angle X-ray scattering 著者: Chen P / Masiewicz P / Perez K |
 登録者 登録者 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-モデル
- 試料
試料
 試料 試料 | 名称: Glutamate/aspartate import solute-binding protein (DEBP), apo-form 試料濃度: 2.2 mg/ml |
|---|---|
| バッファ | 名称: 100 mM NaCl, 20 mM NaPO4, 0.5 mM TCEP / pH: 7.4 / コメント: SEC buffer for PBP experiments |
| 要素 #1755 | タイプ: protein / 記述: Glutamate/aspartate import solute-binding protein / 分子量: 32.052 / 分子数: 1 / 由来: Escherichia coli (strain K12) / 参照: UniProt: P37902 配列: HHHHHHDDAA PAAGSTLDKI AKNGVIVVGH RESSVPFSYY DNQQKVVGYS QDYSNAIVEA VKKKLNKPDL QVKLIPITSQ NRIPLLQNGT FDFECGSTTN NVERQKQAAF SDTIFVVGTR LLTKKGGDIK DFANLKDKAV VVTSGTTSEV LLNKLNEEQK MNMRIISAKD ...配列: HHHHHHDDAA PAAGSTLDKI AKNGVIVVGH RESSVPFSYY DNQQKVVGYS QDYSNAIVEA VKKKLNKPDL QVKLIPITSQ NRIPLLQNGT FDFECGSTTN NVERQKQAAF SDTIFVVGTR LLTKKGGDIK DFANLKDKAV VVTSGTTSEV LLNKLNEEQK MNMRIISAKD HGDSFRTLES GRAVAFMMDD ALLAGERAKA KKPDNWEIVG KPQSQEAYGC MLRKDDPQFK KLMDDTIAQV QTSGEAEKWF DKWFKNPIPP KNLNMNFELS DEMKALFKEP NDKALN |
-実験情報
| ビーム | 設備名称: ESRF BM29 / 地域: Grenoble / 国: France  / 線源: X-ray synchrotron / 波長: 0.09919 Å / スペクトロメータ・検出器間距離: 2.849 mm / 線源: X-ray synchrotron / 波長: 0.09919 Å / スペクトロメータ・検出器間距離: 2.849 mm | |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 1M / タイプ: Dectris / Pixsize x: 172 mm | |||||||||||||||||||||||||||||||||
| スキャン | 測定日: 2018年9月10日 / セル温度: 20 °C / 照射時間: 2 sec. / フレーム数: 12 / 単位: 1/nm /
| |||||||||||||||||||||||||||||||||
| 距離分布関数 P(R) |
| |||||||||||||||||||||||||||||||||
| 結果 | コメント: Free hexahistidine-tagged DEBP after His-Trap and SEC column purification. Sequence assumed based on Clifton et al. 2016, which reports a plasmid containing a direct attachment of N-terminal His tag.
|
 ムービー
ムービー コントローラー
コントローラー



 SASDFH8
SASDFH8




