+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8b9c | ||||||
|---|---|---|---|---|---|---|---|
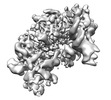
| タイトル | S. cerevisiae pol alpha - replisome complex | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | REPLICATION / helicase / polymerase / pol alpha / priming / replisome | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Inhibition of replication initiation of damaged DNA by RB1/E2F1 / H3-H4 histone complex chaperone activity / maintenance of DNA repeat elements / Unwinding of DNA / replication fork arrest / regulation of nuclear cell cycle DNA replication / DNA replication initiation / meiotic chromosome segregation / RNA-templated DNA biosynthetic process / MCM core complex ...Inhibition of replication initiation of damaged DNA by RB1/E2F1 / H3-H4 histone complex chaperone activity / maintenance of DNA repeat elements / Unwinding of DNA / replication fork arrest / regulation of nuclear cell cycle DNA replication / DNA replication initiation / meiotic chromosome segregation / RNA-templated DNA biosynthetic process / MCM core complex / Assembly of the pre-replicative complex / Switching of origins to a post-replicative state / DNA strand elongation involved in mitotic DNA replication / MCM complex binding / Processive synthesis on the lagging strand / GINS complex / nuclear DNA replication / mitotic DNA replication preinitiation complex assembly / Removal of the Flap Intermediate / premeiotic DNA replication / Polymerase switching / pre-replicative complex assembly involved in nuclear cell cycle DNA replication / telomere capping / anaphase-promoting complex binding / mitotic DNA replication / alpha DNA polymerase:primase complex / Activation of the pre-replicative complex / CMG complex / nuclear pre-replicative complex / DNA replication checkpoint signaling / DNA primase activity / establishment of mitotic sister chromatid cohesion / Activation of ATR in response to replication stress / DNA replication preinitiation complex / MCM complex / replication fork protection complex / primosome complex / DNA replication, synthesis of primer / mitotic DNA replication checkpoint signaling / lagging strand elongation / double-strand break repair via break-induced replication / cellular response to osmotic stress / mitotic DNA replication initiation / single-stranded DNA helicase activity / regulation of DNA-templated DNA replication initiation / mitotic intra-S DNA damage checkpoint signaling / DNA strand elongation involved in DNA replication / silent mating-type cassette heterochromatin formation / mitotic sister chromatid cohesion / DNA synthesis involved in DNA repair / DNA biosynthetic process / leading strand elongation / DNA unwinding involved in DNA replication / replication fork processing / nuclear replication fork / mitotic G2 DNA damage checkpoint signaling / DNA replication origin binding / DNA replication initiation / subtelomeric heterochromatin formation / Ub-specific processing proteases / DNA helicase activity / telomere maintenance / nuclear periphery / replication fork / meiotic cell cycle / helicase activity / transcription elongation by RNA polymerase II / heterochromatin formation / DNA-templated DNA replication / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / double-strand break repair / nuclear envelope / single-stranded DNA binding / 4 iron, 4 sulfur cluster binding / DNA helicase / DNA replication / DNA-directed DNA polymerase / chromosome, telomeric region / DNA-directed DNA polymerase activity / protein stabilization / DNA repair / nucleotide binding / DNA damage response / chromatin binding / ATP hydrolysis activity / mitochondrion / DNA binding / nucleoplasm / ATP binding / nucleus / metal ion binding / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.6 Å | ||||||
 データ登録者 データ登録者 | Jones, M.L. / Yeeles, J.T.P. | ||||||
| 資金援助 |  英国, 1件 英国, 1件
| ||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2023 ジャーナル: Mol Cell / 年: 2023タイトル: How Pol α-primase is targeted to replisomes to prime eukaryotic DNA replication. 著者: Morgan L Jones / Valentina Aria / Yasemin Baris / Joseph T P Yeeles /  要旨: During eukaryotic DNA replication, Pol α-primase generates primers at replication origins to start leading-strand synthesis and every few hundred nucleotides during discontinuous lagging-strand ...During eukaryotic DNA replication, Pol α-primase generates primers at replication origins to start leading-strand synthesis and every few hundred nucleotides during discontinuous lagging-strand replication. How Pol α-primase is targeted to replication forks to prime DNA synthesis is not fully understood. Here, by determining cryoelectron microscopy (cryo-EM) structures of budding yeast and human replisomes containing Pol α-primase, we reveal a conserved mechanism for the coordination of priming by the replisome. Pol α-primase binds directly to the leading edge of the CMG (CDC45-MCM-GINS) replicative helicase via a complex interaction network. The non-catalytic PRIM2/Pri2 subunit forms two interfaces with CMG that are critical for in vitro DNA replication and yeast cell growth. These interactions position the primase catalytic subunit PRIM1/Pri1 directly above the exit channel for lagging-strand template single-stranded DNA (ssDNA), revealing why priming occurs efficiently only on the lagging-strand template and elucidating a mechanism for Pol α-primase to overcome competition from RPA to initiate primer synthesis. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8b9c.cif.gz 8b9c.cif.gz | 2.5 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8b9c.ent.gz pdb8b9c.ent.gz | 表示 |  PDB形式 PDB形式 | |
| PDBx/mmJSON形式 |  8b9c.json.gz 8b9c.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8b9c_validation.pdf.gz 8b9c_validation.pdf.gz | 1.5 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8b9c_full_validation.pdf.gz 8b9c_full_validation.pdf.gz | 1.5 MB | 表示 | |
| XML形式データ |  8b9c_validation.xml.gz 8b9c_validation.xml.gz | 163.5 KB | 表示 | |
| CIF形式データ |  8b9c_validation.cif.gz 8b9c_validation.cif.gz | 261.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/b9/8b9c https://data.pdbj.org/pub/pdb/validation_reports/b9/8b9c ftp://data.pdbj.org/pub/pdb/validation_reports/b9/8b9c ftp://data.pdbj.org/pub/pdb/validation_reports/b9/8b9c | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-DNA replication licensing factor ... , 5種, 5分子 23467
| #1: タンパク質 | 分子量: 98911.539 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: MCM2, YBL023C, YBL0438 / 発現宿主:  |
|---|---|
| #2: タンパク質 | 分子量: 111987.562 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: MCM3, YEL032W, SYGP-ORF23 / 発現宿主:  |
| #3: タンパク質 | 分子量: 105138.375 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: MCM4, CDC54, HCD21, YPR019W, YP9531.13 / 発現宿主:  |
| #5: タンパク質 | 分子量: 113110.211 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: MCM6, YGL201C / 発現宿主:  |
| #6: タンパク質 | 分子量: 95049.875 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: MCM7, CDC47, YBR202W, YBR1441 / 発現宿主:  |
-タンパク質 , 7種, 7分子 5AGPSXY
| #4: タンパク質 | 分子量: 86505.734 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: MCM5, CDC46, YLR274W, L9328.1 / 発現宿主:  |
|---|---|
| #7: タンパク質 | 分子量: 62348.551 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: PRI2, YKL045W, YKL258 / 発現宿主:  |
| #13: タンパク質 | 分子量: 75154.703 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: CDC45, SLD4, YLR103C, L8004.11 / 発現宿主:  |
| #14: タンパク質 | 分子量: 126078.859 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: MRC1, YCL061C, YCL61C/YCL60C / 発現宿主:  |
| #17: タンパク質 | 分子量: 51827.090 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: PRI1, YIR008C, YIB8C / 発現宿主:  参照: UniProt: P10363, 転移酵素; リンを含む基を移すもの; 核酸を移すもの |
| #18: タンパク質 | 分子量: 141296.875 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: TOF1, YNL273W, N0636 / 発現宿主:  |
| #19: タンパク質 | 分子量: 36588.758 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: CSM3, YMR048W, YM9796.01 / 発現宿主:  |
-DNA polymerase alpha ... , 2種, 2分子 BJ
| #8: タンパク質 | 分子量: 78865.938 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: POL12, YBL035C, YBL0414 / 発現宿主:  |
|---|---|
| #20: タンパク質 | 分子量: 167027.766 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: POL1, CDC17, YNL102W, N2181 / 発現宿主:  |
-DNA replication complex GINS protein ... , 4種, 4分子 CDEF
| #9: タンパク質 | 分子量: 24230.576 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: PSF1, YDR013W, PZA208, YD8119.18 / 発現宿主:  |
|---|---|
| #10: タンパク質 | 分子量: 25096.807 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: PSF2, YJL072C, HRF213, J1086 / 発現宿主:  |
| #11: タンパク質 | 分子量: 24437.859 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: PSF3, YOL146W / 発現宿主:  |
| #12: タンパク質 | 分子量: 33983.617 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: SLD5, YDR489W / 発現宿主:  |
-DNA鎖 , 2種, 2分子 QR
| #15: DNA鎖 | 分子量: 26092.645 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)  |
|---|---|
| #16: DNA鎖 | 分子量: 32046.451 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)  |
-非ポリマー , 3種, 12分子 




| #21: 化合物 | ChemComp-ANP / #22: 化合物 | ChemComp-MG / #23: 化合物 | ChemComp-ZN / |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: S. cerevisiae replisome - pol alpha primase complex / タイプ: COMPLEX / Entity ID: #1-#20 / 由来: RECOMBINANT |
|---|---|
| 由来(天然) | 生物種:  |
| 由来(組換発現) | 生物種:  |
| 緩衝液 | pH: 7.6 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3500 nm / 最小 デフォーカス(公称値): 1500 nm |
| 撮影 | 電子線照射量: 39.2 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
| EMソフトウェア | 名称: PHENIX / バージョン: 1.20_4459: / カテゴリ: モデル精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 4.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 53964 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー




























 PDBj
PDBj






















































