+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7zrl | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
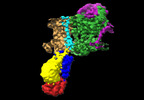
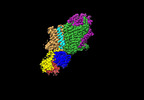
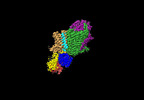
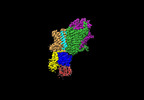
| タイトル | Cryo-EM map of the unphosphorylated KdpFABC complex in the E2-P conformation, under turnover conditions | ||||||||||||||||||
 要素 要素 |
| ||||||||||||||||||
 キーワード キーワード | MEMBRANE PROTEIN / P-type ATPase / superfamily of K+ transporters (SKT) / potassium uptake system | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報P-type K+ transporter / P-type potassium transmembrane transporter activity / potassium:proton antiporter complex / potassium ion-transporting ATPase complex / monoatomic cation transmembrane transport / potassium ion binding / potassium ion transmembrane transport / potassium ion transport / magnesium ion binding / ATP hydrolysis activity ...P-type K+ transporter / P-type potassium transmembrane transporter activity / potassium:proton antiporter complex / potassium ion-transporting ATPase complex / monoatomic cation transmembrane transport / potassium ion binding / potassium ion transmembrane transport / potassium ion transport / magnesium ion binding / ATP hydrolysis activity / ATP binding / plasma membrane 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  | ||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Hielkema, L. / Stock, C. / Silberberg, J.M. / Corey, R.A. / Wunnicke, D. / Dubach, V.R.A. / Stansfeld, P.J. / Haenelt, I. / Paulino, C. | ||||||||||||||||||
| 資金援助 |  英国, 5件 英国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2022 ジャーナル: Elife / 年: 2022タイトル: Inhibited KdpFABC transitions into an E1 off-cycle state. 著者: Jakob M Silberberg / Charlott Stock / Lisa Hielkema / Robin A Corey / Jan Rheinberger / Dorith Wunnicke / Victor R A Dubach / Phillip J Stansfeld / Inga Hänelt / Cristina Paulino /    要旨: KdpFABC is a high-affinity prokaryotic K uptake system that forms a functional chimera between a channel-like subunit (KdpA) and a P-type ATPase (KdpB). At high K levels, KdpFABC needs to be ...KdpFABC is a high-affinity prokaryotic K uptake system that forms a functional chimera between a channel-like subunit (KdpA) and a P-type ATPase (KdpB). At high K levels, KdpFABC needs to be inhibited to prevent excessive K accumulation to the point of toxicity. This is achieved by a phosphorylation of the serine residue in the TGES motif in the A domain of the pump subunit KdpB (KdpB). Here, we explore the structural basis of inhibition by KdpB phosphorylation by determining the conformational landscape of KdpFABC under inhibiting and non-inhibiting conditions. Under turnover conditions, we identified a new inhibited KdpFABC state that we termed E1P tight, which is not part of the canonical Post-Albers transport cycle of P-type ATPases. It likely represents the biochemically described stalled E1P state adopted by KdpFABC upon KdpB phosphorylation. The E1P tight state exhibits a compact fold of the three cytoplasmic domains and is likely adopted when the transition from high-energy E1P states to E2P states is unsuccessful. This study represents a structural characterization of a biologically relevant off-cycle state in the P-type ATPase family and supports the emerging discussion of P-type ATPase regulation by such states. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7zrl.cif.gz 7zrl.cif.gz | 244 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7zrl.ent.gz pdb7zrl.ent.gz | 196.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7zrl.json.gz 7zrl.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7zrl_validation.pdf.gz 7zrl_validation.pdf.gz | 1.1 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7zrl_full_validation.pdf.gz 7zrl_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  7zrl_validation.xml.gz 7zrl_validation.xml.gz | 49.9 KB | 表示 | |
| CIF形式データ |  7zrl_validation.cif.gz 7zrl_validation.cif.gz | 76.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/zr/7zrl https://data.pdbj.org/pub/pdb/validation_reports/zr/7zrl ftp://data.pdbj.org/pub/pdb/validation_reports/zr/7zrl ftp://data.pdbj.org/pub/pdb/validation_reports/zr/7zrl | HTTPS FTP |
-関連構造データ
| 関連構造データ |  14918MC  7zrdC  7zreC  7zrgC  7zrhC  7zriC  7zrjC  7zrkC  7zrmC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 59218.613 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   |
|---|---|
| #2: タンパク質 | 分子量: 72331.844 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   |
| #3: タンパク質 | 分子量: 20281.035 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   |
| #4: タンパク質・ペプチド | 分子量: 2853.463 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   |
| #5: 化合物 | ChemComp-K / |
| 研究の焦点であるリガンドがあるか | Y |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: KdpFABC / タイプ: COMPLEX / Entity ID: #1-#4 / 由来: RECOMBINANT |
|---|---|
| 分子量 | 値: 0.157 MDa / 実験値: NO |
| 由来(天然) | 生物種:  |
| 由来(組換発現) | 生物種:  |
| 緩衝液 | pH: 8 詳細: 10 mM Tris-HCl pH 8, 10 mM MgCl2, 10 mM NaCl and 0.0125% DDM |
| 試料 | 濃度: 3.4 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | 詳細: at 5 mA / グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 |
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE-PROPANE / 湿度: 100 % / 凍結前の試料温度: 293 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TALOS ARCTICA |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 49407 X / 倍率(補正後): 49407 X / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 500 nm / Calibrated defocus min: 500 nm / 最大 デフォーカス(補正後): 2000 nm / Cs: 2.7 mm / C2レンズ絞り径: 100 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER 最高温度: 105 K / 最低温度: 90 K |
| 撮影 | 平均露光時間: 9 sec. / 電子線照射量: 52 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 3 / 実像数: 11428 |
| 電子光学装置 | エネルギーフィルター名称: GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV |
| 画像スキャン | 横: 3838 / 縦: 3710 / 動画フレーム数/画像: 60 / 利用したフレーム数/画像: 1-60 |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.19.2_4158: / 分類: 精密化 | ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 287232 | ||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 47004 / アルゴリズム: BACK PROJECTION / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||
| 原子モデル構築 | 空間: REAL | ||||||||||||||||||||||||||||||||
| 原子モデル構築 | PDB-ID: 6HRB | ||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj





