[English] 日本語
 Yorodumi
Yorodumi- PDB-6peq: GluA2 in complex with its auxiliary subunit CNIH3 - map LBD-TMD-C... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6peq | ||||||
|---|---|---|---|---|---|---|---|
| Title | GluA2 in complex with its auxiliary subunit CNIH3 - map LBD-TMD-C3 - with antagonist ZK200775 -without NTD | ||||||
 Components Components |
| ||||||
 Keywords Keywords | TRANSPORT PROTEIN / ionotropic glutamate receptor / AMPA receptor / cornichon / auxiliary subunit / ion channel / ligand gated ion channel / synaptic transmission / excitatory synaptic transmission / neurotransmitter receptor / stargazin / TARP / ZK200775 / lipid / MPQX | ||||||
| Function / homology |  Function and homology information Function and homology informationCargo concentration in the ER / COPII-mediated vesicle transport / localization within membrane / channel regulator activity / regulation of AMPA receptor activity / spine synapse / dendritic spine cytoplasm / dendritic spine neck / cellular response to amine stimulus / dendritic spine head ...Cargo concentration in the ER / COPII-mediated vesicle transport / localization within membrane / channel regulator activity / regulation of AMPA receptor activity / spine synapse / dendritic spine cytoplasm / dendritic spine neck / cellular response to amine stimulus / dendritic spine head / neurotransmitter receptor localization to postsynaptic specialization membrane / Activation of AMPA receptors / ligand-gated monoatomic cation channel activity / perisynaptic space / AMPA glutamate receptor activity / Trafficking of GluR2-containing AMPA receptors / response to lithium ion / AMPA glutamate receptor clustering / cellular response to glycine / kainate selective glutamate receptor activity / immunoglobulin binding / asymmetric synapse / AMPA glutamate receptor complex / regulation of receptor recycling / extracellularly glutamate-gated ion channel activity / ionotropic glutamate receptor complex / conditioned place preference / Unblocking of NMDA receptors, glutamate binding and activation / glutamate receptor binding / positive regulation of synaptic transmission / regulation of synaptic transmission, glutamatergic / response to fungicide / cytoskeletal protein binding / vesicle-mediated transport / extracellular ligand-gated monoatomic ion channel activity / glutamate-gated receptor activity / cellular response to brain-derived neurotrophic factor stimulus / regulation of long-term synaptic depression / somatodendritic compartment / glutamate-gated calcium ion channel activity / presynaptic active zone membrane / dendrite membrane / ionotropic glutamate receptor binding / excitatory synapse / ionotropic glutamate receptor signaling pathway / dendrite cytoplasm / ligand-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / synaptic membrane / positive regulation of excitatory postsynaptic potential / dendritic shaft / SNARE binding / PDZ domain binding / regulation of membrane potential / protein tetramerization / synaptic transmission, glutamatergic / establishment of protein localization / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / cerebral cortex development / receptor internalization / postsynaptic density membrane / modulation of chemical synaptic transmission / Schaffer collateral - CA1 synapse / terminal bouton / synaptic vesicle / long-term synaptic potentiation / synaptic vesicle membrane / signaling receptor activity / amyloid-beta binding / presynapse / growth cone / presynaptic membrane / scaffold protein binding / dendritic spine / chemical synaptic transmission / perikaryon / postsynaptic membrane / neuron projection / postsynaptic density / external side of plasma membrane / axon / neuronal cell body / dendrite / synapse / protein kinase binding / protein-containing complex binding / glutamatergic synapse / cell surface / endoplasmic reticulum / protein-containing complex / identical protein binding / membrane / plasma membrane Similarity search - Function | ||||||
| Biological species |   | ||||||
| Method | ELECTRON MICROSCOPY / single particle reconstruction / cryo EM / Resolution: 2.97 Å | ||||||
 Authors Authors | Nakagawa, T. | ||||||
| Funding support |  United States, 1items United States, 1items
| ||||||
 Citation Citation |  Journal: Science / Year: 2019 Journal: Science / Year: 2019Title: Structures of the AMPA receptor in complex with its auxiliary subunit cornichon. Authors: Terunaga Nakagawa /  Abstract: In the brain, AMPA-type glutamate receptors (AMPARs) form complexes with their auxiliary subunits and mediate the majority of fast excitatory neurotransmission. Signals transduced by these complexes ...In the brain, AMPA-type glutamate receptors (AMPARs) form complexes with their auxiliary subunits and mediate the majority of fast excitatory neurotransmission. Signals transduced by these complexes are critical for synaptic plasticity, learning, and memory. The two major categories of AMPAR auxiliary subunits are transmembrane AMPAR regulatory proteins (TARPs) and cornichon homologs (CNIHs); these subunits share little homology and play distinct roles in controlling ion channel gating and trafficking of AMPAR. Here, I report high-resolution cryo-electron microscopy structures of AMPAR in complex with CNIH3. Contrary to its predicted membrane topology, CNIH3 lacks an extracellular domain and instead contains four membrane-spanning helices. The protein-protein interaction interface that dictates channel modulation and the lipids surrounding the complex are revealed. These structures provide insights into the molecular mechanism for ion channel modulation and assembly of AMPAR/CNIH3 complexes. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6peq.cif.gz 6peq.cif.gz | 714.5 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6peq.ent.gz pdb6peq.ent.gz | 578.2 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6peq.json.gz 6peq.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/pe/6peq https://data.pdbj.org/pub/pdb/validation_reports/pe/6peq ftp://data.pdbj.org/pub/pdb/validation_reports/pe/6peq ftp://data.pdbj.org/pub/pdb/validation_reports/pe/6peq | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  20330MC  6u5sC  6u6iC  6ucbC  6ud4C  6ud8C M: map data used to model this data C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
|
|---|---|
| 1 |
|
- Components
Components
-Protein , 2 types, 8 molecules ABCDEFGH
| #1: Protein | Mass: 99530.391 Da / Num. of mol.: 4 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   Homo sapiens (human) / References: UniProt: P19491 Homo sapiens (human) / References: UniProt: P19491#2: Protein | Mass: 20262.758 Da / Num. of mol.: 4 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   Homo sapiens (human) / References: UniProt: Q6ZWS4 Homo sapiens (human) / References: UniProt: Q6ZWS4 |
|---|
-Non-polymers , 4 types, 30 molecules 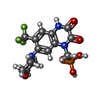

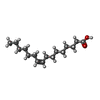




| #3: Chemical | ChemComp-ZK1 / {[ #4: Chemical | ChemComp-OLC / ( #5: Chemical | ChemComp-PAM / #6: Chemical | ChemComp-CLR / |
|---|
-Details
| Has ligand of interest | Y |
|---|---|
| Has protein modification | Y |
-Experimental details
-Experiment
| Experiment | Method: ELECTRON MICROSCOPY |
|---|---|
| EM experiment | Aggregation state: PARTICLE / 3D reconstruction method: single particle reconstruction |
- Sample preparation
Sample preparation
| Component |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Molecular weight | Value: 0.47 MDa / Experimental value: NO | ||||||||||||||||||||||||||||
| Source (natural) |
| ||||||||||||||||||||||||||||
| Source (recombinant) |
| ||||||||||||||||||||||||||||
| Buffer solution | pH: 8 | ||||||||||||||||||||||||||||
| Buffer component |
| ||||||||||||||||||||||||||||
| Specimen | Embedding applied: NO / Shadowing applied: NO / Staining applied: NO / Vitrification applied: YES | ||||||||||||||||||||||||||||
| Specimen support | Grid material: COPPER / Grid mesh size: 300 divisions/in. / Grid type: Quantifoil R1.2/1.3 | ||||||||||||||||||||||||||||
| Vitrification | Instrument: FEI VITROBOT MARK IV / Cryogen name: ETHANE / Humidity: 100 % / Chamber temperature: 277.15 K |
- Electron microscopy imaging
Electron microscopy imaging
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
|---|---|
| Microscopy | Model: FEI TITAN KRIOS |
| Electron gun | Electron source:  FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM |
| Electron lens | Mode: BRIGHT FIELD / Nominal magnification: 81000 X / Nominal defocus max: 2000 nm / Nominal defocus min: 800 nm / Cs: 2.7 mm |
| Specimen holder | Cryogen: NITROGEN / Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER |
| Image recording | Average exposure time: 6 sec. / Electron dose: 58.5 e/Å2 / Film or detector model: GATAN K3 BIOQUANTUM (6k x 4k) / Num. of grids imaged: 1 / Num. of real images: 11340 |
| EM imaging optics | Energyfilter name: GIF Bioquantum / Energyfilter slit width: 20 eV |
- Processing
Processing
| EM software |
| ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF correction | Type: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||
| Particle selection | Num. of particles selected: 6780000 | ||||||||||||||||||||||||||||||||||||
| Symmetry | Point symmetry: C2 (2 fold cyclic) | ||||||||||||||||||||||||||||||||||||
| 3D reconstruction | Resolution: 2.97 Å / Resolution method: FSC 0.143 CUT-OFF / Num. of particles: 188982 / Symmetry type: POINT | ||||||||||||||||||||||||||||||||||||
| Atomic model building | Space: REAL |
 Movie
Movie Controller
Controller

















 PDBj
PDBj















