+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8314 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Architecture of a headful DNA-packaging bacterial virus at 2.89 Angstrom resolution by electron cryo-microscopy | |||||||||
 マップデータ マップデータ | bacteriophage Sf6 icosahedral reconstruction | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Major capsid protein Gp5 / P22 coat protein - gene protein 5 / Gene 5 protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Shigella phage Sf6 (ファージ) Shigella phage Sf6 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.89 Å | |||||||||
 データ登録者 データ登録者 | Zhao H / Li J / Jiang W / Tang L | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2017 ジャーナル: Proc Natl Acad Sci U S A / 年: 2017タイトル: Structure of a headful DNA-packaging bacterial virus at 2.9 Å resolution by electron cryo-microscopy. 著者: Haiyan Zhao / Kunpeng Li / Anna Y Lynn / Keith E Aron / Guimei Yu / Wen Jiang / Liang Tang /  要旨: The enormous prevalence of tailed DNA bacteriophages on this planet is enabled by highly efficient self-assembly of hundreds of protein subunits into highly stable capsids. These capsids can stand ...The enormous prevalence of tailed DNA bacteriophages on this planet is enabled by highly efficient self-assembly of hundreds of protein subunits into highly stable capsids. These capsids can stand with an internal pressure as high as ∼50 atmospheres as a result of the phage DNA-packaging process. Here we report the complete atomic model of the headful DNA-packaging bacteriophage Sf6 at 2.9 Å resolution determined by electron cryo-microscopy. The structure reveals the DNA-inflated, tensed state of a robust protein shell assembled via noncovalent interactions. Remarkable global conformational polymorphism of capsid proteins, a network formed by extended N arms, mortise-and-tenon-like intercapsomer joints, and abundant β-sheet-like mainchain:mainchain intermolecular interactions, confers significant strength yet also flexibility required for capsid assembly and DNA packaging. Differential formations of the hexon and penton are mediated by a drastic α-helix-to-β-strand structural transition. The assembly scheme revealed here may be common among tailed DNA phages and herpesviruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8314.map.gz emd_8314.map.gz | 858.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8314-v30.xml emd-8314-v30.xml emd-8314.xml emd-8314.xml | 12.2 KB 12.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
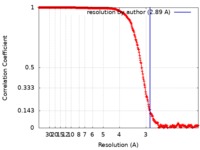
| FSC (解像度算出) |  emd_8314_fsc.xml emd_8314_fsc.xml | 32.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8314.png emd_8314.png | 104.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8314 http://ftp.pdbj.org/pub/emdb/structures/EMD-8314 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8314 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8314 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8314_validation.pdf.gz emd_8314_validation.pdf.gz | 407.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8314_full_validation.pdf.gz emd_8314_full_validation.pdf.gz | 407.3 KB | 表示 | |
| XML形式データ |  emd_8314_validation.xml.gz emd_8314_validation.xml.gz | 22.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8314 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8314 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8314 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8314 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8314.map.gz / 形式: CCP4 / 大きさ: 1.9 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8314.map.gz / 形式: CCP4 / 大きさ: 1.9 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | bacteriophage Sf6 icosahedral reconstruction | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.01 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Shigella phage Sf6
| 全体 | 名称:  Shigella phage Sf6 (ファージ) Shigella phage Sf6 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Shigella phage Sf6
| 超分子 | 名称: Shigella phage Sf6 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 10761 / 生物種: Shigella phage Sf6 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Shigella flexneri (フレクスナー赤痢菌) / 株: M94 Shigella flexneri (フレクスナー赤痢菌) / 株: M94 |
| 分子量 | 理論値: 19.5 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: virus capsid / 直径: 650.0 Å / T番号(三角分割数): 7 |
-分子 #1: bacteriophage sf6 capsid protein gp5
| 分子 | 名称: bacteriophage sf6 capsid protein gp5 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: DEXTRO |
|---|---|
| 由来(天然) | 生物種:  Shigella phage Sf6 (ファージ) Shigella phage Sf6 (ファージ) |
| 配列 | 文字列: MPNNLDSNVS QIVLKKFLPG FMSDLVLAKT VDRQLLAGEI NSSTGDSVSF KRPHQFSSLR TPTGDISGQ NKNNLISGKA TGRVGNYITV AVEYQQLEEA IKLNQLEEIL APVRQRIVTD L ETELAHFM MNNGALSLGS PNTPITKWSD VAQTASFLKD LGVNEGENYA ...文字列: MPNNLDSNVS QIVLKKFLPG FMSDLVLAKT VDRQLLAGEI NSSTGDSVSF KRPHQFSSLR TPTGDISGQ NKNNLISGKA TGRVGNYITV AVEYQQLEEA IKLNQLEEIL APVRQRIVTD L ETELAHFM MNNGALSLGS PNTPITKWSD VAQTASFLKD LGVNEGENYA VMDPWSAQRL AD AQTGLHA SDQLVRTAWE NAQIPTNFGG IRALMSNGLA SRTQGAFGGT LTVKTQPTVT YNA VKDSYQ FTVTLTGATA SVTGFLKAGD QVKFTNTYWL QQQTKQALYN GATPISFTAT VTAD ANSDS GGDVTVTLSG VPIYDTTNPQ YNSVSRQVEA GDAVSVVGTA SQTMKPNLFY NKFFC GLGS IPLPKLHSID SAVATYEGFS IRVHKYADGD ANVQKMRFDL LPAYVCFNPH MGGQFF GNP |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 15 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||
| グリッド | 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV | |||||||||
| 詳細 | purified Sf6 phage |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 9.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: AB INITIO MODEL / 温度因子: 46.6 当てはまり具合の基準: Pseudo-crystallographic R factor and stereochemistry |
|---|---|
| 得られたモデル |  PDB-5l35: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)