+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-4973 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM structure of the partially activated Drs2p-Cdc50p | |||||||||||||||||||||
 Map data Map data | manual b-factor sharpening -60 | |||||||||||||||||||||
 Sample Sample |
| |||||||||||||||||||||
 Keywords Keywords | Lipid Flippase / Lipid Transport / P-type ATPase / PS Transport. | |||||||||||||||||||||
| Function / homology |  Function and homology information Function and homology informationCdc50p-Drs2p complex / actin cortical patch localization / aminophospholipid translocation / phosphatidylcholine flippase activity / Ion transport by P-type ATPases / post-Golgi vesicle-mediated transport / phosphatidylserine flippase activity / phospholipid-translocating ATPase complex / phosphatidylserine floppase activity / ATPase-coupled intramembrane lipid transporter activity ...Cdc50p-Drs2p complex / actin cortical patch localization / aminophospholipid translocation / phosphatidylcholine flippase activity / Ion transport by P-type ATPases / post-Golgi vesicle-mediated transport / phosphatidylserine flippase activity / phospholipid-translocating ATPase complex / phosphatidylserine floppase activity / ATPase-coupled intramembrane lipid transporter activity / phosphatidylethanolamine flippase activity / endocytic recycling / P-type phospholipid transporter / phosphatidylinositol-4-phosphate binding / retrograde transport, endosome to Golgi / phospholipid translocation / Neutrophil degranulation / intracellular protein transport / trans-Golgi network / endocytosis / late endosome membrane / endosome membrane / magnesium ion binding / endoplasmic reticulum / Golgi apparatus / ATP hydrolysis activity / ATP binding / plasma membrane / cytosol Similarity search - Function | |||||||||||||||||||||
| Biological species |   | |||||||||||||||||||||
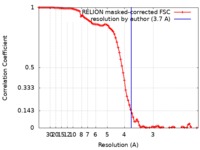
| Method | single particle reconstruction / cryo EM / Resolution: 3.7 Å | |||||||||||||||||||||
 Authors Authors | Timcenko M / Lyons JA | |||||||||||||||||||||
| Funding support |  Denmark, Denmark,  France, 6 items France, 6 items
| |||||||||||||||||||||
 Citation Citation |  Journal: Nature / Year: 2019 Journal: Nature / Year: 2019Title: Structure and autoregulation of a P4-ATPase lipid flippase. Authors: Milena Timcenko / Joseph A Lyons / Dovile Januliene / Jakob J Ulstrup / Thibaud Dieudonné / Cédric Montigny / Miriam-Rose Ash / Jesper Lykkegaard Karlsen / Thomas Boesen / Werner ...Authors: Milena Timcenko / Joseph A Lyons / Dovile Januliene / Jakob J Ulstrup / Thibaud Dieudonné / Cédric Montigny / Miriam-Rose Ash / Jesper Lykkegaard Karlsen / Thomas Boesen / Werner Kühlbrandt / Guillaume Lenoir / Arne Moeller / Poul Nissen /    Abstract: Type 4 P-type ATPases (P4-ATPases) are lipid flippases that drive the active transport of phospholipids from exoplasmic or luminal leaflets to cytosolic leaflets of eukaryotic membranes. The ...Type 4 P-type ATPases (P4-ATPases) are lipid flippases that drive the active transport of phospholipids from exoplasmic or luminal leaflets to cytosolic leaflets of eukaryotic membranes. The molecular architecture of P4-ATPases and the mechanism through which they recognize and transport lipids have remained unknown. Here we describe the cryo-electron microscopy structure of the P4-ATPase Drs2p-Cdc50p, a Saccharomyces cerevisiae lipid flippase that is specific to phosphatidylserine and phosphatidylethanolamine. Drs2p-Cdc50p is autoinhibited by the C-terminal tail of Drs2p, and activated by the lipid phosphatidylinositol-4-phosphate (PtdIns4P or PI4P). We present three structures that represent the complex in an autoinhibited, an intermediate and a fully activated state. The analysis highlights specific features of P4-ATPases and reveals sites of autoinhibition and PI4P-dependent activation. We also observe a putative lipid translocation pathway in this flippase that involves a conserved PISL motif in transmembrane segment 4 and polar residues of transmembrane segments 2 and 5, in particular Lys1018, in the centre of the lipid bilayer. | |||||||||||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_4973.map.gz emd_4973.map.gz | 5.2 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-4973-v30.xml emd-4973-v30.xml emd-4973.xml emd-4973.xml | 33.8 KB 33.8 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_4973_fsc.xml emd_4973_fsc.xml | 9.2 KB | Display |  FSC data file FSC data file |
| Images |  emd_4973.png emd_4973.png | 124.1 KB | ||
| Masks |  emd_4973_msk_1.map emd_4973_msk_1.map | 64 MB |  Mask map Mask map | |
| Filedesc metadata |  emd-4973.cif.gz emd-4973.cif.gz | 9.6 KB | ||
| Others |  emd_4973_additional.map.gz emd_4973_additional.map.gz emd_4973_half_map_1.map.gz emd_4973_half_map_1.map.gz emd_4973_half_map_2.map.gz emd_4973_half_map_2.map.gz | 3.1 MB 49.8 MB 49.7 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4973 http://ftp.pdbj.org/pub/emdb/structures/EMD-4973 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4973 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4973 | HTTPS FTP |
-Related structure data
| Related structure data |  6roiMC  4972C  4974C  6rohC  6rojC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_4973.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_4973.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | manual b-factor sharpening -60 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.077 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
-Mask #1
| File |  emd_4973_msk_1.map emd_4973_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
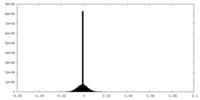
| Density Histograms |
-Additional map: #1
| File | emd_4973_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
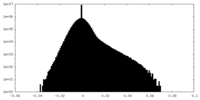
| Density Histograms |
-Half map: #2
| File | emd_4973_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
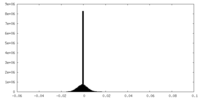
| Density Histograms |
-Half map: #1
| File | emd_4973_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
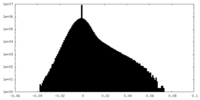
| Density Histograms |
- Sample components
Sample components
-Entire : Binary complex of Drs2p-Cdc50p with the regulatory lipid PI4P
| Entire | Name: Binary complex of Drs2p-Cdc50p with the regulatory lipid PI4P |
|---|---|
| Components |
|
-Supramolecule #1: Binary complex of Drs2p-Cdc50p with the regulatory lipid PI4P
| Supramolecule | Name: Binary complex of Drs2p-Cdc50p with the regulatory lipid PI4P type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1-#2 |
|---|---|
| Source (natural) | Organism:  |
-Macromolecule #1: Probable phospholipid-transporting ATPase DRS2
| Macromolecule | Name: Probable phospholipid-transporting ATPase DRS2 / type: protein_or_peptide / ID: 1 Details: D560 described by Aspartate beryllium trifluoride (BFD) engineered C-terminal GGGG-LVPRGS-BAD-tag Residues 1-104 are removed by proteolysis with thrombin Residues 1364-1460 are removed after ...Details: D560 described by Aspartate beryllium trifluoride (BFD) engineered C-terminal GGGG-LVPRGS-BAD-tag Residues 1-104 are removed by proteolysis with thrombin Residues 1364-1460 are removed after cleaving of the purification tag by thrombin Number of copies: 1 / Enantiomer: LEVO / EC number: P-type phospholipid transporter |
|---|---|
| Source (natural) | Organism:  Strain: ATCC 204508 / S288c |
| Molecular weight | Theoretical: 163.730766 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MNDDRETPPK RKPGEDDTLF DIDFLDDTTS HSGSRSKVTN SHANANYIPP SHVLPEETID LDADDDNIEN DVHENLFMSN NHDDQTSWN ANRFDSDAYQ PQSLRAVKPP GLFARFGNGL KNAFTFKRKK GPESFEMNHY NAVTNNELDD NYLDSRNKFN I KILFNRYI ...String: MNDDRETPPK RKPGEDDTLF DIDFLDDTTS HSGSRSKVTN SHANANYIPP SHVLPEETID LDADDDNIEN DVHENLFMSN NHDDQTSWN ANRFDSDAYQ PQSLRAVKPP GLFARFGNGL KNAFTFKRKK GPESFEMNHY NAVTNNELDD NYLDSRNKFN I KILFNRYI LRKNVGDAEG NGEPRVIHIN DSLANSSFGY SDNHISTTKY NFATFLPKFL FQEFSKYANL FFLCTSAIQQ VP HVSPTNR YTTIGTLLVV LIVSAMKECI EDIKRANSDK ELNNSTAEIF SEAHDDFVEK RWIDIRVGDI IRVKSEEPIP ADT IILSSS EPEGLCYIET ANLDGETNLK IKQSRVETAK FIDVKTLKNM NGKVVSEQPN SSLYTYEGTM TLNDRQIPLS PDQM ILRGA TLRNTAWIFG LVIFTGHETK LLRNATATPI KRTAVEKIIN RQIIALFTVL IVLILISSIG NVIMSTADAK HLSYL YLEG TNKAGLFFKD FLTFWILFSN LVPISLFVTV ELIKYYQAFM IGSDLDLYYE KTDTPTVVRT SSLVEELGQI EYIFS (BFD)KTG TLTRNIMEFK SCSIAGHCYI DKIPEDKTAT VEDGIEVGYR KFDDLKKKLN DPSDEDSPII NDFLTLLATC HT VIPEFQS DGSIKYQAAS PDEGALVQGG ADLGYKFIIR KPNSVTVLLE ETGEEKEYQL LNICEFNSTR KRMSAIFRFP DGS IKLFCK GADTVILERL DDEANQYVEA TMRHLEDYAS EGLRTLCLAM RDISEGEYEE WNSIYNEAAT TLDNRAEKLD EAAN LIEKN LILIGATAIE DKLQDGVPET IHTLQEAGIK IWVLTGDRQE TAINIGMSCR LLSEDMNLLI INEETRDDTE RNLLE KINA LNEHQLSTHD MNTLALVIDG KSLGFALEPE LEDYLLTVAK LCKAVICCRV SPLQKALVVK MVKRKSSSLL LAIGDG AND VSMIQAAHVG VGISGMEGMQ AARSADIAVG QFKFLKKLLL VHGSWSYQRI SVAILYSFYK NTALYMTQFW YVFANAF SG QSIMESWTMS FYNLFFTVWP PFVIGVFDQF VSSRLLERYP QLYKLGQKGQ FFSVYIFWGW IINGFFHSAI VFIGTILI Y RYGFALNMHG ELADHWSWGV TVYTTSVIIV LGKAALVTNQ WTKFTLIAIP GSLLFWLIFF PIYASIFPHA NISREYYGV VKHTYGSGVF WLTLIVLPIF ALVRDFLWKY YKRMYEPETY HVIQEMQKYN ISDSRPHVQQ FQNAIRKVRQ VQRMKKQRGF AFSQAEEGG QEKIVRMYDT TQKRGKYGEL QDASANPFND NNGLGSNDFE SAEPFIENPF ADGNQNSNRF SSSRDDISFD I GGGGLVPR GSGGTAAAPG PAPAPAPASA PAAAAPAGAG TPVTAPLAGT IWKVLASEGQ TVAAGEVLLI LEAMKMETEI RA AQAGTVR GIAVKAGDAV AVGDTLMT UniProtKB: Phospholipid-transporting ATPase DRS2 |
-Macromolecule #2: Cell division control protein 50
| Macromolecule | Name: Cell division control protein 50 / type: protein_or_peptide / ID: 2 / Details: engineered C-terminal GGGG-LVPRGS-GG-10xHistag / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Strain: ATCC 204508 / S288c |
| Molecular weight | Theoretical: 47.371797 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MVSLFKRGKA PPLTKEGPTS KKPPNTAFRQ QRLKAWQPIL SPQSVLPLLI FVACIFTPIG IGLIVSATKV QDLTIDYSHC DTKASTTAF EDIPKKYIKY HFKSKVENKP QWRLTENENG EQSCELQFEI PNDIKKSIFI YYKITNFYQN HRRYVQSFDT K QILGEPIK ...String: MVSLFKRGKA PPLTKEGPTS KKPPNTAFRQ QRLKAWQPIL SPQSVLPLLI FVACIFTPIG IGLIVSATKV QDLTIDYSHC DTKASTTAF EDIPKKYIKY HFKSKVENKP QWRLTENENG EQSCELQFEI PNDIKKSIFI YYKITNFYQN HRRYVQSFDT K QILGEPIK KDDLDTSCSP IRSREDKIIY PCGLIANSMF NDTFSQVLSG IDDTEDYNLT NKHISWSIDR HRFKTTKYNA SD IVPPPNW MKKYPDGYTD ENLPDIHTWE EFQVWMRTAA FPKFYKLTLK NESASLPKGK YQMNIELNYP ISLFGGTKSF VLT TNGAIG GRNMSLGVLY LIVAGLCALF GIIFLVKLIF QPRAMGDHTY LNFDDEENED YEDVHAENTT LREILGGGGL VPRG SGGHH HHHHHHHH UniProtKB: Phospholipid-transporting ATPase accessory subunit CDC50 |
-Macromolecule #5: MAGNESIUM ION
| Macromolecule | Name: MAGNESIUM ION / type: ligand / ID: 5 / Number of copies: 1 / Formula: MG |
|---|---|
| Molecular weight | Theoretical: 24.305 Da |
-Macromolecule #6: (2R)-1-{[(R)-hydroxy{[(1R,2R,3R,4R,5S,6R)-2,3,5,6-tetrahydroxy-4-...
| Macromolecule | Name: (2R)-1-{[(R)-hydroxy{[(1R,2R,3R,4R,5S,6R)-2,3,5,6-tetrahydroxy-4-(phosphonooxy)cyclohexyl]oxy}phosphoryl]oxy}-3-(octadecanoyloxy)propan-2-yl (5Z,8Z,11Z,14Z)-icosa-5,8,11,14-tetraenoate type: ligand / ID: 6 / Number of copies: 1 / Formula: 2Y5 |
|---|---|
| Molecular weight | Theoretical: 967.108 Da |
| Chemical component information |  ChemComp-2Y5: |
-Macromolecule #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
| Macromolecule | Name: 2-acetamido-2-deoxy-beta-D-glucopyranose / type: ligand / ID: 7 / Number of copies: 1 / Formula: NAG |
|---|---|
| Molecular weight | Theoretical: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-Macromolecule #8: water
| Macromolecule | Name: water / type: ligand / ID: 8 / Number of copies: 2 / Formula: HOH |
|---|---|
| Molecular weight | Theoretical: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 0.6 mg/mL |
|---|---|
| Buffer | pH: 7 |
| Grid | Model: C-flat-1.2/1.3 / Material: COPPER / Mesh: 400 / Support film - Material: CARBON / Support film - topology: HOLEY / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Time: 45 sec. / Details: 15 mA |
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 278 K / Instrument: FEI VITROBOT MARK IV |
| Details | 1mM beryllium fluoride 0.1 mg/mL Brain PI4P in DDM |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K2 QUANTUM (4k x 4k) / Detector mode: COUNTING / Average electron dose: 60.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller


























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)