+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4259 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of F-actin in complex with ADP-Pi | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Cytoskeleton / nucleotide states / filament stability / cell migration / STRUCTURAL PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytoskeletal motor activator activity / myosin heavy chain binding / tropomyosin binding / actin filament bundle / troponin I binding / filamentous actin / mesenchyme migration / actin filament bundle assembly / skeletal muscle myofibril / striated muscle thin filament ...cytoskeletal motor activator activity / myosin heavy chain binding / tropomyosin binding / actin filament bundle / troponin I binding / filamentous actin / mesenchyme migration / actin filament bundle assembly / skeletal muscle myofibril / striated muscle thin filament / skeletal muscle thin filament assembly / actin monomer binding / skeletal muscle fiber development / stress fiber / titin binding / actin filament polymerization / actin filament / filopodium / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / calcium-dependent protein binding / lamellipodium / cell body / hydrolase activity / protein domain specific binding / calcium ion binding / positive regulation of gene expression / magnesium ion binding / ATP binding / identical protein binding / cytoplasm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | ||||||||||||
 データ登録者 データ登録者 | Merino F / Pospich S | ||||||||||||
| 資金援助 |  ドイツ, 3件 ドイツ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2018 ジャーナル: Nat Struct Mol Biol / 年: 2018タイトル: Structural transitions of F-actin upon ATP hydrolysis at near-atomic resolution revealed by cryo-EM. 著者: Felipe Merino / Sabrina Pospich / Johanna Funk / Thorsten Wagner / Florian Küllmer / Hans-Dieter Arndt / Peter Bieling / Stefan Raunser /  要旨: The function of actin is coupled to the nucleotide bound to its active site. ATP hydrolysis is activated during polymerization; a delay between hydrolysis and inorganic phosphate (P) release results ...The function of actin is coupled to the nucleotide bound to its active site. ATP hydrolysis is activated during polymerization; a delay between hydrolysis and inorganic phosphate (P) release results in a gradient of ATP, ADP-P and ADP along actin filaments (F-actin). Actin-binding proteins can recognize F-actin's nucleotide state, using it as a local 'age' tag. The underlying mechanism is complex and poorly understood. Here we report six high-resolution cryo-EM structures of F-actin from rabbit skeletal muscle in different nucleotide states. The structures reveal that actin polymerization repositions the proposed catalytic base, His161, closer to the γ-phosphate. Nucleotide hydrolysis and P release modulate the conformational ensemble at the periphery of the filament, thus resulting in open and closed states, which can be sensed by coronin-1B. The drug-like toxin jasplakinolide locks F-actin in an open state. Our results demonstrate in detail how ATP hydrolysis links to F-actin's conformational dynamics and protein interaction. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4259.map.gz emd_4259.map.gz | 59.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4259-v30.xml emd-4259-v30.xml emd-4259.xml emd-4259.xml | 23.9 KB 23.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_4259.png emd_4259.png | 117.1 KB | ||
| マスクデータ |  emd_4259_msk_1.map emd_4259_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-4259.cif.gz emd-4259.cif.gz | 6.9 KB | ||
| その他 |  emd_4259_half_map_1.map.gz emd_4259_half_map_1.map.gz emd_4259_half_map_2.map.gz emd_4259_half_map_2.map.gz | 49.3 MB 49.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4259 http://ftp.pdbj.org/pub/emdb/structures/EMD-4259 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4259 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4259 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4259_validation.pdf.gz emd_4259_validation.pdf.gz | 816.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4259_full_validation.pdf.gz emd_4259_full_validation.pdf.gz | 815.9 KB | 表示 | |
| XML形式データ |  emd_4259_validation.xml.gz emd_4259_validation.xml.gz | 12.3 KB | 表示 | |
| CIF形式データ |  emd_4259_validation.cif.gz emd_4259_validation.cif.gz | 14.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4259 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4259 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4259 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4259 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6fhlMC  3835C  3836C  3837C  3838C  3839C  5onvC  5oocC  5oodC  5ooeC  5oofC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4259.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4259.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_4259_msk_1.map emd_4259_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
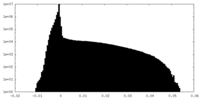
| 密度ヒストグラム |
-ハーフマップ: Second half map Actin-ADPPi
| ファイル | emd_4259_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Second half map Actin-ADPPi | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: First half map Actin-ADPPi
| ファイル | emd_4259_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | First half map Actin-ADPPi | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Filamentous alpha actin in complex with ADP-ADPPi
| 全体 | 名称: Filamentous alpha actin in complex with ADP-ADPPi |
|---|---|
| 要素 |
|
-超分子 #1: Filamentous alpha actin in complex with ADP-ADPPi
| 超分子 | 名称: Filamentous alpha actin in complex with ADP-ADPPi / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Actin, alpha skeletal muscle
| 分子 | 名称: Actin, alpha skeletal muscle / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 41.875633 KDa |
| 配列 | 文字列: DEDETTALVC DNGSGLVKAG FAGDDAPRAV FPSIVGRPRH QGVMVGMGQK DSYVGDEAQS KRGILTLKYP IE(HIC)GII TNW DDMEKIWHHT FYNELRVAPE EHPTLLTEAP LNPKANREKM TQIMFETFNV PAMYVAIQAV LSLYASGRTT GIVLDSG DG ...文字列: DEDETTALVC DNGSGLVKAG FAGDDAPRAV FPSIVGRPRH QGVMVGMGQK DSYVGDEAQS KRGILTLKYP IE(HIC)GII TNW DDMEKIWHHT FYNELRVAPE EHPTLLTEAP LNPKANREKM TQIMFETFNV PAMYVAIQAV LSLYASGRTT GIVLDSG DG VTHNVPIYEG YALPHAIMRL DLAGRDLTDY LMKILTERGY SFVTTAEREI VRDIKEKLCY VALDFENEMA TAASSSSL E KSYELPDGQV ITIGNERFRC PETLFQPSFI GMESAGIHET TYNSIMKCDI DIRKDLYANN VMSGGTTMYP GIADRMQKE ITALAPSTMK IKIIAPPERK YSVWIGGSIL ASLSTFQQMW ITKQEYDEAG PSIVHRKCF UniProtKB: Actin, alpha skeletal muscle |
-分子 #2: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 5 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 5 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: PHOSPHATE ION
| 分子 | 名称: PHOSPHATE ION / タイプ: ligand / ID: 4 / コピー数: 5 / 式: PO4 |
|---|---|
| 分子量 | 理論値: 94.971 Da |
| Chemical component information |  ChemComp-PO4: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
詳細: 5 mM HEPES pH 7.5, 0.05 M KCl, 2 mM MgCl2, 2 mM NaN3, 0.5 mM TCEP, 0.2 mM ADP, 50 mM potassium phosphate. | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 286 K / 装置: FEI VITROBOT MARK III / 詳細: 8s blotting, 1s drain time, -25 force. | ||||||||||||||||||||||||
| 詳細 | Rise 26.9 A, Twist -166.7 degrees |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 球面収差補正装置: Cs-corrected microscope |
| 詳細 | Cs-corrected microscope |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / デジタル化 - 画像ごとのフレーム数: 1-5 / 実像数: 2614 / 平均露光時間: 1.5 sec. / 平均電子線量: 93.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Rosetta iterative refinement combined with MDFF. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER |
| 得られたモデル |  PDB-6fhl: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)