+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of C-terminal LRRK2 bound to MLi-2 (G2019S mutant) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | GtPase / kinase / inhibitors / PROTEIN BINDING | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報caveola neck / negative regulation of protein processing involved in protein targeting to mitochondrion / Wnt signalosome assembly / beta-catenin destruction complex binding / regulation of branching morphogenesis of a nerve / regulation of kidney size / regulation of cell projection organization / tangential migration from the subventricular zone to the olfactory bulb / protein localization to endoplasmic reticulum exit site / GTP-dependent protein kinase activity ...caveola neck / negative regulation of protein processing involved in protein targeting to mitochondrion / Wnt signalosome assembly / beta-catenin destruction complex binding / regulation of branching morphogenesis of a nerve / regulation of kidney size / regulation of cell projection organization / tangential migration from the subventricular zone to the olfactory bulb / protein localization to endoplasmic reticulum exit site / GTP-dependent protein kinase activity / regulation of SNARE complex assembly / regulation of neuroblast proliferation / regulation of ER to Golgi vesicle-mediated transport / peroxidase inhibitor activity / negative regulation of late endosome to lysosome transport / regulation of mitochondrial depolarization / negative regulation of protein targeting to mitochondrion / positive regulation of dopamine receptor signaling pathway / regulation of synaptic vesicle transport / regulation of CAMKK-AMPK signaling cascade / regulation of lysosomal lumen pH / amphisome / co-receptor binding / mitochondrion localization / regulation of dopamine receptor signaling pathway / regulation of retrograde transport, endosome to Golgi / regulation of neuron maturation / positive regulation of microglial cell activation / positive regulation of synaptic vesicle endocytosis / negative regulation of autophagosome assembly / cytoplasmic side of mitochondrial outer membrane / negative regulation of excitatory postsynaptic potential / regulation of cAMP/PKA signal transduction / JUN kinase kinase kinase activity / olfactory bulb development / neuron projection arborization / striatum development / multivesicular body, internal vesicle / regulation of dendritic spine morphogenesis / protein localization to mitochondrion / cellular response to dopamine / positive regulation of mitochondrial outer membrane permeabilization involved in apoptotic signaling pathway / endoplasmic reticulum organization / positive regulation of protein autoubiquitination / Wnt signalosome / positive regulation of programmed cell death / negative regulation of protein processing / GTP metabolic process / syntaxin-1 binding / regulation of canonical Wnt signaling pathway / negative regulation of GTPase activity / regulation of reactive oxygen species metabolic process / lysosome organization / Golgi-associated vesicle / clathrin binding / regulation of locomotion / negative regulation of macroautophagy / PTK6 promotes HIF1A stabilization / protein kinase A binding / neuromuscular junction development / regulation of mitochondrial fission / Golgi organization / exploration behavior / regulation of synaptic vesicle exocytosis / microvillus / intracellular distribution of mitochondria / autolysosome / locomotory exploration behavior / endoplasmic reticulum exit site / negative regulation of Notch signaling pathway / regulation of synaptic vesicle endocytosis / cellular response to manganese ion / Rho protein signal transduction / MAP kinase kinase kinase activity / canonical Wnt signaling pathway / negative regulation of endoplasmic reticulum stress-induced intrinsic apoptotic signaling pathway / presynaptic cytosol / regulation of synaptic transmission, glutamatergic / phagocytic vesicle / JNK cascade / positive regulation of autophagy / tubulin binding / dendrite cytoplasm / GTPase activator activity / SNARE binding / cellular response to starvation / neuron projection morphogenesis / positive regulation of protein ubiquitination / excitatory postsynaptic potential / regulation of membrane potential / determination of adult lifespan / cellular response to reactive oxygen species / mitochondrion organization / trans-Golgi network / calcium-mediated signaling / mitochondrial membrane / regulation of protein stability / autophagy / small GTPase binding / terminal bouton 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
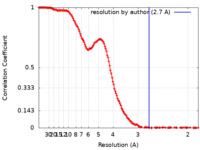
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Sanz-Murillo M / Villagran-Suarez A / Alegrio-Louro J / Leschziner A | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2023 ジャーナル: Sci Adv / 年: 2023タイトル: Inhibition of Parkinson's disease-related LRRK2 by type I and type II kinase inhibitors: Activity and structures. 著者: Marta Sanz Murillo / Amalia Villagran Suarez / Verena Dederer / Deep Chatterjee / Jaime Alegrio Louro / Stefan Knapp / Sebastian Mathea / Andres E Leschziner /   要旨: Mutations in leucine-rich repeat kinase 2 (LRRK2) are a common cause of familial Parkinson's disease (PD) and a risk factor for the sporadic form. Increased kinase activity was shown in patients with ...Mutations in leucine-rich repeat kinase 2 (LRRK2) are a common cause of familial Parkinson's disease (PD) and a risk factor for the sporadic form. Increased kinase activity was shown in patients with both familial and sporadic PD, making LRRK2 kinase inhibitors a major focus of drug development efforts. Although much progress has been made in understanding the structural biology of LRRK2, there are no available structures of LRRK2 inhibitor complexes. To this end, we solved cryo-electron microscopy structures of LRRK2, wild-type and PD-linked mutants, bound to the LRRK2-specific type I inhibitor MLi-2 and the broad-spectrum type II inhibitor GZD-824. Our structures revealed an active-like LRRK2 kinase in the type I inhibitor complex, and an inactive DYG-out in the type II inhibitor complex. Our structural analysis also showed how inhibitor-induced conformational changes in LRRK2 are affected by its autoinhibitory N-terminal repeats. The structures provide a template for the rational development of LRRK2 kinase inhibitors covering both canonical inhibitor binding modes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41754.map.gz emd_41754.map.gz | 194 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41754-v30.xml emd-41754-v30.xml emd-41754.xml emd-41754.xml | 23.3 KB 23.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41754_fsc.xml emd_41754_fsc.xml | 18.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41754.png emd_41754.png | 81.6 KB | ||
| Filedesc metadata |  emd-41754.cif.gz emd-41754.cif.gz | 7.4 KB | ||
| その他 |  emd_41754_additional_1.map.gz emd_41754_additional_1.map.gz emd_41754_half_map_1.map.gz emd_41754_half_map_1.map.gz emd_41754_half_map_2.map.gz emd_41754_half_map_2.map.gz | 7.1 MB 226.8 MB 226.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41754 http://ftp.pdbj.org/pub/emdb/structures/EMD-41754 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41754 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41754 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41754_validation.pdf.gz emd_41754_validation.pdf.gz | 870.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41754_full_validation.pdf.gz emd_41754_full_validation.pdf.gz | 870.4 KB | 表示 | |
| XML形式データ |  emd_41754_validation.xml.gz emd_41754_validation.xml.gz | 21.4 KB | 表示 | |
| CIF形式データ |  emd_41754_validation.cif.gz emd_41754_validation.cif.gz | 28.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41754 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41754 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41754 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41754 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8tzcMC  8txzC  8tyqC  8tzbC  8tzeC  8tzfC  8tzgC  8tzhC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41754.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41754.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.935 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_41754_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
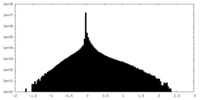
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_41754_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
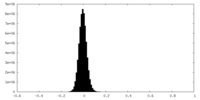
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_41754_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
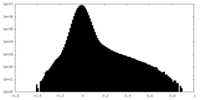
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : C-terminal LRRK2 (G2019S mutation) bound to MLi-2
| 全体 | 名称: C-terminal LRRK2 (G2019S mutation) bound to MLi-2 |
|---|---|
| 要素 |
|
-超分子 #1: C-terminal LRRK2 (G2019S mutation) bound to MLi-2
| 超分子 | 名称: C-terminal LRRK2 (G2019S mutation) bound to MLi-2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 137 KDa |
-分子 #1: Leucine-rich repeat serine/threonine-protein kinase 2
| 分子 | 名称: Leucine-rich repeat serine/threonine-protein kinase 2 タイプ: protein_or_peptide / ID: 1 / 詳細: Several loops are missed in the structure / コピー数: 1 / 光学異性体: LEVO / EC番号: non-specific serine/threonine protein kinase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 136.204797 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: NRMKLMIVGN TGSGKTTLLQ QLMKTKKSDL GMQSATVGID VKDWPIQIRD KRKRDLVLNV WDFAGREEFY STHPHFMTQR ALYLAVYDL SKGQAEVDAM KPWLFNIKAR ASSSPVILVG THLDVSDEKQ RKACMSKITK ELLNKRGFPA IRDYHFVNAT E ESDALAKL ...文字列: NRMKLMIVGN TGSGKTTLLQ QLMKTKKSDL GMQSATVGID VKDWPIQIRD KRKRDLVLNV WDFAGREEFY STHPHFMTQR ALYLAVYDL SKGQAEVDAM KPWLFNIKAR ASSSPVILVG THLDVSDEKQ RKACMSKITK ELLNKRGFPA IRDYHFVNAT E ESDALAKL RKTIINESLN FKIRDQLVVG QLIPDCYVEL EKIILSERKN VPIEFPVIDR KRLLQLVREN QLQLDENELP HA VHFLNES GVLLHFQDPA LQLSDLYFVE PKWLCKIMAQ ILTVKVEGCP KHPKGIISRR DVEKFLSKKR KFPKNYMSQY FKL LEKFQI ALPIGEEYLL VPSSLSDHRP VIELPHCENS EIIIRLYEMP YFPMGFWSRL INRLLEISPY MLSGRERALR PNRM YWRQG IYLNWSPEAY CLVGSEVLDN HPESFLKITV PSCRKGCILL GQVVDHIDSL MEEWFPGLLE IDICGEGETL LKKWA LYSF NDGEEHQKIL LDDLMKKAEE GDLLVNPDQP RLTIPISQIA PDLILADLPR NIMLNNDELE FEQAPEFLLG DGSFGS VYR AAYEGEEVAV KIFNKHTSLR LLRQELVVLC HLHHPSLISL LAAGIRPRML VMELASKGSL DRLLQQDKAS LTRTLQH RI ALHVADGLRY LHSAMIIYRD LKPHNVLLFT LYPNAAIIAK IADYSIAQYC CRMGIKTSEG TPGFRAPEVA RGNVIYNQ Q ADVYSFGLLL YDILTTGGRI VEGLKFPNEF DELEIQGKLP DPVKEYGCAP WPMVEKLIKQ CLKENPQERP TSAQVFDIL NSAELVCLTR RILLPKNVIV ECMVATHHNS RNASIWLGCG HTDRGQLSFL DLNTEGYTSE EVADSRILCL ALVHLPVEKE SWIVSGTQS GTLLVINTED GKKRHTLEKM TDSVTCLYCN SFSKQSKQKN FLLVGTADGK LAIFEDKTVK LKGAAPLKIL N IGNVSTPL MCLSESTNST ERNVMWGGCG TKIFSFSNDF TIQKLIETRT SQLFSYAAFS DSNIITVVVD TALYIAKQNS PV VEVWDKK TEKLCGLIDC VHFLREVMVK ENKESKHKMS YSGRVKTLCL QKNTALWIGT GGGHILLLDL STRRLIRVIY NFC NSVRVM MTAQLGSLKN VMLVLGYNRK NTEGTQKQKE IQSCLTVWDI NLPHEVQNLE KHIEVRKELA EKMRRTSVE UniProtKB: Leucine-rich repeat serine/threonine-protein kinase 2 |
-分子 #2: E11 DARPin
| 分子 | 名称: E11 DARPin / タイプ: protein_or_peptide / ID: 2 詳細: Some areas are missing due to the flexibility of the protein コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 19.766912 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRGSHHHHHH HHGSDLGKKL LEAARAGQDD EVRILMANGA DVNATDEAGV TPLHLAADSG HLEIVEVLLK TGADVNAWDH YGFTPLHLA AHVGHLEIVE VLLKAGADVN AQDHAGWTPL HLAALYGHLE IVEVLLKHGA DVNAQDMWGE TPFDLAIDNG N EDIAEVLQ KAAKLNDYKD DDDK |
-分子 #3: (2~{R},6~{S})-2,6-dimethyl-4-[6-[5-(1-methylcyclopropyl)oxy-1~{H}...
| 分子 | 名称: (2~{R},6~{S})-2,6-dimethyl-4-[6-[5-(1-methylcyclopropyl)oxy-1~{H}-indazol-3-yl]pyrimidin-4-yl]morpholine タイプ: ligand / ID: 3 / コピー数: 1 / 式: A1N |
|---|---|
| 分子量 | 理論値: 379.456 Da |
| Chemical component information |  ChemComp-A1N: |
-分子 #4: GUANOSINE-5'-DIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: GDP |
|---|---|
| 分子量 | 理論値: 443.201 Da |
| Chemical component information |  ChemComp-GDP: |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 1 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.822 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||||||||||||||
| グリッド | モデル: UltrAuFoil R2/2 / 材質: GOLD / メッシュ: 200 / 前処理 - タイプ: PLASMA CLEANING | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: Blot force = 3 Blot time = 4 seconds Wait time = 20 seconds. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 1 / 実像数: 8788 / 平均露光時間: 11.0 sec. / 平均電子線量: 55.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)