+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3949 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
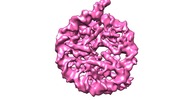
| タイトル | Nucleosome breathing : Class 3 | |||||||||
 マップデータ マップデータ | Nucleosome breathing : Class 3 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | nucleosome / nucleosome breathing / hexasome / GENE REGULATION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報structural constituent of chromatin / nucleosome / nucleosome assembly / protein heterodimerization activity / DNA binding / nucleoplasm / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 | ||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.1 Å | |||||||||
 データ登録者 データ登録者 | Bilokapic S / Halic M | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2018 ジャーナル: Nat Struct Mol Biol / 年: 2018タイトル: Histone octamer rearranges to adapt to DNA unwrapping. 著者: Silvija Bilokapic / Mike Strauss / Mario Halic /  要旨: Nucleosomes, the basic units of chromatin, package and regulate expression of eukaryotic genomes. Although the structure of the intact nucleosome is well characterized, little is known about ...Nucleosomes, the basic units of chromatin, package and regulate expression of eukaryotic genomes. Although the structure of the intact nucleosome is well characterized, little is known about structures of partially unwrapped, transient intermediates. In this study, we present nine cryo-EM structures of distinct conformations of nucleosome and subnucleosome particles. These structures show that initial DNA breathing induces conformational changes in the histone octamer, particularly in histone H3, that propagate through the nucleosome and prevent symmetrical DNA opening. Rearrangements in the H2A-H2B dimer strengthen interaction with the unwrapping DNA and promote nucleosome stability. In agreement with this, cross-linked H2A-H2B that cannot accommodate unwrapping of the DNA is not stably maintained in the nucleosome. H2A-H2B release and DNA unwrapping occur simultaneously, indicating that DNA is essential in stabilizing the dimer in the nucleosome. Our structures reveal intrinsic nucleosomal plasticity that is required for nucleosome stability and might be exploited by extrinsic protein factors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3949.map.gz emd_3949.map.gz | 10.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3949-v30.xml emd-3949-v30.xml emd-3949.xml emd-3949.xml | 16.9 KB 16.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3949.png emd_3949.png | 249.8 KB | ||
| Filedesc metadata |  emd-3949.cif.gz emd-3949.cif.gz | 5.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3949 http://ftp.pdbj.org/pub/emdb/structures/EMD-3949 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3949 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3949 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3949_validation.pdf.gz emd_3949_validation.pdf.gz | 223 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3949_full_validation.pdf.gz emd_3949_full_validation.pdf.gz | 222.1 KB | 表示 | |
| XML形式データ |  emd_3949_validation.xml.gz emd_3949_validation.xml.gz | 5.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3949 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3949 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3949 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3949 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6eshMC  3925C  3926C  3929C  3930C  3931C  3947C  3948C  3950C  6esfC  6esgC  6esiC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3949.map.gz / 形式: CCP4 / 大きさ: 18.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3949.map.gz / 形式: CCP4 / 大きさ: 18.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Nucleosome breathing : Class 3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Nucleosome
| 全体 | 名称: Nucleosome |
|---|---|
| 要素 |
|
-超分子 #1: Nucleosome
| 超分子 | 名称: Nucleosome / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 200 KDa |
-超分子 #2: Nucleosome
| 超分子 | 名称: Nucleosome / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種: |
-超分子 #3: DNA
| 超分子 | 名称: DNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #5-#6 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: Histone H3.2
| 分子 | 名称: Histone H3.2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 15.30393 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ARTKQTARKS TGGKAPRKQL ATKAARKSAP ATGGVKKPHR YRPGTVALRE IRRYQKSTEL LIRKLPFQRL VREIAQDFKT DLRFQSSAV MALQEASEAY LVALFEDTNL CAIHAKRVTI MPKDIQLARR IRGERA UniProtKB: Histone H3.2 |
-分子 #2: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 11.263231 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGRGKGGKGL GKGGAKRHRK VLRDNIQGIT KPAIRRLARR GGVKRISGLI YEETRGVLKV FLENVIRDAV TYTEHAKRKT VTAMDVVYA LKRQGRTLYG FGG UniProtKB: Histone H4 |
-分子 #3: Histone H2A
| 分子 | 名称: Histone H2A / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 13.978241 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGRGKQGGKT RAKAKTRSSR AGLQFPVGRV HRLLRKGNYA ERVGAGAPVY LAAVLEYLTA EILELAGNAA RDNKKTRIIP RHLQLAVRN DEELNKLLGR VTIAQGGVLP NIQSVLLPKK TESSKSAKSK UniProtKB: Histone H2A |
-分子 #4: Histone H2B 1.1
| 分子 | 名称: Histone H2B 1.1 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 13.524752 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AKSAPAPKKG SKKAVTKTQK KDGKKRRKTR KESYAIYVYK VLKQVHPDTG ISSKAMSIMN SFVNDVFERI AGEASRLAHY NKRSTITSR EIQTAVRLLL PGELAKHAVS EGTKAVTKYT SAK UniProtKB: Histone H2B 1.1 |
-分子 #5: DNA (137-MER)
| 分子 | 名称: DNA (137-MER) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 45.604047 KDa |
| 配列 | 文字列: (DA)(DC)(DA)(DG)(DG)(DA)(DT)(DG)(DT)(DA) (DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG) (DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG) (DA) (DG)(DT)(DA)(DA)(DT) ...文字列: (DA)(DC)(DA)(DG)(DG)(DA)(DT)(DG)(DT)(DA) (DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG) (DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG) (DA) (DG)(DT)(DA)(DA)(DT)(DC)(DC)(DC) (DC)(DT)(DT)(DG)(DG)(DC)(DG)(DG)(DT)(DT) (DA)(DA) (DA)(DA)(DC)(DG)(DC)(DG)(DG) (DG)(DG)(DG)(DA)(DC)(DA)(DG)(DC)(DG)(DC) (DG)(DT)(DA) (DC)(DG)(DT)(DG)(DC)(DG) (DT)(DT)(DT)(DA)(DA)(DG)(DC)(DG)(DG)(DT) (DG)(DC)(DT)(DA) (DG)(DA)(DG)(DC)(DT) (DG)(DT)(DC)(DT)(DA)(DC)(DG)(DA)(DC)(DC) (DA)(DA)(DT)(DT)(DG) (DA)(DG)(DC)(DG) (DG)(DC)(DC)(DT)(DC)(DG)(DG)(DC)(DA)(DC) (DC)(DG)(DG)(DG)(DA)(DT) (DT)(DC)(DT) (DC)(DC)(DA)(DG) |
-分子 #6: DNA (137-MER)
| 分子 | 名称: DNA (137-MER) / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 45.145754 KDa |
| 配列 | 文字列: (DC)(DT)(DG)(DG)(DA)(DG)(DA)(DA)(DT)(DC) (DC)(DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA) (DG)(DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA) (DA)(DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA) (DG) (DA)(DC)(DA)(DG)(DC) ...文字列: (DC)(DT)(DG)(DG)(DA)(DG)(DA)(DA)(DT)(DC) (DC)(DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA) (DG)(DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA) (DA)(DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA) (DG) (DA)(DC)(DA)(DG)(DC)(DT)(DC)(DT) (DA)(DG)(DC)(DA)(DC)(DC)(DG)(DC)(DT)(DT) (DA)(DA) (DA)(DC)(DG)(DC)(DA)(DC)(DG) (DT)(DA)(DC)(DG)(DC)(DG)(DC)(DT)(DG)(DT) (DC)(DC)(DC) (DC)(DC)(DG)(DC)(DG)(DT) (DT)(DT)(DT)(DA)(DA)(DC)(DC)(DG)(DC)(DC) (DA)(DA)(DG)(DG) (DG)(DG)(DA)(DT)(DT) (DA)(DC)(DT)(DC)(DC)(DC)(DT)(DA)(DG)(DT) (DC)(DT)(DC)(DC)(DA) (DG)(DG)(DC)(DA) (DC)(DG)(DT)(DG)(DT)(DC)(DA)(DG)(DA)(DT) (DA)(DT)(DA)(DT)(DA)(DC) (DA)(DT)(DC) (DC)(DT)(DG)(DT) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #0 - 検出モード: COUNTING / #0 - 平均電子線量: 80.0 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: FEI FALCON II (4k x 4k) #1 - 検出モード: INTEGRATING / #1 - 平均電子線量: 100.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















