+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21955 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM reconstruction of VP5*/VP8* assembly from rhesus rotavirus particles - Upright conformation | |||||||||
 マップデータ マップデータ | Cryo-EM reconstruction of VP5*/VP8* assembly from rhesus rotavirus particles - Upright conformation | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral intermediate capsid / host cell endoplasmic reticulum lumen / T=13 icosahedral viral capsid / host cell rough endoplasmic reticulum / host cytoskeleton / viral outer capsid / permeabilization of host organelle membrane involved in viral entry into host cell / symbiont entry into host cell via permeabilization of inner membrane / host cell endoplasmic reticulum-Golgi intermediate compartment / receptor-mediated virion attachment to host cell ...viral intermediate capsid / host cell endoplasmic reticulum lumen / T=13 icosahedral viral capsid / host cell rough endoplasmic reticulum / host cytoskeleton / viral outer capsid / permeabilization of host organelle membrane involved in viral entry into host cell / symbiont entry into host cell via permeabilization of inner membrane / host cell endoplasmic reticulum-Golgi intermediate compartment / receptor-mediated virion attachment to host cell / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / structural molecule activity / membrane / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Rotavirus A (strain RVA/Monkey/United States/RRV/1975/G3P5B[3]) (ウイルス) / Rotavirus A (strain RVA/Monkey/United States/RRV/1975/G3P5B[3]) (ウイルス) /  RV-A (ウイルス) RV-A (ウイルス) | |||||||||
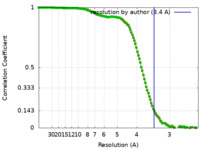
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Herrmann T / Harrison SC / Jenni S | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: Functional refolding of the penetration protein on a non-enveloped virus. 著者: Tobias Herrmann / Raúl Torres / Eric N Salgado / Cristina Berciu / Daniel Stoddard / Daniela Nicastro / Simon Jenni / Stephen C Harrison /  要旨: A non-enveloped virus requires a membrane lesion to deliver its genome into a target cell. For rotaviruses, membrane perforation is a principal function of the viral outer-layer protein, VP4. Here we ...A non-enveloped virus requires a membrane lesion to deliver its genome into a target cell. For rotaviruses, membrane perforation is a principal function of the viral outer-layer protein, VP4. Here we describe the use of electron cryomicroscopy to determine how VP4 performs this function and show that when activated by cleavage to VP8* and VP5*, VP4 can rearrange on the virion surface from an 'upright' to a 'reversed' conformation. The reversed structure projects a previously buried 'foot' domain outwards into the membrane of the host cell to which the virion has attached. Electron cryotomograms of virus particles entering cells are consistent with this picture. Using a disulfide mutant of VP4, we have also stabilized a probable intermediate in the transition between the two conformations. Our results define molecular mechanisms for the first steps of the penetration of rotaviruses into the membranes of target cells and suggest similarities with mechanisms postulated for other viruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21955.map.gz emd_21955.map.gz | 117 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21955-v30.xml emd-21955-v30.xml emd-21955.xml emd-21955.xml | 24 KB 24 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21955_fsc.xml emd_21955_fsc.xml emd_21955_fsc_2.xml emd_21955_fsc_2.xml | 14.6 KB 14.6 KB | 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21955.png emd_21955.png | 104.4 KB | ||
| マスクデータ |  emd_21955_msk_1.map emd_21955_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| その他 |  emd_21955_half_map_1.map.gz emd_21955_half_map_1.map.gz emd_21955_half_map_2.map.gz emd_21955_half_map_2.map.gz | 98.2 MB 98.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21955 http://ftp.pdbj.org/pub/emdb/structures/EMD-21955 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21955 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21955 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21955_validation.pdf.gz emd_21955_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21955_full_validation.pdf.gz emd_21955_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_21955_validation.xml.gz emd_21955_validation.xml.gz | 18.7 KB | 表示 | |
| CIF形式データ |  emd_21955_validation.cif.gz emd_21955_validation.cif.gz | 24.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21955 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21955 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21955 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21955 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21955.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21955.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM reconstruction of VP5*/VP8* assembly from rhesus rotavirus particles - Upright conformation | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.231 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
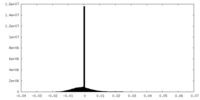
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_21955_msk_1.map emd_21955_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
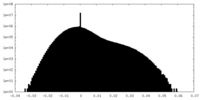
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_21955_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_21955_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rotavirus VP4, VP6, VP7 assembly in the spike conformation
| 全体 | 名称: Rotavirus VP4, VP6, VP7 assembly in the spike conformation |
|---|---|
| 要素 |
|
-超分子 #1: Rotavirus VP4, VP6, VP7 assembly in the spike conformation
| 超分子 | 名称: Rotavirus VP4, VP6, VP7 assembly in the spike conformation タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: Obtained from wild-type recoated rhesus rotavirus particles (wt rcTLPs) |
|---|---|
| 由来(天然) | 生物種:  Rotavirus A (strain RVA/Monkey/United States/RRV/1975/G3P5B[3]) (ウイルス) Rotavirus A (strain RVA/Monkey/United States/RRV/1975/G3P5B[3]) (ウイルス) |
| 分子量 | 理論値: 1.7 MDa |
-分子 #1: Outer capsid protein VP4
| 分子 | 名称: Outer capsid protein VP4 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rotavirus A (strain RVA/Monkey/United States/RRV/1975/G3P5B[3]) (ウイルス) Rotavirus A (strain RVA/Monkey/United States/RRV/1975/G3P5B[3]) (ウイルス)株: RVA/Monkey/United States/RRV/1975/G3P5B[3] |
| 分子量 | 理論値: 86.655586 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASLIYRQLL TNSYTVDLSD EIQEIGSTKT QNVTINLGPF AQTGYAPVNW GPGETNDSTT VEPVLDGPYQ PTTFNPPVDY WMLLAPTAA GVVVEGTNNT DRWLATILVE PNVTSETRSY TLFGTQEQIT IANASQTQWK FIDVVKTTQN GSYSQYGPLQ S TPKLYAVM ...文字列: MASLIYRQLL TNSYTVDLSD EIQEIGSTKT QNVTINLGPF AQTGYAPVNW GPGETNDSTT VEPVLDGPYQ PTTFNPPVDY WMLLAPTAA GVVVEGTNNT DRWLATILVE PNVTSETRSY TLFGTQEQIT IANASQTQWK FIDVVKTTQN GSYSQYGPLQ S TPKLYAVM KHNGKIYTYN GETPNVTTKY YSTTNYDSVN MTAFCDFYII PREEESTCTE YINNGLPPIQ NTRNIVPLAL SA RNIISHR AQANEDIVVS KTSLWKEMQY NRDITIRFKF ASSIVKSGGL GYKWSEISFK PANYQYTYTR DGEEVTAHTT CSV NGMNDF NFNGGSLPTD FVISRYEVIK ENSYVYVDYW DDSQAFRNMV YVRSLAANLN SVICTGGDYS FALPVGQWPV MTGG AVSLH SAGVTLSTQF TDFVSLNSLR FRFRLTVEEP SFSITRTRVS RLYGLPAANP NNGKEYYEVA GRFSLISLVP SNDDY QTPI TNSVTVRQDL ERQLGELREE FNALSQEIAM SQLIDLALLP LDMFSMFSGI KSTIDAAKSM ATSVMKKFKK SGLANS VST LTDSLSDAAS SISRGASIRS VGSSASAWTD VSTQITDVSS SVSSISTQTS TISRRLRLKE MATQTEGMNF DDISAAV LK TKIDRSTQIS PNTLPDIVTE ASEKFIPNRA YRVINNDEVF EAGTDGRFFA YRVETFDEIP FDVQKFADLV TDSPVISA I IDFKTLKNLN DNYGISRQQA FNLLRSDPRV LREFINQDNP IIRNRIEQLI MQCRL |
-分子 #2: Intermediate capsid protein VP6
| 分子 | 名称: Intermediate capsid protein VP6 / タイプ: protein_or_peptide / ID: 2 / コピー数: 18 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  RV-A (ウイルス) / 株: RVA/Monkey/United States/RRV/1975/G3P5B[3] RV-A (ウイルス) / 株: RVA/Monkey/United States/RRV/1975/G3P5B[3] |
| 分子量 | 理論値: 44.934766 KDa |
| 配列 | 文字列: MDVLYSLSKT LKDARDKIVE GTLYSNVSDL IQQFNQMIIT MNGNEFQTGG IGNLPIRNWN FDFGLLGTTL LNLDANYVET ARNTIDYFV DFVDNVCMDE MVRESQRNGI APQSDSLRKL SGIKFKRINF DNSSEYIENW NLQNRRQRTG FTFHKPNIFP Y SASFTLNR ...文字列: MDVLYSLSKT LKDARDKIVE GTLYSNVSDL IQQFNQMIIT MNGNEFQTGG IGNLPIRNWN FDFGLLGTTL LNLDANYVET ARNTIDYFV DFVDNVCMDE MVRESQRNGI APQSDSLRKL SGIKFKRINF DNSSEYIENW NLQNRRQRTG FTFHKPNIFP Y SASFTLNR SQPAHDNLMG TMWLNAGSEI QVAGFDYSCA INAPANIQQF EHIVQLRRVL TTATITLLPD AERFSFPRVI NS ADGATTW YFNPVILRPN NVEVEFLLNG QIINTYQARF GTIIARNFDT IRLSFQLMRP PNMTPAVAAL FPNAQPFEHH ATV GLTLRI ESAVCESVLA DASKTMLANV TSVRQEYAIP VGPVFPPGMN WTDLITNYSP SREDNLQRVF TVASIRSMLV K |
-分子 #3: Outer capsid glycoprotein VP7
| 分子 | 名称: Outer capsid glycoprotein VP7 / タイプ: protein_or_peptide / ID: 3 / コピー数: 18 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rotavirus A (strain RVA/Monkey/United States/RRV/1975/G3P5B[3]) (ウイルス) Rotavirus A (strain RVA/Monkey/United States/RRV/1975/G3P5B[3]) (ウイルス)株: RVA/Monkey/United States/RRV/1975/G3P5B[3] |
| 分子量 | 理論値: 37.136531 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MYGIEYTTVL TFLISLILLN YILKSLTRMM DFIIYRFLFI VVILSPLLKA QNYGINLPIT GSMDTAYANS TQEETFLTST LCLYYPTEA ATEINDNSWK DTLSQLFLTK GWPTGSVYFK EYTDIASFSV DPQLYCDYNV VLMKYDATLQ LDMSELADLI L NEWLCNPM ...文字列: MYGIEYTTVL TFLISLILLN YILKSLTRMM DFIIYRFLFI VVILSPLLKA QNYGINLPIT GSMDTAYANS TQEETFLTST LCLYYPTEA ATEINDNSWK DTLSQLFLTK GWPTGSVYFK EYTDIASFSV DPQLYCDYNV VLMKYDATLQ LDMSELADLI L NEWLCNPM DITLYYYQQT DEANKWISMG SSCTIKVCPL NTQTLGIGCL TTDTATFEEV ATAEKLVITD VVDGVNHKLD VT TATCTIR NCKKLGPREN VAVIQVGGSD VLDITADPTT APQTERMMRI NWKKWWQVFY TVVDYVNQII QAMSKRSRSL NSA AFYYRI |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 18 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #5: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 5 / コピー数: 54 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.2 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 4C / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 298.15 K / 装置: GATAN CRYOPLUNGE 3 / 詳細: 4 ul sample volume, 4 sec blotting time. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3701 pixel / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 1 / 実像数: 4107 / 平均露光時間: 10.0 sec. / 平均電子線量: 33.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 1.0 µm / 倍率(補正後): 40605 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 27500 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




















 Z
Z Y
Y X
X