+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21663 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human Cohesin-NIPBL-DNA complex without STAG1 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Protein-DNA complex / ATPase / DNA-binding protein / Genome organization / Sister chromatid cohesion / Transcription regulation / CELL CYCLE / CELL CYCLE-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報eye morphogenesis / external genitalia morphogenesis / gallbladder development / SMC loading complex / Scc2-Scc4 cohesin loading complex / ear morphogenesis / mitotic cohesin loading / response to DNA damage checkpoint signaling / regulation of hair cycle / negative regulation of mitotic metaphase/anaphase transition ...eye morphogenesis / external genitalia morphogenesis / gallbladder development / SMC loading complex / Scc2-Scc4 cohesin loading complex / ear morphogenesis / mitotic cohesin loading / response to DNA damage checkpoint signaling / regulation of hair cycle / negative regulation of mitotic metaphase/anaphase transition / positive regulation of sister chromatid cohesion / meiotic cohesin complex / Cohesin Loading onto Chromatin / maintenance of mitotic sister chromatid cohesion / Establishment of Sister Chromatid Cohesion / forelimb morphogenesis / cohesin loader activity / embryonic viscerocranium morphogenesis / establishment of meiotic sister chromatid cohesion / mitotic cohesin complex / cohesin complex / negative regulation of G2/M transition of mitotic cell cycle / uterus morphogenesis / establishment of protein localization to chromatin / negative regulation of glial cell apoptotic process / regulation of developmental growth / embryonic digestive tract morphogenesis / replication-born double-strand break repair via sister chromatid exchange / cellular response to X-ray / integrator complex / lateral element / positive regulation of neuron migration / mediator complex binding / chromo shadow domain binding / establishment of mitotic sister chromatid cohesion / positive regulation of multicellular organism growth / metanephros development / chromatin looping / positive regulation of ossification / digestive tract development / reciprocal meiotic recombination / embryonic forelimb morphogenesis / lncRNA binding / sister chromatid cohesion / face morphogenesis / negative regulation of interleukin-1 beta production / microtubule motor activity / mitotic sister chromatid cohesion / stem cell population maintenance / dynein complex binding / beta-tubulin binding / fat cell differentiation / mitotic spindle pole / outflow tract morphogenesis / regulation of DNA replication / mitotic sister chromatid segregation / somatic stem cell population maintenance / positive regulation of interleukin-10 production / regulation of embryonic development / negative regulation of tumor necrosis factor production / chromosome, centromeric region / mitotic spindle assembly / developmental growth / SUMOylation of DNA damage response and repair proteins / heart morphogenesis / protein localization to chromatin / Resolution of Sister Chromatid Cohesion / Meiotic synapsis / meiotic cell cycle / condensed nuclear chromosome / chromosome segregation / promoter-specific chromatin binding / sensory perception of sound / response to radiation / protein localization / brain development / cognition / kinetochore / nuclear matrix / histone deacetylase binding / spindle pole / Separation of Sister Chromatids / transcription corepressor activity / double-strand break repair / chromosome / mitotic cell cycle / midbody / double-stranded DNA binding / DNA-binding transcription factor binding / DNA recombination / Estrogen-dependent gene expression / negative regulation of neuron apoptotic process / response to hypoxia / chromatin remodeling / protein heterodimerization activity / cell division / intracellular membrane-bounded organelle / DNA repair / apoptotic process / DNA damage response 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Shi ZB / Gao H / Bai XC / Yu H | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2020 ジャーナル: Science / 年: 2020タイトル: Cryo-EM structure of the human cohesin-NIPBL-DNA complex. 著者: Zhubing Shi / Haishan Gao / Xiao-Chen Bai / Hongtao Yu /   要旨: As a ring-shaped adenosine triphosphatase (ATPase) machine, cohesin organizes the eukaryotic genome by extruding DNA loops and mediates sister chromatid cohesion by topologically entrapping DNA. How ...As a ring-shaped adenosine triphosphatase (ATPase) machine, cohesin organizes the eukaryotic genome by extruding DNA loops and mediates sister chromatid cohesion by topologically entrapping DNA. How cohesin executes these fundamental DNA transactions is not understood. Using cryo-electron microscopy (cryo-EM), we determined the structure of human cohesin bound to its loader NIPBL and DNA at medium resolution. Cohesin and NIPBL interact extensively and together form a central tunnel to entrap a 72-base pair DNA. NIPBL and DNA promote the engagement of cohesin's ATPase head domains and ATP binding. The hinge domains of cohesin adopt an "open washer" conformation and dock onto the STAG1 subunit. Our structure explains the synergistic activation of cohesin by NIPBL and DNA and provides insight into DNA entrapment by cohesin. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21663.map.gz emd_21663.map.gz | 49.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21663-v30.xml emd-21663-v30.xml emd-21663.xml emd-21663.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
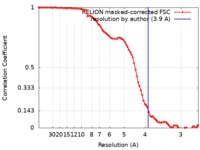
| FSC (解像度算出) |  emd_21663_fsc.xml emd_21663_fsc.xml | 8.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21663.png emd_21663.png | 136.4 KB | ||
| Filedesc metadata |  emd-21663.cif.gz emd-21663.cif.gz | 9.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21663 http://ftp.pdbj.org/pub/emdb/structures/EMD-21663 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21663 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21663 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21663_validation.pdf.gz emd_21663_validation.pdf.gz | 627.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21663_full_validation.pdf.gz emd_21663_full_validation.pdf.gz | 626.8 KB | 表示 | |
| XML形式データ |  emd_21663_validation.xml.gz emd_21663_validation.xml.gz | 10.4 KB | 表示 | |
| CIF形式データ |  emd_21663_validation.cif.gz emd_21663_validation.cif.gz | 13.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21663 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21663 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21663 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21663 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21663.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21663.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human Cohesin-NIPBL-DNA Complex without STAG1
| 全体 | 名称: Human Cohesin-NIPBL-DNA Complex without STAG1 |
|---|---|
| 要素 |
|
-超分子 #1: Human Cohesin-NIPBL-DNA Complex without STAG1
| 超分子 | 名称: Human Cohesin-NIPBL-DNA Complex without STAG1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 820 KDa |
-分子 #1: Structural maintenance of chromosomes protein 1A
| 分子 | 名称: Structural maintenance of chromosomes protein 1A / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 143.484109 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MGFLKLIEIE NFKSYKGRQI IGPFQRFTAI IGPNGSGKSN LMDAISFVLG EKTSNLRVKT LRDLIHGAPV GKPAANRAFV SMVYSEEGA EDRTFARVIV GGSSEYKINN KVVQLHEYSE ELEKLGILIK ARNFLVFQGA VESIAMKNPK ERTALFEEIS R SGELAQEY ...文字列: MGFLKLIEIE NFKSYKGRQI IGPFQRFTAI IGPNGSGKSN LMDAISFVLG EKTSNLRVKT LRDLIHGAPV GKPAANRAFV SMVYSEEGA EDRTFARVIV GGSSEYKINN KVVQLHEYSE ELEKLGILIK ARNFLVFQGA VESIAMKNPK ERTALFEEIS R SGELAQEY DKRKKEMVKA EEDTQFNYHR KKNIAAERKE AKQEKEEADR YQRLKDEVVR AQVQLQLFKL YHNEVEIEKL NK ELASKNK EIEKDKKRMD KVEDELKEKK KELGKMMREQ QQIEKEIKEK DSELNQKRPQ YIKAKENTSH KIKKLEAAKK SLQ NAQKHY KKRKGDMDEL EKEMLSVEKA RQEFEERMEE ESQSQGRDLT LEENQVKKYH RLKEEASKRA ATLAQELEKF NRDQ KADQD RLDLEERKKV ETEAKIKQKL REIEENQKRI EKLEEYITTS KQSLEEQKKL EGELTEEVEM AKRRIDEINK ELNQV MEQL GDARIDRQES SRQQRKAEIM ESIKRLYPGS VYGRLIDLCQ PTQKKYQIAV TKVLGKNMDA IIVDSEKTGR DCIQYI KEQ RGEPETFLPL DYLEVKPTDE KLRELKGAKL VIDVIRYEPP HIKKALQYAC GNALVCDNVE DARRIAFGGH QRHKTVA LD GTLFQKSGVI SGGASDLKAK ARRWDEKAVD KLKEKKERLT EELKEQMKAK RKEAELRQVQ SQAHGLQMRL KYSQSDLE Q TKTRHLALNL QEKSKLESEL ANFGPRINDI KRIIQSRERE MKDLKEKMNQ VEDEVFEEFC REIGVRNIRE FEEEKVKRQ NEIAKKRLEF ENQKTRLGIQ LDFEKNQLKE DQDKVHMWEQ TVKKDENEIE KLKKEEQRHM KIIDETMAQL QDLKNQHLAK KSEVNDKNH EMEEIRKKLG GANKEMTHLQ KEVTAIETKL EQKRSDRHNL LQACKMQDIK LPLSKGTMDD ISQEEGSSQG E DSVSGSQR ISSIYAREAL IEIDYGDLCE DLKDAQAEEE IKQEMNTLQQ KLNEQQSVLQ RIAAPNMKAM EKLESVRDKF QE TSDEFEA ARKRAKKAKQ AFEQIKKERF DRFNACFESV ATNIDEIYKA LSRNSSAQAF LGPENPEEPY LDGINYNCVA PGK RFRPMD NLSGGEKTVA ALALLFAIHS YKPAPFFVLD QIDAALDNTN IGKVANYIKE QSTCNFQAIV ISLKEEFYTK AESL IGVYP EQGDCVISKV LTFDLTKYPD ANPNPNEQ UniProtKB: Structural maintenance of chromosomes protein 1A |
-分子 #2: Structural maintenance of chromosomes protein 3
| 分子 | 名称: Structural maintenance of chromosomes protein 3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 141.770578 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MYIKQVIIQG FRSYRDQTIV DPFSSKHNVI VGRNGSGKSN FFYAIQFVLS DEFSHLRPEQ RLALLHEGTG PRVISAFVEI IFDNSDNRL PIDKEEVSLR RVIGAKKDQY FLDKKMVTKN DVMNLLESAG FSRSNPYYIV KQGKINQMAT APDSQRLKLL R EVAGTRVY ...文字列: MYIKQVIIQG FRSYRDQTIV DPFSSKHNVI VGRNGSGKSN FFYAIQFVLS DEFSHLRPEQ RLALLHEGTG PRVISAFVEI IFDNSDNRL PIDKEEVSLR RVIGAKKDQY FLDKKMVTKN DVMNLLESAG FSRSNPYYIV KQGKINQMAT APDSQRLKLL R EVAGTRVY DERKEESISL MKETEGKREK INELLKYIEE RLHTLEEEKE ELAQYQKWDK MRRALEYTIY NQELNETRAK LD ELSAKRE TSGEKSRQLR DAQQDARDKM EDIERQVREL KTKISAMKEE KEQLSAERQE QIKQRTKLEL KAKDLQDELA GNS EQRKRL LKERQKLLEK IEEKQKELAE TEPKFNSVKE KEERGIARLA QATQERTDLY AKQGRGSQFT SKEERDKWIK KELK SLDQA INDKKRQIAA IHKDLEDTEA NKEKNLEQYN KLDQDLNEVK ARVEELDRKY YEVKNKKDEL QSERNYLWRE ENAEQ QALA AKREDLEKKQ QLLRAATGKA ILNGIDSINK VLDHFRRKGI NQHVQNGYHG IVMNNFECEP AFYTCVEVTA GNRLFY HIV DSDEVSTKIL MEFNKMNLPG EVTFLPLNKL DVRDTAYPET NDAIPMISKL RYNPRFDKAF KHVFGKTLIC RSMEVST QL ARAFTMDCIT LEGDQVSHRG ALTGGYYDTR KSRLELQKDV RKAEEELGEL EAKLNENLRR NIERINNEID QLMNQMQQ I ETQQRKFKAS RDSILSEMKM LKEKRQQSEK TFMPKQRSLQ SLEASLHAME STRESLKAEL GTDLLSQLSL EDQKRVDAL NDEIRQLQQE NRQLLNERIK LEGIITRVET YLNENLRKRL DQVEQELNEL RETEGGTVLT ATTSELEAIN KRVKDTMARS EDLDNSIDK TEAGIKELQK SMERWKNMEK EHMDAINHDT KELEKMTNRQ GMLLKKKEEC MKKIRELGSL PQEAFEKYQT L SLKQLFRK LEQCNTELKK YSHVNKKALD QFVNFSEQKE KLIKRQEELD RGYKSIMELM NVLELRKYEA IQLTFKQVSK NF SEVFQKL VPGGKATLVM KKGDVEGSQS QDEGEGSGES ERGSGSQSSV PSVDQFTGVG IRVSFTGKQG EMREMQQLSG GQK SLVALA LIFAIQKCDP APFYLFDQID QALDAQHRKA VSDMIMELAV HAQFITTTFR PELLESADKF YGVKFRNKVS HIDV ITAEM AKDFVEDDTT HG UniProtKB: Structural maintenance of chromosomes protein 3 |
-分子 #3: Double-strand-break repair protein rad21 homolog
| 分子 | 名称: Double-strand-break repair protein rad21 homolog / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 71.556102 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MFYAHFVLSK RGPLAKIWLA AHWDKKLTKA HVFECNLESS VESIISPKVK MALRTSGHLL LGVVRIYHRK AKYLLADCNE AFIKIKMAF RPGVVDLPEE NREAAYNAIT LPEEFHDFDQ PLPDLDDIDV AQQFSLNQSR VEEITMREEV GNISILQEND F GDFGMDDR ...文字列: MFYAHFVLSK RGPLAKIWLA AHWDKKLTKA HVFECNLESS VESIISPKVK MALRTSGHLL LGVVRIYHRK AKYLLADCNE AFIKIKMAF RPGVVDLPEE NREAAYNAIT LPEEFHDFDQ PLPDLDDIDV AQQFSLNQSR VEEITMREEV GNISILQEND F GDFGMDDR EIMAEGSAFE DDDMLVSTTT SNLLLESEQS TSNLNEKINH LEYEDQYKDD NFGEGNDGGI LDDKLISNND GG IFDDPPA LSEAGVMLPE QPAHDDMDED DNVSMGGPDS PASVDPVEPM PTMTDQTTLV PNEEEAFALE PIDITVKETK AKR KRKLIV DSVKELDSKT IRAQLSDYSD IVTTLDLAPP TKKLMMWKET GGVEKLFSLP AQPLWNNRLL KLFTRCLTPL VPED LRKRR KGGEADNLDE FLKEFENPEV PREDQQQQHQ QRDVIDEPII EEPSALQESV MEASRTNIDE SAMPPPPPQG VKRKA GQID PEPVMPPQQV EQMEIPPVEL PPEEPPNICQ LIPELELLPE KEKEKEKEKE DDEEEEDEDA SGGDQDQEER RWNKRT QQM LHGLQRALAK TGAESISLLE LCRNTNRKQA AAKFYSFLVL KKQQAIELTQ EEPYSDIIAT PGPRFHII UniProtKB: Double-strand-break repair protein rad21 homolog |
-分子 #4: Nipped-B-like protein
| 分子 | 名称: Nipped-B-like protein / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 186.77825 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: PSLSEVARKM KKKEKQKKRK AYEPKLTPEE MMDSSTFKRF TASIENILDN LEDMDFTAFG DDDEIPQELL LGKHQLNELG SESAKIKAM GIMDKLSTDK TVKVLNILEK NIQDGSKLST LLNHNNDTEE EERLWRDLIM ERVTKSADAC LTTINIMTSP N MPKAVYIE ...文字列: PSLSEVARKM KKKEKQKKRK AYEPKLTPEE MMDSSTFKRF TASIENILDN LEDMDFTAFG DDDEIPQELL LGKHQLNELG SESAKIKAM GIMDKLSTDK TVKVLNILEK NIQDGSKLST LLNHNNDTEE EERLWRDLIM ERVTKSADAC LTTINIMTSP N MPKAVYIE DVIERVIQYT KFHLQNTLYP QYDPVYRLDP HGGGLLSSKA KRAKCSTHKQ RVIVMLYNKV CDIVSSLSEL LE IQLLTDT TILQVSSMGI TPFFVENVSE LQLCAIKLVT AVFSRYEKHR QLILEEIFTS LARLPTSKRS LRNFRLNSSD MDG EPMYIQ MVTALVLQLI QCVVHLPSSE KDSNAEEDSN KKIDQDVVIT NSYETAMRTA QNFLSIFLKK CGSKQGEEDY RPLF ENFVQ DLLSTVNKPE WPAAELLLSL LGRLLVHQFS NKSTEMALRV ASLDYLGTVA ARLRKDAVTS KMDQGSIERI LKQVS GGED EIQQLQKALL DYLDENTETD PSLVFSRKFY IAQWFRDTTL ETEKAMKSQK DEESSEGTHH AKEIETTGQI MHRAEN RKK FLRSIIKTTP SQFSTLKMNS DTVDYDDACL IVRYLASMRP FAQSFDIYLT QILRVLGENA IAVRTKAMKC LSEVVAV DP SILARLDMQR GVHGRLMDNS TSVREAAVEL LGRFVLCRPQ LAEQYYDMLI ERILDTGISV RKRVIKILRD ICIEQPTF P KITEMCVKMI RRVNDEEGIK KLVNETFQKL WFTPTPHNDK EAMTRKILNI TDVVAACRDT GYDWFEQLLQ NLLKSEEDS SYKPVKKACT QLVDNLVEHI LKYEESLADS DNKGVNSGRL VACITTLFLF SKIRPQLMVK HAMTMQPYLT TKCSTQNDFM VICNVAKIL ELVVPLMEHP SETFLATIEE DLMKLIIKYG MTVVQHCVSC LGAVVNKVTQ NFKFVWACFN RYYGAISKLK S QHQEDPNN TSLLTNKPAL LRSLFTVGAL CRHFDFDLED FKGNSKVNIK DKVLELLMYF TKHSDEEVQT KAIIGLGFAF IQ HPSLMFE QEVKNLYNNI LSDKNSSVNL KIQVLKNLQT YLQEEDTRMQ QADRDWKKVA KQEDLKEMGD VSSGMSSSIM QLY LKQVLE AFFHTQSSVR HFALNVIALT LNQGLIHPVQ CVPYLIAMGT DPEPAMRNKA DQQLVEIDKK YAGFIHMKAV AGMK MSYQV QQAINTCLKD PVRGFRQDES SSALCSHLYS MIRGNRQHRR AFLISLLNLF DDTAKTDVTM LLYIADNLAC FPYQT QEEP LFIMHHIDIT LSVSGSNLLQ SFKESMVKDK RKERKSSPSK ENESSDSEEE VSRPRKSRKR VDSDSDSDSE DDINSV MKC LPENSAPLIE FANVSQGILL LLMLKQHLKN LCGFSDSKIQ KYSPSESAKV YDKAINRKTG VHFHPKQTLD FLRSDMA NS KITEEVKRSI VKQYLDFKLL MEHLDPDEEE EEGEVSASTN ARNKAITSLL GGGSPKNNTA AETEDDESDG EDRGGGTS G SLRRSKRNSD STELAAQMNE SVDVMDVIAI CCPKYKDRPQ IARVVQKTSS GFSVQWMAGS YSGSWTEAKR RDGRKLVPW VDTIKESDII YKKIALTSAN KLTNKVVQTL RSLYAAKDGT SS UniProtKB: Nipped-B-like protein |
-分子 #5: DNA (43-MER)
| 分子 | 名称: DNA (43-MER) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.422931 KDa |
| 配列 | 文字列: (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA) (DA)(DA)(DA) |
-分子 #6: DNA (43-MER)
| 分子 | 名称: DNA (43-MER) / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.035333 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT) (DT)(DT)(DT) |
-分子 #7: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 7 / コピー数: 2 / 式: ANP |
|---|---|
| 分子量 | 理論値: 506.196 Da |
| Chemical component information |  ChemComp-ANP: |
-分子 #8: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 8 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 5796 / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-6wge: |
 ムービー
ムービー コントローラー
コントローラー