+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20202 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Asymmetric focsued reconstruction of human norovirus GII.2 Snow Mountain Virus strain VLP asymmetric unit in T=1 symmetry | |||||||||
 マップデータ マップデータ | Asymmetric focused reconstruction of human norovirus GII.2 Snow Mountain Virus strain VLP asymmetric unit in T=1 symmetry | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Caliciviridae / Norovirus / GII.2 / Snow Mountain Virus / VIRUS LIKE PARTICLE | |||||||||
| 機能・相同性 | Calicivirus coat protein C-terminal / Calicivirus coat protein C-terminal / Calicivirus coat protein / Calicivirus coat protein / Picornavirus/Calicivirus coat protein / Viral coat protein subunit / Viral protein 1 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Snow Mountain virus (ウイルス) Snow Mountain virus (ウイルス) | |||||||||
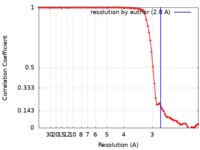
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Jung J / Grant T | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2019 ジャーナル: Proc Natl Acad Sci U S A / 年: 2019タイトル: High-resolution cryo-EM structures of outbreak strain human norovirus shells reveal size variations. 著者: James Jung / Timothy Grant / Dennis R Thomas / Chris W Diehnelt / Nikolaus Grigorieff / Leemor Joshua-Tor /  要旨: Noroviruses are a leading cause of foodborne illnesses worldwide. Although GII.4 strains have been responsible for most norovirus outbreaks, the assembled virus shell structures have been available ...Noroviruses are a leading cause of foodborne illnesses worldwide. Although GII.4 strains have been responsible for most norovirus outbreaks, the assembled virus shell structures have been available in detail for only a single strain (GI.1). We present high-resolution (2.6- to 4.1-Å) cryoelectron microscopy (cryo-EM) structures of GII.4, GII.2, GI.7, and GI.1 human norovirus outbreak strain virus-like particles (VLPs). Although norovirus VLPs have been thought to exist in a single-sized assembly, our structures reveal polymorphism between and within genogroups, with small, medium, and large particle sizes observed. Using asymmetric reconstruction, we were able to resolve a Zn metal ion adjacent to the coreceptor binding site, which affected the structural stability of the shell. Our structures serve as valuable templates for facilitating vaccine formulations. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20202.map.gz emd_20202.map.gz | 365.3 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20202-v30.xml emd-20202-v30.xml emd-20202.xml emd-20202.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20202_fsc.xml emd_20202_fsc.xml | 9.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20202.png emd_20202.png | 138 KB | ||
| Filedesc metadata |  emd-20202.cif.gz emd-20202.cif.gz | 6.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20202 http://ftp.pdbj.org/pub/emdb/structures/EMD-20202 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20202 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20202 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20202_validation.pdf.gz emd_20202_validation.pdf.gz | 361.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20202_full_validation.pdf.gz emd_20202_full_validation.pdf.gz | 361 KB | 表示 | |
| XML形式データ |  emd_20202_validation.xml.gz emd_20202_validation.xml.gz | 8.6 KB | 表示 | |
| CIF形式データ |  emd_20202_validation.cif.gz emd_20202_validation.cif.gz | 11.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20202 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20202 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20202 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20202 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20202.map.gz / 形式: CCP4 / 大きさ: 10 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20202.map.gz / 形式: CCP4 / 大きさ: 10 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Asymmetric focused reconstruction of human norovirus GII.2 Snow Mountain Virus strain VLP asymmetric unit in T=1 symmetry | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Snow Mountain virus
| 全体 | 名称:  Snow Mountain virus (ウイルス) Snow Mountain virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Snow Mountain virus
| 超分子 | 名称: Snow Mountain virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1 / NCBI-ID: 52276 / 生物種: Snow Mountain virus / Sci species strain: GII.2 / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 3.56 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: VP1 / 直径: 310.0 Å / T番号(三角分割数): 1 |
-分子 #1: Viral protein 1
| 分子 | 名称: Viral protein 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Snow Mountain virus (ウイルス) Snow Mountain virus (ウイルス) |
| 分子量 | 理論値: 59.321809 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKMASNDAAP STDGAAGLVP ESNNEVMALE PVAGAALAAP VTGQTNIIDP WIRANFVQAP NGEFTVSPRN APGEVLLNLE LGPELNPYL AHLARMYNGY AGGMEVQVML AGNAFTAGKL VFAAVPPHFP VENLSPQQIT MFPHVIIDVR TLEPVLLPLP D VRNNFFHY ...文字列: MKMASNDAAP STDGAAGLVP ESNNEVMALE PVAGAALAAP VTGQTNIIDP WIRANFVQAP NGEFTVSPRN APGEVLLNLE LGPELNPYL AHLARMYNGY AGGMEVQVML AGNAFTAGKL VFAAVPPHFP VENLSPQQIT MFPHVIIDVR TLEPVLLPLP D VRNNFFHY NQKDDPKMRI VAMLYTPLRS NGSGDDVFTV SCRVLTRPSP DFDFTYLVPP TVESKTKPFT LPILTLGELS NS RFPVSID QMYTSPNEVI SVQCQNGRCT LDGELQGTTQ LQVSGICAFK GEVTAHLQDN DHLYNITITN LNGSPFDPSE DIP APLGVP DFQGRVFGVI TQRDKQNAAG QSQPANRGHD AVVPTYTAQY TPKLGQVQIG TWQTDDLKVN QPVKFTPVGL NDTE HFNQW VVPRYAGALN LNTNLAPSVA PVFPGERLLF FRSYLPLKGG YGNPAIDCLL PQEWVQHFYQ EAAPSMSEVA LVRYI NPDT GRALFEAKLH RAGFMTVSSN TSAPVVVPAN GYFRFDSWVN QFYSLAPMGT GNGRRRIQ UniProtKB: Viral protein 1 |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 124 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL |
|---|---|
| 緩衝液 | pH: 5.75 |
| グリッド | 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 前処理 - タイプ: GLOW DISCHARGE / 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 295 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 実像数: 2809 / 平均露光時間: 7.0 sec. / 平均電子線量: 69.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 225-532 / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-6ouc: |
 ムービー
ムービー コントローラー
コントローラー


























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)