+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12309 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Trimeric efflux pump Klebsiella TolC in complex with KlebC | |||||||||
 マップデータ マップデータ | LocScale sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Efflux pump / trimer / complex / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 | Type I secretion outer membrane protein, TolC / : / Outer membrane efflux protein / Outer membrane efflux protein / efflux transmembrane transporter activity / cell outer membrane / Outer membrane channel protein TolC 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Klebsiella quasipneumoniae (バクテリア) Klebsiella quasipneumoniae (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Webby MN / Housden NG | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Toxin import through the antibiotic efflux channel TolC. 著者: Nicholas G Housden / Melissa N Webby / Edward D Lowe / Tarick J El-Baba / Renata Kaminska / Christina Redfield / Carol V Robinson / Colin Kleanthous /  要旨: Bacteria often secrete diffusible protein toxins (bacteriocins) to kill bystander cells during interbacterial competition. Here, we use biochemical, biophysical and structural analyses to show how a ...Bacteria often secrete diffusible protein toxins (bacteriocins) to kill bystander cells during interbacterial competition. Here, we use biochemical, biophysical and structural analyses to show how a bacteriocin exploits TolC, a major outer-membrane antibiotic efflux channel in Gram-negative bacteria, to transport itself across the outer membrane of target cells. Klebicin C (KlebC), a rRNase toxin produced by Klebsiella pneumoniae, binds TolC of a related species (K. quasipneumoniae) with high affinity through an N-terminal, elongated helical hairpin domain common amongst bacteriocins. The KlebC helical hairpin opens like a switchblade to bind TolC. A cryo-EM structure of this partially translocated state, at 3.1 Å resolution, reveals that KlebC associates along the length of the TolC channel. Thereafter, the unstructured N-terminus of KlebC protrudes beyond the TolC iris, presenting a TonB-box sequence to the periplasm. Association with proton-motive force-linked TonB in the inner membrane drives toxin import through the channel. Finally, we demonstrate that KlebC binding to TolC blocks drug efflux from bacteria. Our results indicate that TolC, in addition to its known role in antibiotic export, can function as a protein import channel for bacteriocins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12309.map.gz emd_12309.map.gz | 50 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12309-v30.xml emd-12309-v30.xml emd-12309.xml emd-12309.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12309_fsc.xml emd_12309_fsc.xml | 13.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12309.png emd_12309.png | 39.7 KB | ||
| Filedesc metadata |  emd-12309.cif.gz emd-12309.cif.gz | 5.8 KB | ||
| その他 |  emd_12309_additional_1.map.gz emd_12309_additional_1.map.gz emd_12309_additional_2.map.gz emd_12309_additional_2.map.gz emd_12309_additional_3.map.gz emd_12309_additional_3.map.gz | 57.2 MB 80.6 MB 9.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12309 http://ftp.pdbj.org/pub/emdb/structures/EMD-12309 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12309 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12309 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12309_validation.pdf.gz emd_12309_validation.pdf.gz | 439 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12309_full_validation.pdf.gz emd_12309_full_validation.pdf.gz | 438.6 KB | 表示 | |
| XML形式データ |  emd_12309_validation.xml.gz emd_12309_validation.xml.gz | 11.8 KB | 表示 | |
| CIF形式データ |  emd_12309_validation.cif.gz emd_12309_validation.cif.gz | 15.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12309 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12309 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12309 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12309 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12309.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12309.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | LocScale sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.822 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Sharpened map from relion locres job. Requires Zflip
| ファイル | emd_12309_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map from relion locres job. Requires Zflip | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
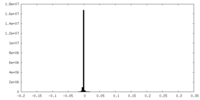
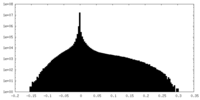
| 密度ヒストグラム |
-追加マップ: Unsharpened map from relion refine3D job. Requires zflip
| ファイル | emd_12309_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map from relion refine3D job. Requires zflip | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
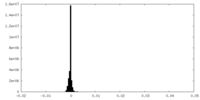
| 密度ヒストグラム |
-追加マップ: Postprocess masked map from relion. Requires Zflip
| ファイル | emd_12309_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Postprocess masked map from relion. Requires Zflip | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
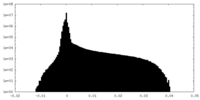
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homotrimer of TolC from klebsiella in complex with KlebC
| 全体 | 名称: Homotrimer of TolC from klebsiella in complex with KlebC |
|---|---|
| 要素 |
|
-超分子 #1: Homotrimer of TolC from klebsiella in complex with KlebC
| 超分子 | 名称: Homotrimer of TolC from klebsiella in complex with KlebC タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Klebsiella quasipneumoniae (バクテリア) Klebsiella quasipneumoniae (バクテリア) |
-分子 #1: Outer membrane channel protein
| 分子 | 名称: Outer membrane channel protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Klebsiella quasipneumoniae (バクテリア) Klebsiella quasipneumoniae (バクテリア) |
| 分子量 | 理論値: 54.058676 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKKLLPILIG LSLTGFSAMS QAENLLQVYQ QARISNPDLR KSAADRDAAF EKINEARSPL LPQLGLGADY TYTSGFRDYK DQNSNVTSG SLQLTQVLFD MSKWRALTLQ EKAAGIQDVT YQTDQQTLIL NTATAYFKVL AAIDTLSYTE AQKQAIYRQL D QTTQRFNV ...文字列: MKKLLPILIG LSLTGFSAMS QAENLLQVYQ QARISNPDLR KSAADRDAAF EKINEARSPL LPQLGLGADY TYTSGFRDYK DQNSNVTSG SLQLTQVLFD MSKWRALTLQ EKAAGIQDVT YQTDQQTLIL NTATAYFKVL AAIDTLSYTE AQKQAIYRQL D QTTQRFNV GLVAITDVQN ARSQYDAVLA NEVTARNDLD NAVEELRQVT GNYYPELASL NVDGFKTSKP QAVNALLKEA EN RNLSLLQ ARLNQDLARE QIRQAQDGHL PTLDLNASSG VSNNRYSGSK SISQDADIGQ NKIGLSFSLP LYQGGMVNSQ VKQ AQYNFV GASEQLESAH RSVVQTVRSS FNNVNASISS INAYKQAVVS AQSSLDAMEA GYSVGTRTIV DVLDATTTLY NAKQ QLSNA RYNYLINELN IKSALGTLNE QDLIALNNTL GKPISTSADS VAPENPQQDA TADGYGNTTA AMKPASARTT THSSG SNPF RQLEHHHHHH UniProtKB: Outer membrane channel protein TolC |
-分子 #2: Klebicin C activity
| 分子 | 名称: Klebicin C activity / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Klebsiella quasipneumoniae (バクテリア) Klebsiella quasipneumoniae (バクテリア) |
| 分子量 | 理論値: 28.629162 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MADNQPVPLT PAPPGMVSLG VNENGEEEMT VIGGDGSGTG FSGNEAPIIP GSGSLQADLG KKSLTRLQAE SSAAIHATAK WTTENLAKT QAAQAERAKA AMLSQQAAKA KQAKLTQHLK DVVDRALQNN KTRPTVIDLA HQNNQQMAAM AEFIGRQKAI E EARKKAER ...文字列: MADNQPVPLT PAPPGMVSLG VNENGEEEMT VIGGDGSGTG FSGNEAPIIP GSGSLQADLG KKSLTRLQAE SSAAIHATAK WTTENLAKT QAAQAERAKA AMLSQQAAKA KQAKLTQHLK DVVDRALQNN KTRPTVIDLA HQNNQQMAAM AEFIGRQKAI E EARKKAER EAKRAEEAYQ AALRAQEEEQ RKQAEIERKL QEARKQEAAA KAKAEADRIA AEKAEAEARA KAEAERRKAE EA RKALFAK AGIKDTPGCL EHHHHHH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.9 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)