+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11720 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Class D GPCR Ste2 dimer coupled to two G proteins | |||||||||
 マップデータ マップデータ | Map1 | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mating projection / : / protein localization to mating projection tip / PLC beta mediated events / G-protein activation / Acetylcholine regulates insulin secretion / G alpha (q) signalling events / ADP signalling through P2Y purinoceptor 1 / Fatty Acids bound to GPR40 (FFAR1) regulate insulin secretion / : ...mating projection / : / protein localization to mating projection tip / PLC beta mediated events / G-protein activation / Acetylcholine regulates insulin secretion / G alpha (q) signalling events / ADP signalling through P2Y purinoceptor 1 / Fatty Acids bound to GPR40 (FFAR1) regulate insulin secretion / : / G-protein beta/gamma-subunit complex / adaptation of signaling pathway by response to pheromone involved in conjugation with cellular fusion / : / G alpha (12/13) signalling events / chemotropism / Cdc24p-Far1p-Gbetagamma complex / mating pheromone activity / mating-type factor pheromone receptor activity / nuclear migration involved in conjugation with cellular fusion / mating / G protein-coupled receptor homodimeric complex / Cooperation of PDCL (PhLP1) and TRiC/CCT in G-protein beta folding / karyogamy involved in conjugation with cellular fusion / G-protein gamma-subunit binding / establishment of protein localization to plasma membrane / pheromone-dependent signal transduction involved in conjugation with cellular fusion / invasive growth in response to glucose limitation / pheromone binding / cupric ion binding / G-protein alpha-subunit binding / cell periphery / G protein-coupled receptor binding / G-protein beta/gamma-subunit complex binding / small GTPase binding / G-protein beta-subunit binding / heterotrimeric G-protein complex / scaffold protein binding / endosome membrane / endosome / G protein-coupled receptor signaling pathway / GTPase activity / GTP binding / protein kinase binding / signal transduction / extracellular region / metal ion binding / plasma membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
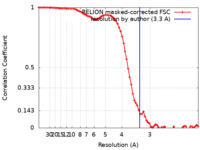
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Velazhahan V / Tate C | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: Structure of the class D GPCR Ste2 dimer coupled to two G proteins. 著者: Vaithish Velazhahan / Ning Ma / Gáspár Pándy-Szekeres / Albert J Kooistra / Yang Lee / David E Gloriam / Nagarajan Vaidehi / Christopher G Tate /     要旨: G-protein-coupled receptors (GPCRs) are divided phylogenetically into six classes, denoted A to F. More than 370 structures of vertebrate GPCRs (belonging to classes A, B, C and F) have been ...G-protein-coupled receptors (GPCRs) are divided phylogenetically into six classes, denoted A to F. More than 370 structures of vertebrate GPCRs (belonging to classes A, B, C and F) have been determined, leading to a substantial understanding of their function. By contrast, there are no structures of class D GPCRs, which are found exclusively in fungi where they regulate survival and reproduction. Here we determine the structure of a class D GPCR, the Saccharomyces cerevisiae pheromone receptor Ste2, in an active state coupled to the heterotrimeric G protein Gpa1-Ste4-Ste18. Ste2 was purified as a homodimer coupled to two G proteins. The dimer interface of Ste2 is formed by the N terminus, the transmembrane helices H1, H2 and H7, and the first extracellular loop ECL1. We establish a class D1 generic residue numbering system (CD1) to enable comparisons with orthologues and with other GPCR classes. The structure of Ste2 bears similarities in overall topology to class A GPCRs, but the transmembrane helix H4 is shifted by more than 20 Å and the G-protein-binding site is a shallow groove rather than a cleft. The structure provides a template for the design of novel drugs to target fungal GPCRs, which could be used to treat numerous intractable fungal diseases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
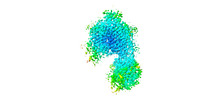
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11720.map.gz emd_11720.map.gz | 3.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11720-v30.xml emd-11720-v30.xml emd-11720.xml emd-11720.xml | 39.6 KB 39.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11720_fsc.xml emd_11720_fsc.xml | 7.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11720.png emd_11720.png | 124.3 KB | ||
| その他 |  emd_11720_additional_1.map.gz emd_11720_additional_1.map.gz emd_11720_half_map_1.map.gz emd_11720_half_map_1.map.gz emd_11720_half_map_2.map.gz emd_11720_half_map_2.map.gz | 2.2 MB 27.2 MB 27.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11720 http://ftp.pdbj.org/pub/emdb/structures/EMD-11720 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11720 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11720 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11720_validation.pdf.gz emd_11720_validation.pdf.gz | 399.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11720_full_validation.pdf.gz emd_11720_full_validation.pdf.gz | 398.8 KB | 表示 | |
| XML形式データ |  emd_11720_validation.xml.gz emd_11720_validation.xml.gz | 13 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11720 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11720 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11720 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11720 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ad3MC  7qa8C  7qb9C  7qbcC  7qbiC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10550 (タイトル: Structure of the class D GPCR Ste2 dimer coupled to two G proteins EMPIAR-10550 (タイトル: Structure of the class D GPCR Ste2 dimer coupled to two G proteinsData size: 6.4 TB / Data #1: LMB Krios1 Movies [micrographs - multiframe] / Data #2: LMB Krios2 Movies [micrographs - multiframe] / Data #3: eBic Krios1 Movies [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11720.map.gz / 形式: CCP4 / 大きさ: 35.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11720.map.gz / 形式: CCP4 / 大きさ: 35.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.047 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Map2
| ファイル | emd_11720_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
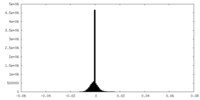
| 密度ヒストグラム |
-ハーフマップ: Halfmap1 for calculating map1
| ファイル | emd_11720_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap1 for calculating map1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
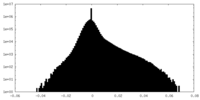
| 密度ヒストグラム |
-ハーフマップ: Halfmap2 for calculating map1
| ファイル | emd_11720_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap2 for calculating map1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
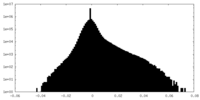
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Ste2 dimer coupled to two G proteins
+超分子 #1: Ste2 dimer coupled to two G proteins
+超分子 #2: Ste2 dimer coupled to two G proteins
+超分子 #3: Alpha-factor mating pheromone
+超分子 #4: Guanine nucleotide-binding protein alpha-1 subunit,Guanine nucleo...
+分子 #1: Pheromone alpha factor receptor
+分子 #2: Alpha-factor mating pheromone
+分子 #3: STE4 isoform 1
+分子 #4: Guanine nucleotide-binding protein alpha-1 subunit,Guanine nucleo...
+分子 #5: Guanine nucleotide-binding protein subunit gamma
+分子 #6: 2-acetamido-2-deoxy-beta-D-glucopyranose
+分子 #7: CHOLESTEROL HEMISUCCINATE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Solutions were made fresh and filtered | ||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV | ||||||||||||||||||
| 詳細 | The sample was purified as a monodisperse complex |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 温度 | 最低: 70.0 K / 最高: 70.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum SE / エネルギーフィルター - スリット幅: 20 eV |
| 詳細 | eBIC Krios1 |
| 撮影 | Image recording ID: 1 フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 2.7 µm / 最小 デフォーカス(補正後): 0.9 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 電子顕微鏡法 #1~
電子顕微鏡法 #1~
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 温度 | 最低: 70.0 K / 最高: 70.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum SE / エネルギーフィルター - スリット幅: 20 eV |
| 詳細 | LMB Krios1 |
| 撮影 | Image recording ID: 2 フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 2.7 µm / 最小 デフォーカス(補正後): 0.9 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 電子顕微鏡法 #1~~
電子顕微鏡法 #1~~
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 温度 | 最低: 70.0 K / 最高: 70.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum SE / エネルギーフィルター - スリット幅: 20 eV |
| 詳細 | LMB Krios 2 |
| 撮影 | Image recording ID: 3 フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 2.7 µm / 最小 デフォーカス(補正後): 0.9 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー























 Z
Z Y
Y X
X




















 Trichoplusia ni (イラクサキンウワバ)
Trichoplusia ni (イラクサキンウワバ)