+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-11720 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Class D GPCR Ste2 dimer coupled to two G proteins | |||||||||
 Map data Map data | Map1 | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Fungal GPCR / Dimer / Complex / Class D / Active State / MEMBRANE PROTEIN | |||||||||
| Function / homology |  Function and homology information Function and homology informationprotein localization to mating projection tip / PLC beta mediated events / : / Acetylcholine regulates insulin secretion / G alpha (q) signalling events / ADP signalling through P2Y purinoceptor 1 / : / mating projection / G-protein beta/gamma-subunit complex / regulation of pheromone-dependent signal transduction involved in conjugation with cellular fusion ...protein localization to mating projection tip / PLC beta mediated events / : / Acetylcholine regulates insulin secretion / G alpha (q) signalling events / ADP signalling through P2Y purinoceptor 1 / : / mating projection / G-protein beta/gamma-subunit complex / regulation of pheromone-dependent signal transduction involved in conjugation with cellular fusion / chemotropism / Cdc24p-Far1p-Gbetagamma complex / G alpha (12/13) signalling events / CDC42 GTPase cycle / mating pheromone activity / mating-type factor pheromone receptor activity / High laminar flow shear stress activates signaling by PIEZO1 and PECAM1:CDH5:KDR in endothelial cells / nuclear migration involved in conjugation with cellular fusion / mating / G protein-coupled receptor homodimeric complex / response to pheromone triggering conjugation with cellular fusion / karyogamy involved in conjugation with cellular fusion / regulation of carbohydrate metabolic process / Cooperation of PDCL (PhLP1) and TRiC/CCT in G-protein beta folding / G-protein gamma-subunit binding / establishment of protein localization to plasma membrane / pheromone-dependent signal transduction involved in conjugation with cellular fusion / invasive growth in response to glucose limitation / cupric ion binding / G-protein alpha-subunit binding / cell periphery / G protein-coupled receptor binding / small GTPase binding / G-protein beta/gamma-subunit complex binding / G-protein beta-subunit binding / heterotrimeric G-protein complex / signaling receptor complex adaptor activity / scaffold protein binding / cell cortex / endosome / endosome membrane / G protein-coupled receptor signaling pathway / GTPase activity / protein kinase binding / GTP binding / signal transduction / extracellular region / metal ion binding / plasma membrane / cytosol / cytoplasm Similarity search - Function | |||||||||
| Biological species |   | |||||||||
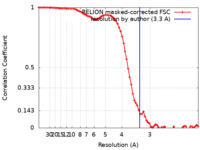
| Method | single particle reconstruction / cryo EM / Resolution: 3.3 Å | |||||||||
 Authors Authors | Velazhahan V / Tate C | |||||||||
| Funding support |  United Kingdom, 2 items United Kingdom, 2 items
| |||||||||
 Citation Citation |  Journal: Nature / Year: 2021 Journal: Nature / Year: 2021Title: Structure of the class D GPCR Ste2 dimer coupled to two G proteins. Authors: Vaithish Velazhahan / Ning Ma / Gáspár Pándy-Szekeres / Albert J Kooistra / Yang Lee / David E Gloriam / Nagarajan Vaidehi / Christopher G Tate /     Abstract: G-protein-coupled receptors (GPCRs) are divided phylogenetically into six classes, denoted A to F. More than 370 structures of vertebrate GPCRs (belonging to classes A, B, C and F) have been ...G-protein-coupled receptors (GPCRs) are divided phylogenetically into six classes, denoted A to F. More than 370 structures of vertebrate GPCRs (belonging to classes A, B, C and F) have been determined, leading to a substantial understanding of their function. By contrast, there are no structures of class D GPCRs, which are found exclusively in fungi where they regulate survival and reproduction. Here we determine the structure of a class D GPCR, the Saccharomyces cerevisiae pheromone receptor Ste2, in an active state coupled to the heterotrimeric G protein Gpa1-Ste4-Ste18. Ste2 was purified as a homodimer coupled to two G proteins. The dimer interface of Ste2 is formed by the N terminus, the transmembrane helices H1, H2 and H7, and the first extracellular loop ECL1. We establish a class D1 generic residue numbering system (CD1) to enable comparisons with orthologues and with other GPCR classes. The structure of Ste2 bears similarities in overall topology to class A GPCRs, but the transmembrane helix H4 is shifted by more than 20 Å and the G-protein-binding site is a shallow groove rather than a cleft. The structure provides a template for the design of novel drugs to target fungal GPCRs, which could be used to treat numerous intractable fungal diseases. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_11720.map.gz emd_11720.map.gz | 3.4 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-11720-v30.xml emd-11720-v30.xml emd-11720.xml emd-11720.xml | 39.8 KB 39.8 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_11720_fsc.xml emd_11720_fsc.xml | 7.5 KB | Display |  FSC data file FSC data file |
| Images |  emd_11720.png emd_11720.png | 124.3 KB | ||
| Filedesc metadata |  emd-11720.cif.gz emd-11720.cif.gz | 8 KB | ||
| Others |  emd_11720_additional_1.map.gz emd_11720_additional_1.map.gz emd_11720_half_map_1.map.gz emd_11720_half_map_1.map.gz emd_11720_half_map_2.map.gz emd_11720_half_map_2.map.gz | 2.2 MB 27.2 MB 27.2 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11720 http://ftp.pdbj.org/pub/emdb/structures/EMD-11720 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11720 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11720 | HTTPS FTP |
-Related structure data
| Related structure data |  7ad3MC  7qa8C  7qb9C  7qbcC  7qbiC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | |
| EM raw data |  EMPIAR-10550 (Title: Structure of the class D GPCR Ste2 dimer coupled to two G proteins EMPIAR-10550 (Title: Structure of the class D GPCR Ste2 dimer coupled to two G proteinsData size: 6.4 TB / Data #1: LMB Krios1 Movies [micrographs - multiframe] / Data #2: LMB Krios2 Movies [micrographs - multiframe] / Data #3: eBic Krios1 Movies [micrographs - multiframe]) |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_11720.map.gz / Format: CCP4 / Size: 35.3 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_11720.map.gz / Format: CCP4 / Size: 35.3 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Map1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.047 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
-Additional map: Map2
| File | emd_11720_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Map2 | ||||||||||||
| Projections & Slices |
| ||||||||||||
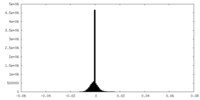
| Density Histograms |
-Half map: Halfmap1 for calculating map1
| File | emd_11720_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Halfmap1 for calculating map1 | ||||||||||||
| Projections & Slices |
| ||||||||||||
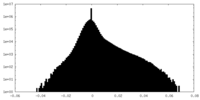
| Density Histograms |
-Half map: Halfmap2 for calculating map1
| File | emd_11720_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Halfmap2 for calculating map1 | ||||||||||||
| Projections & Slices |
| ||||||||||||
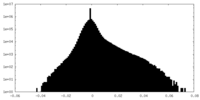
| Density Histograms |
- Sample components
Sample components
+Entire : Ste2 dimer coupled to two G proteins
+Supramolecule #1: Ste2 dimer coupled to two G proteins
+Supramolecule #2: Ste2 dimer coupled to two G proteins
+Supramolecule #3: Alpha-factor mating pheromone
+Supramolecule #4: Guanine nucleotide-binding protein alpha-1 subunit,Guanine nucleo...
+Macromolecule #1: Pheromone alpha factor receptor
+Macromolecule #2: Alpha-factor mating pheromone
+Macromolecule #3: STE4 isoform 1
+Macromolecule #4: Guanine nucleotide-binding protein alpha-1 subunit,Guanine nucleo...
+Macromolecule #5: Guanine nucleotide-binding protein subunit gamma
+Macromolecule #6: 2-acetamido-2-deoxy-beta-D-glucopyranose
+Macromolecule #7: CHOLESTEROL HEMISUCCINATE
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 0.7 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Buffer | pH: 7.5 Component:
Details: Solutions were made fresh and filtered | ||||||||||||||||||
| Grid | Model: Quantifoil R1.2/1.3 / Material: GOLD / Mesh: 300 / Support film - Material: CARBON / Support film - topology: HOLEY / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Time: 120 sec. / Pretreatment - Atmosphere: AIR | ||||||||||||||||||
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 277 K / Instrument: FEI VITROBOT MARK IV | ||||||||||||||||||
| Details | The sample was purified as a monodisperse complex |
- Electron microscopy #1
Electron microscopy #1
| Microscopy ID | 1 |
|---|---|
| Microscope | FEI TITAN KRIOS |
| Temperature | Min: 70.0 K / Max: 70.0 K |
| Specialist optics | Energy filter - Name: GIF Quantum SE / Energy filter - Slit width: 20 eV |
| Details | eBIC Krios1 |
| Image recording | Image recording ID: 1 / Film or detector model: GATAN K2 QUANTUM (4k x 4k) / Detector mode: COUNTING / Average electron dose: 45.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | C2 aperture diameter: 70.0 µm / Calibrated defocus max: 2.7 µm / Calibrated defocus min: 0.9 µm / Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.7 µm / Nominal defocus min: 0.9 µm / Nominal magnification: 130000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
- Electron microscopy #1~
Electron microscopy #1~
| Microscopy ID | 1 |
|---|---|
| Microscope | FEI TITAN KRIOS |
| Temperature | Min: 70.0 K / Max: 70.0 K |
| Specialist optics | Energy filter - Name: GIF Quantum SE / Energy filter - Slit width: 20 eV |
| Details | LMB Krios1 |
| Image recording | Image recording ID: 2 / Film or detector model: GATAN K2 QUANTUM (4k x 4k) / Average electron dose: 48.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | C2 aperture diameter: 50.0 µm / Calibrated defocus max: 2.7 µm / Calibrated defocus min: 0.9 µm / Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.7 µm / Nominal defocus min: 0.9 µm / Nominal magnification: 105000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
- Electron microscopy #1~~
Electron microscopy #1~~
| Microscopy ID | 1 |
|---|---|
| Microscope | FEI TITAN KRIOS |
| Temperature | Min: 70.0 K / Max: 70.0 K |
| Specialist optics | Energy filter - Name: GIF Quantum SE / Energy filter - Slit width: 20 eV |
| Details | LMB Krios 2 |
| Image recording | Image recording ID: 3 / Film or detector model: GATAN K2 QUANTUM (4k x 4k) / Average electron dose: 40.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | C2 aperture diameter: 50.0 µm / Calibrated defocus max: 2.7 µm / Calibrated defocus min: 0.9 µm / Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.7 µm / Nominal defocus min: 0.9 µm / Nominal magnification: 105000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
+ Image processing #1
Image processing #1
+ Image processing #2
Image processing #2
+ Image processing #3
Image processing #3
+ Image processing #4
Image processing #4
-Atomic model buiding 1
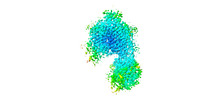
| Initial model |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| Details | Manual building was performed in Coot iterated with real space refinement in PHENIX. | ||||||||
| Refinement | Space: REAL / Overall B value: 112 / Target criteria: Correlation coefficient | ||||||||
| Output model |  PDB-7ad3: |
 Movie
Movie Controller
Controller























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)








































 Trichoplusia ni (cabbage looper)
Trichoplusia ni (cabbage looper)