+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0774 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Rat GluD1 receptor(compact conformation) in complex with 7-CKA and Calcium ions | |||||||||
 マップデータ マップデータ | final refined map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報glutamate receptor activity / regulation of postsynapse organization / social behavior / GABA-ergic synapse / synaptic transmission, glutamatergic / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / postsynaptic density membrane / modulation of chemical synaptic transmission / postsynaptic membrane / glutamatergic synapse ...glutamate receptor activity / regulation of postsynapse organization / social behavior / GABA-ergic synapse / synaptic transmission, glutamatergic / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / postsynaptic density membrane / modulation of chemical synaptic transmission / postsynaptic membrane / glutamatergic synapse / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 8.1 Å | |||||||||
 データ登録者 データ登録者 | Burada AP / Kumar J | |||||||||
| 資金援助 |  インド, 1件 インド, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2020 ジャーナル: Nat Struct Mol Biol / 年: 2020タイトル: Cryo-EM structures of the ionotropic glutamate receptor GluD1 reveal a non-swapped architecture. 著者: Ananth Prasad Burada / Rajesh Vinnakota / Janesh Kumar /  要旨: Ionotropic orphan delta (GluD) receptors are not gated by glutamate or any other endogenous ligand but are grouped with ionotropic glutamate receptors (iGluRs) based on sequence similarity. GluD1 ...Ionotropic orphan delta (GluD) receptors are not gated by glutamate or any other endogenous ligand but are grouped with ionotropic glutamate receptors (iGluRs) based on sequence similarity. GluD1 receptors play critical roles in synaptogenesis and synapse maintenance and have been implicated in neuronal disorders, including schizophrenia, cognitive deficits, and cerebral ataxia. Here we report cryo-EM structures of the rat GluD1 receptor complexed with calcium and the ligand 7-chlorokynurenic acid (7-CKA), elucidating molecular architecture and principles of receptor assembly. The structures reveal a non-swapped architecture at the interface of the extracellular amino-terminal domain (ATD) and the ligand-binding domain (LBD). This finding is in contrast with structures of other families of iGluRs, where the dimer partners between the ATD and LBD layers are swapped. Our results demonstrate that principles of architecture and symmetry are not conserved between delta receptors and other iGluRs and provide a molecular blueprint for understanding the functions of the 'orphan' class of iGluRs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0774.map.gz emd_0774.map.gz | 226.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0774-v30.xml emd-0774-v30.xml emd-0774.xml emd-0774.xml | 17.6 KB 17.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_0774.png emd_0774.png | 41.1 KB | ||
| その他 |  emd_0774_half_map_1.map.gz emd_0774_half_map_1.map.gz emd_0774_half_map_2.map.gz emd_0774_half_map_2.map.gz | 226.8 MB 226.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0774 http://ftp.pdbj.org/pub/emdb/structures/EMD-0774 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0774 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0774 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0774_validation.pdf.gz emd_0774_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0774_full_validation.pdf.gz emd_0774_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_0774_validation.xml.gz emd_0774_validation.xml.gz | 15.8 KB | 表示 | |
| CIF形式データ |  emd_0774_validation.cif.gz emd_0774_validation.cif.gz | 18.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0774 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0774 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0774 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0774 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6kssMC  0773C  6kspC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10428 (タイトル: Cryo-EM Structure of GluD1-Orphan Delta Receptor Reveals a Novel Architecture in the Ionotropic Glutamate Receptor Family EMPIAR-10428 (タイトル: Cryo-EM Structure of GluD1-Orphan Delta Receptor Reveals a Novel Architecture in the Ionotropic Glutamate Receptor FamilyData size: 6.5 TB Data #1: ionotropioc Glutamate receptor Delta1 [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0774.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0774.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | final refined map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.067 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: half map A
| ファイル | emd_0774_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
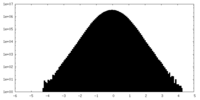
| 投影像・断面図 |
| ||||||||||||
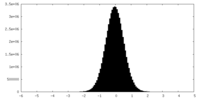
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_0774_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
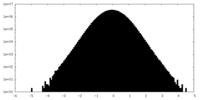
| 投影像・断面図 |
| ||||||||||||
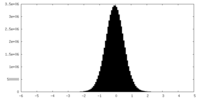
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GluD1
| 全体 | 名称: GluD1 |
|---|---|
| 要素 |
|
-超分子 #1: GluD1
| 超分子 | 名称: GluD1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Recombinantly expressed protein |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293S GnTI- / 組換プラスミド: pEG BACMAM Homo sapiens (ヒト) / 組換細胞: HEK293S GnTI- / 組換プラスミド: pEG BACMAM |
| 分子量 | 実験値: 388 KDa |
-分子 #1: Glutamate receptor ionotropic, delta-1
| 分子 | 名称: Glutamate receptor ionotropic, delta-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 95.718039 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DSIIHIGAIF EENAAKDDRV FQLAVSDLSL NDDILQSEKI TYSIKVIEAN NPFQAVQEAC DLMTQGILAL VTSTGCASAN ALQSLTDAM HIPHLFVQRN PGGSPRTACH LNPSPDGEAY TLASRPPVRL NDVMLRLVTE LRWQKFVMFY DSEYDIRGLQ S FLDQASRL ...文字列: DSIIHIGAIF EENAAKDDRV FQLAVSDLSL NDDILQSEKI TYSIKVIEAN NPFQAVQEAC DLMTQGILAL VTSTGCASAN ALQSLTDAM HIPHLFVQRN PGGSPRTACH LNPSPDGEAY TLASRPPVRL NDVMLRLVTE LRWQKFVMFY DSEYDIRGLQ S FLDQASRL GLDVSLQKVD KNISHVFTSL FTTMKTEELN RYRDTLRRAI LLLSPQGAHS FINEAVETNL ASKDSHWVFV NE EISDPEI LDLVHSALGR MTVVRQIFPS AKDNQKCMRN NHRISSLLCD PQEGYLQMLQ ISNLYLYDSV LMLANAFHRK LED RKWHSM ASLNCIRKST KPWNGGRSML DTIKKGHITG LTGVMEFRED SSNPYVQFEI LGTTYSETFG KDMRKLATWD SEKG LNGSL QERPMGSRLQ GLTLKVVTVL EEPFVMVAEN ILGQPKRYKG FSIDVLDALA KALGFKYEIY QAPDGRYGHQ LHNTS WNGM IGELISKRAD LAISAITITP ERESVVDFSK RYMDYSVGIL IKKPEEKISI FSLFAPFDFA VWACIAAAIP VVGVLI FVL NRIQAVRSQS ATQPRPSASA TLHSAIWIVY GAFVQQGGES SVNSVAMRIV MGSWWLFTLI VCSSYTANLA AFLTVSR MD SPVRTFQDLS KQLEMSYGTV RDSAVYEYFR AKGTNPLEQD STFAELWRTI SKNGGADNCV SNPSEGIRKA KKGNYAFL W DVAVVEYAAL TDDDCSVTVI GNSISSKGYG IALQHGSPYR DLFSQRILEL QDTGDLDVLK QKWWPHTGRC DLTSHSSAQ TDGKSLKLHS FAGVFCILAI GLLLACLVAA LELWWNSNRC HQETPKEDKE VGLVPR |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.9 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 20mM Tris, 150mM NaCl, 0.75mM DDM, 0.025mM CHS |
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 0-40 / 実像数: 4120 / 平均露光時間: 6.0 sec. / 平均電子線量: 40.38 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-6kss: |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X