+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0707 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of apo Machupo virus polymerase | |||||||||
 マップデータ マップデータ | apo Machupo virus polymerase | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative stranded viral RNA replication / cap snatching / virion component / host cell cytoplasm / 加水分解酵素; エステル加水分解酵素 / hydrolase activity / RNA-directed RNA polymerase / RNA-dependent RNA polymerase activity / nucleotide binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Machupo mammarenavirus (ウイルス) / Machupo mammarenavirus (ウイルス) /  Machupo virus (ウイルス) Machupo virus (ウイルス) | |||||||||
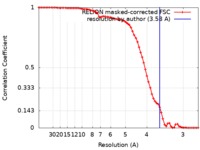
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.58 Å | |||||||||
 データ登録者 データ登録者 | Peng R / Xu X / Jing J / Peng Q / Gao GF / Shi Y | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Structural insight into arenavirus replication machinery. 著者: Ruchao Peng / Xin Xu / Jiamei Jing / Min Wang / Qi Peng / Sheng Liu / Ying Wu / Xichen Bao / Peiyi Wang / Jianxun Qi / George F Gao / Yi Shi /  要旨: Arenaviruses can cause severe haemorrhagic fever and neurological diseases in humans and other animals, exemplified by Lassa mammarenavirus, Machupo mammarenavirus and lymphocytic choriomeningitis ...Arenaviruses can cause severe haemorrhagic fever and neurological diseases in humans and other animals, exemplified by Lassa mammarenavirus, Machupo mammarenavirus and lymphocytic choriomeningitis virus, posing great threats to public health. These viruses encode a large multi-domain RNA-dependent RNA polymerase for transcription and replication of the viral genome. Viral polymerases are one of the leading antiviral therapeutic targets. However, the structure of arenavirus polymerase is not yet known. Here we report the near-atomic resolution structures of Lassa and Machupo virus polymerases in both apo and promoter-bound forms. These structures display a similar overall architecture to influenza virus and bunyavirus polymerases but possess unique local features, including an arenavirus-specific insertion domain that regulates the polymerase activity. Notably, the ordered active site of arenavirus polymerase is inherently switched on, without the requirement for allosteric activation by 5'-viral RNA, which is a necessity for both influenza virus and bunyavirus polymerases. Moreover, dimerization could facilitate the polymerase activity. These findings advance our understanding of the mechanism of arenavirus replication and provide an important basis for developing antiviral therapeutics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0707.map.gz emd_0707.map.gz | 2.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0707-v30.xml emd-0707-v30.xml emd-0707.xml emd-0707.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0707_fsc.xml emd_0707_fsc.xml | 7.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0707.png emd_0707.png | 82.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0707 http://ftp.pdbj.org/pub/emdb/structures/EMD-0707 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0707 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0707 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0707_validation.pdf.gz emd_0707_validation.pdf.gz | 362.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0707_full_validation.pdf.gz emd_0707_full_validation.pdf.gz | 361.9 KB | 表示 | |
| XML形式データ |  emd_0707_validation.xml.gz emd_0707_validation.xml.gz | 9.3 KB | 表示 | |
| CIF形式データ |  emd_0707_validation.cif.gz emd_0707_validation.cif.gz | 12.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0707 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0707 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0707 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0707 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0707.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0707.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | apo Machupo virus polymerase | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.36 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Machupo virus polymerase
| 全体 | 名称: Machupo virus polymerase |
|---|---|
| 要素 |
|
-超分子 #1: Machupo virus polymerase
| 超分子 | 名称: Machupo virus polymerase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Machupo mammarenavirus (ウイルス) Machupo mammarenavirus (ウイルス) |
| 組換発現 | 生物種:  Spodoptera aff. frugiperda 1 BOLD-2017 (蝶・蛾) Spodoptera aff. frugiperda 1 BOLD-2017 (蝶・蛾)組換細胞: HiFive |
| 分子量 | 実験値: 250 KDa |
-分子 #1: RNA-directed RNA polymerase L
| 分子 | 名称: RNA-directed RNA polymerase L / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: RNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Machupo virus (ウイルス) Machupo virus (ウイルス) |
| 分子量 | 理論値: 250.416062 KDa |
| 組換発現 | 生物種:  Spodoptera aff. frugiperda 1 BOLD-2017 (蝶・蛾) Spodoptera aff. frugiperda 1 BOLD-2017 (蝶・蛾) |
| 配列 | 文字列: MDEYVQELKG LIRKHIPERC EFGHQKVTFL SQVHPSPLLT EGFKLLSSLV ELESCEAHAC QANTDQRFVD VILSDNGILC PTLPKVIPD GFKLTGKTLI LLETFVRVNP DEFEKKWKAD MSKLLNLKHD LQKSGVTLVP IVDGRSNYNN RFVADWVIER I RWLLIEIL ...文字列: MDEYVQELKG LIRKHIPERC EFGHQKVTFL SQVHPSPLLT EGFKLLSSLV ELESCEAHAC QANTDQRFVD VILSDNGILC PTLPKVIPD GFKLTGKTLI LLETFVRVNP DEFEKKWKAD MSKLLNLKHD LQKSGVTLVP IVDGRSNYNN RFVADWVIER I RWLLIEIL KASKSMLEID IEDQEYQRLI HSLSNVKNQS LGLENLEHLK RNSLDYDERL NESLFIGLKG DIRESTVREE LI KLKLWFK DEVFSKGLGK FKLTDRRELL ESLSSLGAHL DSDVSSCPFC NNKLMEIVYN VTFSCVERTD GVATVDQQFS TTH SNIEKH YLSVLSLCNK IKGLKVFNTR RNTLLFLDLI MVNLMVDISD SCQDAIESLR KSGLIVGQMV MLVNDRVLDI LEAV KLIRK KIGTNPNWVK NCSKILERSH PEIWHHLSTL IKQPDFNSLI SIAQHLVSDR PIMRYSVERG SDKICRHKLF QEMSS FEQM RLFKTLSSIS LSLINSMKTS FSSRLLVNER EFSKYFGNVR LRECYAQRFY LAESLVGFLF YQKTGERSRC YSVYLS DNG VMSEQGSFYC DPKRFFLPVF SDEVLAGMCE EMTSWLDFDT GLMNDTGPIL RLLVLAILCS PSKRNQTFLQ GLRYFLM AF ANQIHHIDLT SKLVVECKSS SEVVVQRLAV GLFIRLLSGE SDASLFFSRR FKYLLNVSYL CHLITKETPD RLTDQIKC F EKFIEPKVKF GCAVVNPSLN GKLTVDQEDI MINGLKKFFS KSLRDTEDVQ TPGVCKELLN YCVSLFNRGK LKVSGELKN NPFRPNITST ALDLSSNKSV VIPKLDELGN ILSTYDKEKL VSACVSSMAE RFKTKGRYNL DPDSTDYLIL KNLTGLVSAG PKAKSTQEE LSLMYEALTE EQVESFNEIK HDVQVALAKM ADNSVNTRTK NLGRADNSVK NGNNPLDNLW SPFGVMKEIR A EVSLHEVK DFDPDVLPPE VYKELCDAVY KSSEKCNFFL EGVLDVCPLG LLLKNLTTSS YVDEEYFMCF KYLLIQGHFD QK LGSYEHK SRSRLGFTDE TLRLKDEVRL SIRESNSEAI ADKLDKSYFT NAALRNLCFY SEDSPTEFTS ISSNSGNLKF GLS YKEQVG SNRELYVGDL NTKLMTRLVE DFSEAVGNSM KYTCLNSEKE FERAICDMKM AVNNGDLSCS YDHSKWGPTM SPAL FLALL QMLELRTPVD RSKIDLDSVK SILKWHLHKV VEVPINVAEA YCIGKLKRSL GLMGCGSTSL SEEFFHQTMQ LNGQI PSHI MSVLDMGQGI LHNTSDLYGL ITEQFLCYAL DLLYDVIPVS YTSSDDQITL IKTPSLDIEG GSDAAEWLEM ICFHEF LSS KLNKFVSPKS VIGTFVAEFK SRFFVMGEET PLLTKFVAAA LHNVKCKTPT QLSETIDTIC DQCIANGVST KIVTRIS KR VNQLIRYSGY GETPFGAIED QDVKDWVDGS RGYRLQRKIE AIFHDDKETS FIRNCARKVF NDIKRGRIFE ENLINLIG R GGDEALTGFL QYAGCSEQEV NRVLNYRWVN LSSFGDLRLV LRTKLMTSRR VLEREEVPTL IKTLQSKLSR NFTKGVKKI LAESINKSAF QSSVASGFIG FCKSMGSKCV RDGKGGFLYI KEVYSGVSAC TCEICALKPK IIYCNNSLNK VSQFSKPILW DYFSLVLTN ACELGEWVFS TVKEPQKPLV LNNQNFFWAV KPKVVRQIED QLGMNHVLQS IRRNYPVLFD EHLTPFMNDL Q VSRTMDSG RLKFLDVCIA LDMMNENLGI ISHLLKTRDN SVYIVKQSDC ALAHIRQSSY TDWELGLSPQ QICTNFKTQL VL SSMVNPL VLSTSCLKSF FWFNEVLELE DDSQIELAEL TDFALMVKNQ NVSRAMFVED IAMGYVVSNF EGVRISLSNV MVD GVQLPP QEKAPDIGEL FGLKAENVIV GLVVQIDHVR MSTKFKLKRK MVYSFSLECI MDVGEIQNKE VILKVVAVDQ SVSG SGGNH MLLDGVSVVA SLPLFTGQAS FDLAAMLIES NLAGSNDNFL MRNVTLDLGG FSPELSDKYS YRLSGPENQE DPLVL KDGA FYVGGERLST YKVEFTGDLV VKALGALEDD ESVVSMLHQL WPYLKATSQV ILFQQEDFTI VHDLYKKQLT KSIESF GEW IEFTNFKVAY SKSLKELVIS DTQGSFRLKG VMCRPLASTP QVEDIE |
-分子 #2: MANGANESE (II) ION
| 分子 | 名称: MANGANESE (II) ION / タイプ: ligand / ID: 2 / コピー数: 1 / 式: MN |
|---|---|
| 分子量 | 理論値: 54.938 Da |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6kld: |
 ムービー
ムービー コントローラー
コントローラー