+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12484 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the folate-specific ECF transporter complex in MSP2N2 lipid nanodiscs | |||||||||||||||
 マップデータ マップデータ | Inward-facing apo conformation of the folate-specific ECF transporter complex in MSP2N2 lipid nanodiscs at 2.7 A resolution sharpened at -44 A^2. | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報トランスロカーゼ /  加水分解酵素; 酸無水物に作用; 酸無水物に作用・物質の膜輸送を触媒する / transmembrane transporter activity / ATPase-coupled transmembrane transporter activity / ATP-binding cassette (ABC) transporter complex / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・物質の膜輸送を触媒する / transmembrane transporter activity / ATPase-coupled transmembrane transporter activity / ATP-binding cassette (ABC) transporter complex /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / ATP binding /  生体膜 / 生体膜 /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Lactobacillus delbrueckii subsp. bulgaricus (strain ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778) (ラクトバチルス・デルブルエッキイー) Lactobacillus delbrueckii subsp. bulgaricus (strain ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778) (ラクトバチルス・デルブルエッキイー) | |||||||||||||||
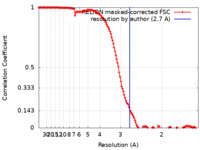
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.7 Å クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||||||||
 データ登録者 データ登録者 | Thangaratnarajah C / Rheinberger J / Paulino C / Slotboom DJ | |||||||||||||||
| 資金援助 |  オランダ, 4件 オランダ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2021 ジャーナル: Proc Natl Acad Sci U S A / 年: 2021タイトル: Insights into the bilayer-mediated toppling mechanism of a folate-specific ECF transporter by cryo-EM. 著者: Chancievan Thangaratnarajah / Jan Rheinberger / Cristina Paulino / Dirk J Slotboom /  要旨: Energy-coupling factor (ECF)-type transporters are small, asymmetric membrane protein complexes (∼115 kDa) that consist of a membrane-embedded, substrate-binding protein (S component) and a ...Energy-coupling factor (ECF)-type transporters are small, asymmetric membrane protein complexes (∼115 kDa) that consist of a membrane-embedded, substrate-binding protein (S component) and a tripartite ATP-hydrolyzing module (ECF module). They import micronutrients into bacterial cells and have been proposed to use a highly unusual transport mechanism, in which the substrate is dragged across the membrane by a toppling motion of the S component. However, it remains unclear how the lipid bilayer could accommodate such a movement. Here, we used cryogenic electron microscopy at 200 kV to determine structures of a folate-specific ECF transporter in lipid nanodiscs and detergent micelles at 2.7- and 3.4-Å resolution, respectively. The structures reveal an irregularly shaped bilayer environment around the membrane-embedded complex and suggest that toppling of the S component is facilitated by protein-induced membrane deformations. In this way, structural remodeling of the lipid bilayer environment is exploited to guide the transport process. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12484.map.gz emd_12484.map.gz | 202.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12484-v30.xml emd-12484-v30.xml emd-12484.xml emd-12484.xml | 27.5 KB 27.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12484_fsc.xml emd_12484_fsc.xml | 13.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12484.png emd_12484.png | 149.5 KB | ||
| マスクデータ |  emd_12484_msk_1.map emd_12484_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| その他 |  emd_12484_additional_1.map.gz emd_12484_additional_1.map.gz emd_12484_half_map_1.map.gz emd_12484_half_map_1.map.gz emd_12484_half_map_2.map.gz emd_12484_half_map_2.map.gz | 171.3 MB 172 MB 172 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12484 http://ftp.pdbj.org/pub/emdb/structures/EMD-12484 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12484 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12484 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7nnuMC  7nntC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10942 (タイトル: Cryo-EM structure of the folate-specific ECF transporter complex in MSP2N2 lipid nanodiscs (200 kV) EMPIAR-10942 (タイトル: Cryo-EM structure of the folate-specific ECF transporter complex in MSP2N2 lipid nanodiscs (200 kV)Data size: 1.1 TB Data #1: Unaligned movie frames of ECF-FolT2 in MSP2N2 lipid nanodiscs (Dataset 1) [micrographs - multiframe] Data #2: Unaligned movie frames of ECF-FolT2 in MSP2N2 lipid nanodiscs (Dataset 2) [micrographs - multiframe] Data #3: Unaligned movie frames of ECF-FolT2 in MSP2N2 lipid nanodiscs (Dataset 3) [micrographs - multiframe] Data #4: Unaligned movie frames of ECF-FolT2 in MSP2N2 lipid nanodiscs (Dataset 3) [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12484.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12484.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Inward-facing apo conformation of the folate-specific ECF transporter complex in MSP2N2 lipid nanodiscs at 2.7 A resolution sharpened at -44 A^2. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.771 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_12484_msk_1.map emd_12484_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
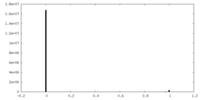
| 密度ヒストグラム |
-追加マップ: Sharpened map obtained with LocalDeblur used for model...
| ファイル | emd_12484_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map obtained with LocalDeblur used for model building and figures. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
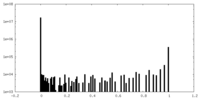
| 密度ヒストグラム |
-ハーフマップ: Half map 1 used during refinement and FSC...
| ファイル | emd_12484_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 used during refinement and FSC gold-standard resolution calculation. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
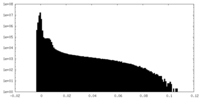
| 密度ヒストグラム |
-ハーフマップ: Half map 2 used during refinement and FSC...
| ファイル | emd_12484_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 used during refinement and FSC gold-standard resolution calculation. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
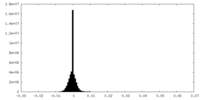
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Folate-specific ECF transporter complex
| 全体 | 名称: Folate-specific ECF transporter complex |
|---|---|
| 要素 |
|
-超分子 #1: Folate-specific ECF transporter complex
| 超分子 | 名称: Folate-specific ECF transporter complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Lactobacillus delbrueckii subsp. bulgaricus (strain ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778) (ラクトバチルス・デルブルエッキイー) Lactobacillus delbrueckii subsp. bulgaricus (strain ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778) (ラクトバチルス・デルブルエッキイー)株: ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778 |
| 組換発現 | 生物種:   Escherichia coli MC1061 (大腸菌) / 組換プラスミド: p2BAD Escherichia coli MC1061 (大腸菌) / 組換プラスミド: p2BAD |
-分子 #1: Energy-coupling factor transporter ATP-binding protein EcfA1
| 分子 | 名称: Energy-coupling factor transporter ATP-binding protein EcfA1 タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: トランスロカーゼ |
|---|---|
| 由来(天然) | 生物種:   Lactobacillus delbrueckii subsp. bulgaricus (strain ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778) (ラクトバチルス・デルブルエッキイー) Lactobacillus delbrueckii subsp. bulgaricus (strain ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778) (ラクトバチルス・デルブルエッキイー)株: ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778 |
| 分子量 | 理論値: 33.166418 KDa |
| 組換発現 | 生物種:   Escherichia coli MC1061 (大腸菌) Escherichia coli MC1061 (大腸菌) |
| 配列 | 文字列: MHHHHHHHHH HGENLYFQGS DNIISFDHVT FTYPDSPRPA LSDLSFAIER GSWTALIGHN GSGKSTVSKL INGLLAPDDL DKSSITVDG VKLGADTVWE VREKVGIVFQ NPDNQFVGAT VSDDVAFGLE NRAVPRPEML KIVAQAVADV GMADYADSEP S NLSGGQKQ ...文字列: MHHHHHHHHH HGENLYFQGS DNIISFDHVT FTYPDSPRPA LSDLSFAIER GSWTALIGHN GSGKSTVSKL INGLLAPDDL DKSSITVDG VKLGADTVWE VREKVGIVFQ NPDNQFVGAT VSDDVAFGLE NRAVPRPEML KIVAQAVADV GMADYADSEP S NLSGGQKQ RVAIAGILAV KPQVIILDES TSMLDPEGKE QILDLVRKIK EDNNLTVISI THDLEEAAGA DQVLVLDDGQ LL DQGKPEE IFPKVEMLKR IGLDIPFVYR LKQLLKERGI VLPDEIDDDE KLVQSLWQLN SKM |
-分子 #2: Energy-coupling factor transporter ATP-binding protein EcfA2
| 分子 | 名称: Energy-coupling factor transporter ATP-binding protein EcfA2 タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO EC番号:  加水分解酵素; 酸無水物に作用; 酸無水物に作用・物質の膜輸送を触媒する 加水分解酵素; 酸無水物に作用; 酸無水物に作用・物質の膜輸送を触媒する |
|---|---|
| 由来(天然) | 生物種:   Lactobacillus delbrueckii subsp. bulgaricus (strain ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778) (ラクトバチルス・デルブルエッキイー) Lactobacillus delbrueckii subsp. bulgaricus (strain ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778) (ラクトバチルス・デルブルエッキイー)株: ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778 |
| 分子量 | 理論値: 31.672156 KDa |
| 組換発現 | 生物種:   Escherichia coli MC1061 (大腸菌) Escherichia coli MC1061 (大腸菌) |
| 配列 | 文字列: MAIKFENVSY VYSPGSPLEA IGLDQLNFSL EEGKFIALVG HTGSGKSTLM QHFNALLKPT SGKIEIAGYT ITPETGNKGL KDLRRKVSL AFQFSEAQLF ENTVLKDVEY GPRNFGFSED EAREAALKWL KKVGLKDDLI EHSPFDLSGG QMRRVALAGV L AYEPEIIC ...文字列: MAIKFENVSY VYSPGSPLEA IGLDQLNFSL EEGKFIALVG HTGSGKSTLM QHFNALLKPT SGKIEIAGYT ITPETGNKGL KDLRRKVSL AFQFSEAQLF ENTVLKDVEY GPRNFGFSED EAREAALKWL KKVGLKDDLI EHSPFDLSGG QMRRVALAGV L AYEPEIIC LDEPAAGLDP MGRLEMMQLF KDYQAAGHTV ILVTHNMDDV ADYADDVLAL EHGRLIKHAS PKEVFKDSEW LQ KHHLAEP RSARFAAKLE AAGLKLPGQP LTMPELADAI KQSLKGGEHE |
-分子 #3: Conserved hypothetical membrane protein
| 分子 | 名称: Conserved hypothetical membrane protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Lactobacillus delbrueckii subsp. bulgaricus (strain ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778) (ラクトバチルス・デルブルエッキイー) Lactobacillus delbrueckii subsp. bulgaricus (strain ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778) (ラクトバチルス・デルブルエッキイー)株: ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778 |
| 分子量 | 理論値: 20.483604 KDa |
| 組換発現 | 生物種:   Escherichia coli MC1061 (大腸菌) Escherichia coli MC1061 (大腸菌) |
| 配列 | 文字列: MKSESKVSSK LELRELVLLA MVIAIKVILG QFKVGNATLQ VGLGFIGSVM LGYLFGPWWG FAGGALSDLV SSVIFGNLGG FFIGFTLTA ALGPMIYGFF LYKQPIQIWR VIASVICVTV ICNIGLNTLW VSMMYGINFM VALSSRILKE MITPWIQMVA V WFILEGLS RVKLSRKFWS HPQFEK |
-分子 #4: Energy-coupling factor transporter transmembrane protein EcfT
| 分子 | 名称: Energy-coupling factor transporter transmembrane protein EcfT タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Lactobacillus delbrueckii subsp. bulgaricus (strain ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778) (ラクトバチルス・デルブルエッキイー) Lactobacillus delbrueckii subsp. bulgaricus (strain ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778) (ラクトバチルス・デルブルエッキイー)株: ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778 |
| 分子量 | 理論値: 30.290283 KDa |
| 組換発現 | 生物種:   Escherichia coli MC1061 (大腸菌) Escherichia coli MC1061 (大腸菌) |
| 配列 | 文字列: MSKIIIGRYL PGTTFVYRVD PRAKLLTTFY FIIMIFLANN WVSYLVISIF GLAYVFATGL KARVFWDGVK PMIWMIVFTS LLQTFFMAG GKVYWHWWIF TLSSEGLING LYVFIRFAMI ILVSTVMTVT TKPLEIADAM EWMLTPLKLF KVNVGMISLV I SIALRFVP ...文字列: MSKIIIGRYL PGTTFVYRVD PRAKLLTTFY FIIMIFLANN WVSYLVISIF GLAYVFATGL KARVFWDGVK PMIWMIVFTS LLQTFFMAG GKVYWHWWIF TLSSEGLING LYVFIRFAMI ILVSTVMTVT TKPLEIADAM EWMLTPLKLF KVNVGMISLV I SIALRFVP TLFDQTVKIM NAQRSRGADF NDGGLVKRAK SVVPMLVPLF IDSLEVALDL STAMESRGYK GSEGRTRYRI LE WSKVDLI PVAYCLLLTI LMITTRKH |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 35 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 7.9 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 20 mM Tris, pH 8.0, 150 mM NaCl |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 詳細: 5 mA |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 288 K / 装置: FEI VITROBOT MARK IV 詳細: 2.9 mM fluorinated Fos-choline 8 was added prior to sample application onto grids. Grids were blotted for 3-4 sec.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 165000 Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 165000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 7676 pixel / デジタル化 - サイズ - 縦: 7420 pixel / デジタル化 - 画像ごとのフレーム数: 1-60 / 撮影したグリッド数: 3 / 実像数: 4722 / 平均露光時間: 9.0 sec. / 平均電子線量: 51.1 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X