+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9902 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | S-OPA1 coated liposome tube at GTPgamaS bound state | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
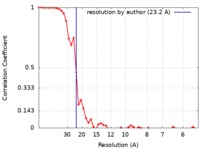
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 23.2 Å | ||||||||||||
 データ登録者 データ登録者 | Zhang D / Zhang Y / Sun F | ||||||||||||
| 資金援助 |  中国, 3件 中国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Cryo-EM structures of S-OPA1 reveal its interactions with membrane and changes upon nucleotide binding. 著者: Danyang Zhang / Yan Zhang / Jun Ma / Chunmei Zhu / Tongxin Niu / Wenbo Chen / Xiaoyun Pang / Yujia Zhai / Fei Sun /  要旨: Mammalian mitochondrial inner membrane fusion is mediated by optic atrophy 1 (OPA1). Under physiological conditions, OPA1 undergoes proteolytic processing to form a membrane-anchored long isoform (L- ...Mammalian mitochondrial inner membrane fusion is mediated by optic atrophy 1 (OPA1). Under physiological conditions, OPA1 undergoes proteolytic processing to form a membrane-anchored long isoform (L-OPA1) and a soluble short isoform (S-OPA1). A combination of L-OPA1 and S-OPA1 is essential for efficient membrane fusion; however, the relevant mechanism is not well understood. In this study, we investigate the cryo-electron microscopic structures of S-OPA1-coated liposomes in nucleotide-free and GTPγS-bound states. S-OPA1 exhibits a general dynamin-like structure and can assemble onto membranes in a helical array with a dimer building block. We reveal that hydrophobic residues in its extended membrane-binding domain are critical for its tubulation activity. The binding of GTPγS triggers a conformational change and results in a rearrangement of the helical lattice and tube expansion similar to that of S-Mgm1. These observations indicate that S-OPA1 adopts a dynamin-like power stroke membrane remodeling mechanism during mitochondrial inner membrane fusion. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9902.map.gz emd_9902.map.gz | 6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9902-v30.xml emd-9902-v30.xml emd-9902.xml emd-9902.xml | 15.7 KB 15.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9902_fsc.xml emd_9902_fsc.xml | 5.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9902.png emd_9902.png | 73.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9902 http://ftp.pdbj.org/pub/emdb/structures/EMD-9902 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9902 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9902 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9902_validation.pdf.gz emd_9902_validation.pdf.gz | 78.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9902_full_validation.pdf.gz emd_9902_full_validation.pdf.gz | 77.7 KB | 表示 | |
| XML形式データ |  emd_9902_validation.xml.gz emd_9902_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9902 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9902 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9902 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9902 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9902.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9902.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.72 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : truncated S-OPA1(253-960) coated liposomal tube at GTPgamas bound...
| 全体 | 名称: truncated S-OPA1(253-960) coated liposomal tube at GTPgamas bound state |
|---|---|
| 要素 |
|
-超分子 #1: truncated S-OPA1(253-960) coated liposomal tube at GTPgamas bound...
| 超分子 | 名称: truncated S-OPA1(253-960) coated liposomal tube at GTPgamas bound state タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: 1mg/ml S-OPA1(253-960) was incubated with 1mg/ml liposome at room temperature for 30 min. Then GTPgamaS was added to a final concentration of 1mM, and another 30 min incubation was performed. |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / Organelle: Mitochondria / 細胞中の位置: mitochondrial inner membrane Homo sapiens (ヒト) / Organelle: Mitochondria / 細胞中の位置: mitochondrial inner membrane |
| 組換発現 | 生物種:  |
-分子 #1: Dynamin-like 120 kDa protein, mitochondrial, short form for isofo...
| 分子 | 名称: Dynamin-like 120 kDa protein, mitochondrial, short form for isoform 1; Optic atrophy protein 1 (OPA1), short form for isoform 1 タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPGSDKGIHH RKLKKSLIDM YSEVLDVLSD YDASYNTQDH LPRVVVVGDQ SAGKTSVLEM IAQARIFPRG SGEMMTRSPV KVTLSEGPHH VALFKDSSRE FDLTKEEDLA ALRHEIELRM RKNVKEGCTV SPETISLNVK GPGLQRMVLV DLPGVINTVT SGMAPDTKET ...文字列: GPGSDKGIHH RKLKKSLIDM YSEVLDVLSD YDASYNTQDH LPRVVVVGDQ SAGKTSVLEM IAQARIFPRG SGEMMTRSPV KVTLSEGPHH VALFKDSSRE FDLTKEEDLA ALRHEIELRM RKNVKEGCTV SPETISLNVK GPGLQRMVLV DLPGVINTVT SGMAPDTKET IFSISKAYMQ NPNAIILCIQ DGSVDAERSI VTDLVSQMDP HGRRTIFVLT KVDLAEKNVA SPSRIQQIIE GKLFPMKALG YFAVVTGKGN SSESIEAIRE YEEEFFQNSK LLKTSMLKAH QVTTRNLSLA VSDCFWKMVR ESVEQQADSF KATRFNLETE WKNNYPRLRE LDRNELFEKA KNEILDEVIS LSQVTPKHWE EILQQSLWER VSTHVIENIY LPAAQTMNSG TFNTTVDIKL KQWTDKQLPN KAVEVAWETL QEEFSRFMTE PKGKEHDDIF DKLKEAVKEE SIKRHKWNDF AEDSLRVIQH NALEDRSISD KQQWDAAIYF MEEALQARLK DTENAIENMV GPDWKKRWLY WKNRTQEQCV HNETKNELEK MLKCNEEHPA YLASDEITTV RKNLESRGVE VDPSLIKDTW HQVYRRHFLK TALNHCNLCR RGFYYYQRHF VDSELECNDV VLFWRIQRML AITANTLRQQ LTNTEVRRLE KNVKEVLEDF AEDGEKKIKL LTGKRVQLAE DLKKVREIQE KLDAFIEALH QEK |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: OTHER | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 289 K / 装置: FEI VITROBOT MARK IV 詳細: The grid was blotted 3.5s with a force 1 before plunging. | ||||||||||||
| 詳細 | 1mg/ml SOPA1(delta 196-252) was incubated with 1mg/ml liposome at room temperature for 30 min. Then GTPgamaS was added to a final concentration of 1mM, and another 30 min incubation was performed. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-20 / 平均露光時間: 1.0 sec. / 平均電子線量: 3.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 6.0 µm / 最小 デフォーカス(公称値): 4.0 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)