+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9398 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of the LbCas12a-crRNA-AcrVA4 dimer | |||||||||
 マップデータ マップデータ | LbCas12a-crRNA-AcrVA4 dimer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | UNKNOWN FUNCTION-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Lachnospiraceae bacterium ND2006 (バクテリア) / Lachnospiraceae bacterium ND2006 (バクテリア) /  Moraxella bovoculi (バクテリア) Moraxella bovoculi (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.38 Å | |||||||||
 データ登録者 データ登録者 | Chang L / Li Z | |||||||||
 引用 引用 |  ジャーナル: Cell Host Microbe / 年: 2019 ジャーナル: Cell Host Microbe / 年: 2019タイトル: Structural Basis for the Inhibition of CRISPR-Cas12a by Anti-CRISPR Proteins. 著者: Heng Zhang / Zhuang Li / Courtney M Daczkowski / Clinton Gabel / Andrew D Mesecar / Leifu Chang /  要旨: CRISPR-Cas12a (Cpf1), a type V CRISPR-associated nuclease, provides bacterial immunity against bacteriophages and plasmids but also serves as a tool for genome editing. Foreign nucleic acids are ...CRISPR-Cas12a (Cpf1), a type V CRISPR-associated nuclease, provides bacterial immunity against bacteriophages and plasmids but also serves as a tool for genome editing. Foreign nucleic acids are integrated into the CRISPR locus, prompting transcription of CRISPR RNAs (crRNAs) that guide Cas12a cleavage of foreign complementary DNA. However, mobile genetic elements counteract Cas12a with inhibitors, notably type V-A anti-CRISPRs (AcrVAs). We present cryoelectron microscopy structures of Cas12a-crRNA bound to AcrVA1 and AcrVA4 at 3.5 and 3.3 Å resolutions, respectively. AcrVA1 is sandwiched between the recognition (REC) and nuclease (NUC) lobes of Cas12a and inserts into the binding pocket for the protospacer-adjacent motif (PAM), a short DNA sequence guiding Cas12a targeting. AcrVA1 cleaves crRNA in a Cas12a-dependent manner, inactivating Cas12a-crRNA complexes. The AcrVA4 dimer is anchored around the crRNA pseudoknot of Cas12a-crRNA, preventing required conformational changes for crRNA-DNA heteroduplex formation. These results uncover molecular mechanisms for CRISPR-Cas12a inhibition, providing insights into bacteria-phage dynamics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9398.map.gz emd_9398.map.gz | 4.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9398-v30.xml emd-9398-v30.xml emd-9398.xml emd-9398.xml | 12.3 KB 12.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9398.png emd_9398.png | 54.2 KB | ||
| Filedesc metadata |  emd-9398.cif.gz emd-9398.cif.gz | 6.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9398 http://ftp.pdbj.org/pub/emdb/structures/EMD-9398 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9398 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9398 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9398_validation.pdf.gz emd_9398_validation.pdf.gz | 388.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9398_full_validation.pdf.gz emd_9398_full_validation.pdf.gz | 388.1 KB | 表示 | |
| XML形式データ |  emd_9398_validation.xml.gz emd_9398_validation.xml.gz | 6 KB | 表示 | |
| CIF形式データ |  emd_9398_validation.cif.gz emd_9398_validation.cif.gz | 6.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9398 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9398 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9398 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9398 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6nm9MC  0445C  0446C  0447C  0449C  6nmaC  6nmcC  6nmdC  6nmeC  6omvC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9398.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9398.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
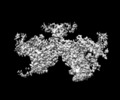
| 注釈 | LbCas12a-crRNA-AcrVA4 dimer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.066 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
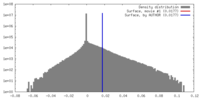
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : protein complex 5
| 全体 | 名称: protein complex 5 |
|---|---|
| 要素 |
|
-超分子 #1: protein complex 5
| 超分子 | 名称: protein complex 5 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Lachnospiraceae bacterium ND2006 (バクテリア) Lachnospiraceae bacterium ND2006 (バクテリア) |
-分子 #1: AcrVA4
| 分子 | 名称: AcrVA4 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Moraxella bovoculi (バクテリア) Moraxella bovoculi (バクテリア) |
| 分子量 | 理論値: 27.369162 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MYEIKLNDTL IHQTDDRVNA FVAYRYLLRR GDLPKCENIA RMYYDGKVIK TDVIDHDSVH SDEQAKVSNN DIIKMAISEL GVNNFKSLI KKQGYPFSNG HINSWFTDDP VKSKTMHNDE MYLVVQALIR ACIIKEIDLY TEQLYNIIKS LPYDKRPNVV Y SDQPLDPN ...文字列: MYEIKLNDTL IHQTDDRVNA FVAYRYLLRR GDLPKCENIA RMYYDGKVIK TDVIDHDSVH SDEQAKVSNN DIIKMAISEL GVNNFKSLI KKQGYPFSNG HINSWFTDDP VKSKTMHNDE MYLVVQALIR ACIIKEIDLY TEQLYNIIKS LPYDKRPNVV Y SDQPLDPN NLDLSEPELW AEQVGECMRY AHNDQPCFYI GSTKRELRVN YIVPVIGVRD EIERVMTLEE VRNLHK UniProtKB: Uncharacterized protein |
-分子 #2: Cpf1
| 分子 | 名称: Cpf1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lachnospiraceae bacterium ND2006 (バクテリア) Lachnospiraceae bacterium ND2006 (バクテリア) |
| 分子量 | 理論値: 143.750219 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSKLEKFTNC YSLSKTLRFK AIPVGKTQEN IDNKRLLVED EKRAEDYKGV KKLLDRYYLS FINDVLHSIK LKNLNNYISL FRKKTRTEK ENKELENLEI NLRKEIAKAF KGNEGYKSLF KKDIIETILP EFLDDKDEIA LVNSFNGFTT AFTGFFDNRE N MFSEEAKS ...文字列: MSKLEKFTNC YSLSKTLRFK AIPVGKTQEN IDNKRLLVED EKRAEDYKGV KKLLDRYYLS FINDVLHSIK LKNLNNYISL FRKKTRTEK ENKELENLEI NLRKEIAKAF KGNEGYKSLF KKDIIETILP EFLDDKDEIA LVNSFNGFTT AFTGFFDNRE N MFSEEAKS TSIAFRCINE NLTRYISNMD IFEKVDAIFD KHEVQEIKEK ILNSDYDVED FFEGEFFNFV LTQEGIDVYN AI IGGFVTE SGEKIKGLNE YINLYNQKTK QKLPKFKPLY KQVLSDRESL SFYGEGYTSD EEVLEVFRNT LNKNSEIFSS IKK LEKLFK NFDEYSSAGI FVKNGPAIST ISKDIFGEWN VIRDKWNAEY DDIHLKKKAV VTEKYEDDRR KSFKKIGSFS LEQL QEYAD ADLSVVEKLK EIIIQKVDEI YKVYGSSEKL FDADFVLEKS LKKNDAVVAI MKDLLDSVKS FENYIKAFFG EGKET NRDE SFYGDFVLAY DILLKVDHIY DAIRNYVTQK PYSKDKFKLY FQNPQFMGGW DKDKETDYRA TILRYGSKYY LAIMDK KYA KCLQKIDKDD VNGNYEKINY KLLPGPNKML PKVFFSKKWM AYYNPSEDIQ KIYKNGTFKK GDMFNLNDCH KLIDFFK DS ISRYPKWSNA YDFNFSETEK YKDIAGFYRE VEEQGYKVSF ESASKKEVDK LVEEGKLYMF QIYNKDFSDK SHGTPNLH T MYFKLLFDEN NHGQIRLSGG AELFMRRASL KKEELVVHPA NSPIANKNPD NPKKTTTLSY DVYKDKRFSE DQYELHIPI AINKCPKNIF KINTEVRVLL KHDDNPYVIG IDRGERNLLY IVVVDGKGNI VEQYSLNEII NNFNGIRIKT DYHSLLDKKE KERFEARQN WTSIENIKEL KAGYISQVVH KICELVEKYD AVIALEDLNS GFKNSRVKVE KQVYQKFEKM LIDKLNYMVD K KSNPCATG GALKGYQITN KFESFKSMST QNGFIFYIPA WLTSKIDPST GFVNLLKTKY TSIADSKKFI SSFDRIMYVP EE DLFEFAL DYKNFSRTDA DYIKKWKLYS YGNRIRIFRN PKKNNVFDWE EVCLTSAYKE LFNKYGINYQ QGDIRALLCE QSD KAFYSS FMALMSLMLQ MRNSITGRTD VDFLISPVKN SDGIFYDSRN YEAQENAILP KNADANGAYN IARKVLWAIG QFKK AEDEK LDKVKIAISN KEWLEYAQTS VK UniProtKB: Cpf1 |
-分子 #3: RNA (25-MER)
| 分子 | 名称: RNA (25-MER) / タイプ: rna / ID: 3 / コピー数: 2 |
|---|---|
| 由来(天然) | 生物種:  Lachnospiraceae bacterium ND2006 (バクテリア) Lachnospiraceae bacterium ND2006 (バクテリア) |
| 分子量 | 理論値: 12.879634 KDa |
| 配列 | 文字列: AAUUUCUACU AAGUGUAGAU GGAAAUUAGG UGCGCUUGGC |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 35.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C2 (2回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.38 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cisTEM (ver. 1.0.0) / 使用した粒子像数: 47609 |
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)